Complémentation de l’activité d’épissage par un complexe Galectin-3 - U1 snRNP sur perles
Summary
Cet article décrit les procédures expérimentales pour (a) l’épuisement du PNSNS U1 à partir d’extraits nucléaires, avec perte concomitante de l’activité d’épissage; et b) reconstitution de l’activité d’épissage dans l’extrait appauvri en U1 par des particules de galectine-3 -U1 snRNP liées à des billes couplées de manière covalente à des anticorps anti-galectine-3.
Abstract
Les expériences classiques d’épuisement-reconstitution indiquent que la galectine-3 est un facteur d’épissage requis dans les extraits nucléaires. Le mécanisme d’incorporation de la galectine-3 dans la voie d’épissage est abordé dans cet article. La sédimentation d’extraits nucléaires de cellules HeLa sur des gradients de glycérol de 12% à 32% donne des fractions enrichies en une particule endogène ~10S qui contient de la galectine-3 et du snRNP U1. Nous décrivons maintenant un protocole visant à épuiser les extraits nucléaires de SnRNP U1 avec perte concomitante d’activité d’épissage. L’activité d’épissage dans l’extrait appauvri en U1 peut être reconstituée par la particule de galectine-3 – U1 snRNP piégée sur des billes d’agarose couplées de manière covalente à des anticorps anti-galectine-3. Les résultats indiquent que le complexe ternaire pré-ARNm galectine-3 – U1 snRNP – est un complexe fonctionnel E conduisant à des intermédiaires et des produits de la réaction d’épissage et que la galectine-3 pénètre dans la voie d’épissage par son association avec U1 snRNP. Le schéma consistant à utiliser des complexes d’affinité ou d’immuno-sélection sur des billes pour reconstituer l’activité d’épissage dans des extraits appauvris d’un facteur d’épissage spécifique peut être généralement applicable à d’autres systèmes.
Introduction
La production de la plupart des ARN messagers eucaryotes (ARNm) implique l’élimination des introns et la ligature des exons dans un processus nucléaire appelé épissage pré-ARNm1. Deux classes de complexes ARN-protéine (RNP) dirigent le traitement de l’ARN pré-messager en ARNm mature via des complexes splicéosomiques. Une classe, les RNP pré-messagers naissants, est formée co-transcriptionnellement par la liaison de protéines RNP nucléaires hétérogènes et d’autres protéines de liaison à l’ARN, y compris certains membres de la famille SR, produisant des complexes hnRNP2. Les petits RNP nucléaires de deuxième classe, riches en uraciles (SNNP U avec des SNRNA U1, U2, U4, U5 et U6) sont associés à des protéines spécifiques à l’U et au noyau3,4. Les SNNP U interagissent de manière ordonnée avec des régions spécifiques des RNP pré-messagers dans une voie de remodelage dynamique à mesure que les introns sont excisés et que les exons sont ligaturés pour produire des ARNm matures5. De nombreuses protéines nucléaires supplémentaires participent à ces événements de traitement6.
La galectine-1 (Gal1) et la galectine-3 (Gal3) sont deux protéines qui sont des facteurs requis dans la voie d’épissage, comme le montrent les études d’épuisement-reconstitution7,8. L’élimination des deux galectines de l’épissage d’extraits nucléaires compétents (NE) abolit l’assemblage et l’épissage des épissages à un stade précoce. L’ajout de l’une ou l’autre galectine à un NE doublement appauvri restaure les deux activités. Gal1 et Gal3 sont des composants des spliceosomes actifs, comme en témoigne l’immunoprécipitation spécifique du pré-ARNm, des intermédiaires d’épissage et de l’ARNm mature par antisérum spécifique pour Gal1 ou Gal39. Il est important de noter que Gal3 s’associe à des particules endogènes contenant de l’ARN U dans le NE en dehors de la voie d’épissage, comme le montre la précipitation des snRNP par l’antisérum anti-Gal310. Enfin, le silence de Gal3 dans les cellules HeLa modifie les schémas d’épissage de nombreux gènes11.
Dans le NE pré-incubé pour désassembler les spliceosomes préformés12, les snRNP se retrouvent dans de multiples complexes sédimentant dans des gradients de glycérol de 7S à plus de 60S. Bien que le fractionnement du gradient de glycérol soit une technique courante pour l’isolement des complexes et composants splicéosomiques (voir références13,14,15 par exemple), nous avons étendu cette méthode en caractérisant des fractions spécifiques à l’aide d’immunoprécipitations d’anticorps. Un snRNP sédimentant à 10S ne contient que de l’ARNS U1 avec Gal3. L’immunoprécipitation de la fraction 10S avec des antisérums spécifiques pour Gal3 ou U1 snRNP co-précipite à la fois U1 et Gal3 indiquant que certaines des monoparticules SnRNP U1 sont liées à Gal310. Comme U1 snRNP est le premier complexe qui se lie au pré-mRNP dans l’assemblage splicéosomal1,5, cette étape représente un site d’entrée potentiel pour Gal3 dans la voie d’épissage. Sur cette base, nous avons montré que les monoparticules de snRNP 10S Gal3-U1 liées à des perles contenant des anti-Gal3 ont restauré l’activité d’épissage à un NE appauvri en U1 snRNP, établissant ce complexe comme un mécanisme par lequel Gal3 est recruté dans la voie splicéosomale16. Cela contraste avec les tentatives d’isoler les spliceosomes à des stades spécifiques de la réaction d’épissage et de catalogage des facteurs associés17,18. Dans de telles études, la présence de certains facteurs à un moment donné est déterminée, mais pas le mécanisme par lequel ils ont été chargés.
Nous avions précédemment décrit en détail la préparation du NE, le substrat d’épissage, l’assemblage du mélange réactionnel d’épissage et l’analyse des produits dans notre documentation sur le rôle des galectines dans l’épissage pré-ARNm19. Nous décrivons maintenant les procédures expérimentales de fractionnement d’extraits nucléaires pour obtenir une fraction enrichie en complexe snRNP Gal3 – U1 et pour l’immuno-sélection de ce dernier complexe afin de reconstituer l’activité d’épissage dans un extrait nucléaire appauvri en U1.
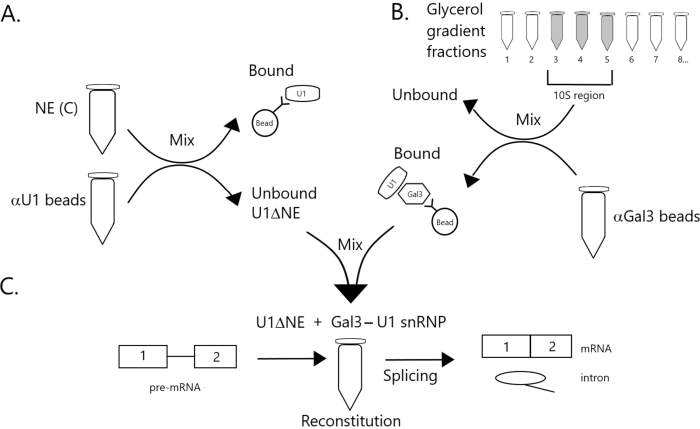
Figure 1 : Schéma illustrant la complémentarité de l’activité d’épissage dans l’extrait nucléaire appauvri en U1 snRNP par un complexe snRNP Gal3-U1 sur des billes. (A) NE dans le tampon C (NE(C)) est incubé avec des billes de protéine A-sépharose couplées de manière covalente avec des billes anti-U1 snRNP (αU1). La fraction non liée est appauvrie en U1 snRNP (U1ΔNE). (B) LE NE dans le tampon D (NE(D)) est fractionné sur un gradient de glycérol de 12 % à 32 % par ultracentrifugation. Les fractions correspondant à la région 10S (fractions 3-5) sont combinées et mélangées avec des billes couplées de manière covalente avec des anticorps anti-Gal3 (billes αGal3). Le matériau lié aux perles contient une monoparticule Gal3-U1 snRNP. (C) Le complexe snRNP Gal3-U1 de la partie (B) est mélangé avec U1ΔNE de la partie (A) dans un essai d’épissage utilisant un substrat pré-ARNm MINX marqué 32P et les intermédiaires et produits de la réaction d’épissage sont analysés par électrophorèse sur gel et autoradiographie. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocol
Representative Results
Discussion
Ce rapport fournit les détails expérimentaux qui documentent un complexe snRNP Gal3 – U1 piégé sur des billes enrobées anti-Gal3 peut se lier au substrat pré-ARNm et ce complexe ternaire peut restaurer l’activité d’épissage à un NE appauvri en U1 snRNP. Gal3 est un membre d’une famille de protéines isolées à l’origine sur la base de son activité de liaison aux glucides spécifiques au galactose23 . Les premières études d’immunofluorescence et de fractionnement subcellulair…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ce travail a été soutenu par la subvention MCB-0092919 de la National Science Foundation et la subvention de recherche intra-muros 09-CDFP-2001 de la Michigan State University (à RJP) et par la subvention GM-38740 des National Institutes of Health et le projet MICL02455 du Michigan AgBioResearch (à JLW).
Le substrat pré-ARNm MINX utilisé dans les essais d’épissage était un cadeau aimable du Dr Susan Berget (Baylor College of Medicine, Houston, TX, États-Unis).
Materials
| anti-U1 snRNP | The Binding Site | Hu ENA-RNP #33471 | human autoimmune serum specific for U1 snRNP |
| bottle top vacuum filter | Fisher Scientific | Corning 431153 (0.22 μm; PES 150 ml) | for filtering solutions containing Tris |
| centrifuge | International Equipment Company | IEC Model PR-6 | for pelletting Sepharose beads in immunoprecipitation |
| diethylpyrocarbonate (DEPC) | Sigma-Aldrich | 159220-5G | for treatment of water used in preparation of all solutions |
| dimethylpimelimidate (DMP) | Sigma-Aldrich | 80490-5G | for cross-linking antibody to Sepharose beads |
| electrophoresis cell | BioRad Laboratories, Inc | Mini-Protean II | for SDS-PAGE separation of proteins |
| ethanolamine | Sigma-Aldrich | 411000-100ml | for blocking after the cross-linking reaction |
| gel electrophoresis system | Hoefer, Inc | HSI SE 500 Series | for separating snRNAs by gel electrophoresis |
| gel slab dryer | BioRad | Model 224 | for drying gel slabs for autoradiography |
| Hybond ECL membrane | GE Healthcare | RPN3032D (0.2 μm; 30 cm x 3 m) | for immunoblotting of proteins on membrane |
| microdialyzer (12 x 100 μl sample capacity) | Pierce | Microdialyzer System 100 | for exchanging the buffer of nuclear extract |
| microdialyzer membranes (8K cutoff) | Pierce | 66310 | for exchanging the buffer of nuclear extract |
| non-fat dry milk | Spartan Stores | Spartan Instant Non-fat Dry Milk | |
| Protein A Sepharose CL-4B | Millipore-Sigma | GE 17-0780-01 | for coupling antibody to beads |
| Proteinase K | Millipore-Sigma | P2308-5mg | for stopping the splicing reaction to isolate the RNAs |
| RNasin | Promega | N2111 | for inhibiting ribonuclease activity |
| rocker/rotator | Lab Industries, Inc | Labquake Shaker 400-110 | for mixing protein solutions in coupling reactions and in immunoprecipitation |
| Safety-Solve | Research Products International Corp. | No. 111177 | scintillation counting cocktail for determination of radioactivity in splicing substrate |
| scintillation counter | Beckman Instruments | LS6000SC | scintillation counter for determination of radioactivity |
| speed vaccum concentrator | Savant | SVC 100H | for drying ethanol-precipitated RNA pellets |
| Transphor electrophoresis unit | Hoefer, Inc | Hoefer TE Series Transphor | for protein transfer from SDS-PAGE to blotting membrane |
References
- Hoskins, A. A., Moore, M. J. The spliceosome: a flexible, reversible macromolecular machine. Trends In Biochemical Sciences. 37, 179-188 (2012).
- Choi, Y. D., Grabowski, P., Sharp, P. A., Dreyfuss, G. Heterogeneous nuclear ribonucleoproteins: role in RNA splicing. Science. 231, 1534-1539 (1986).
- Lerner, M., Steitz, J. A. Snurps and scyrps. Cell. 25, 298-300 (1981).
- Maniatis, T., Reed, R. The role of small nuclear ribonucleoprotein particles in pre-mRNA splicing. Nature. 325, 673-678 (1987).
- Hoskins, A. A., et al. Ordered and dynamic assembly of single spliceosomes. Science. 331, 1289-1295 (2011).
- Coppin, L., Leclerc, J., Vincent, A., Porchet, N., Pigny, P. Messenger RNA life-cycle in cancer: emerging role of conventional and non-conventional RNA-binding proteins. International Journal of Molecular Sciences. 19, 650-676 (2018).
- Dagher, S. F., Wang, J. L., Patterson, R. J. Identification of galectin-3 as a factor in pre-mRNA splicing. Proceedings of the National Academy of Sciences of the United States of America. 92, 1213-1217 (1995).
- Vyakarnam, A., Dagher, S. F., Wang, J. L., Patterson, R. J. Evidence for a role for galectin-1 in pre-mRNA splicing. Molecular and Cellular Biology. 17, 4730-4737 (1997).
- Wang, W., Park, J. W., Wang, J. L., Patterson, R. J. Immunoprecipitation of spliceosomal RNAs by antisera to galectin-1 and galectin-3. Nucleic Acids Research. 34, 5166-5174 (2006).
- Haudek, K. C., Voss, P. G., Locascio, L. E., Wang, J. L., Patterson, R. J. A mechanism for incorporation of galectin-3 into the spliceosome through its association with U1 snRNP. Biochemistry. 48, 7705-7712 (2009).
- Fritsch, K., et al. Galectin-3 interacts with components of the nuclear ribonucleoprotein complex. BMC Cancer. 16, 502-511 (2016).
- Conway, G. C., Krainer, A. R., Spector, D. L., Roberts, R. J. Multiple splicing factors are released from endogenous complexes during in vitro pre-mRNA splicing. Molecular and Cellular Biology. 9, 5273-5280 (1989).
- Dery, K. J., Yean, S. L., Lin, R. J. Assembly and glycerol gradient isolation of yeast spliceosomes containing transcribed or synthetic U6 snRNA. Methods in Molecular Biology. 488, 41-63 (2008).
- Yoshimoto, R., Kataoka, N., Okawa, K., Ohno, M. Isolation and characterization of post-splicing lariat-intron complexes. Nucleic Acids Research. 37, 891-902 (2009).
- Malca, H., Shomron, N., Ast, G. The U1 snRNP base pairs with the 5′ splice site within a penta-snRNP complex. Molecular and Cellular Biology. 23, 3442-3455 (2003).
- Haudek, K. C., Voss, P. G., Wang, J. L., Patterson, R. J. A 10S galectin-3 – snRNP complex assembles into active spliceosomes. Nucleic Acids Research. 44, 6391-6397 (2016).
- Rappsilber, J., Ryder, U., Lamond, A. I., Mann, M. Large-scale proteomic analysis of the human spliceosome. Genome Research. 12, 1231-1245 (2002).
- Jurica, M. S., Moore, M. J. Capturing splicing complexes to study structure and mechanism. Methods. 28, 336-345 (2002).
- Patterson, R. J., Haudek, K. C., Voss, P. G., Wang, J. L. Examination of the role of galectins in pre-mRNA splicing. Methods in Molecular Biology. 1207, 431-449 (2015).
- Dignam, J. D., Lebovitz, R. M., Roeder, R. G. Accurate transcription initiation by RNA polymerase II in a soluble extract from isolated mammalian nuclei. Nucleic Acids Research. 11, 1475-1489 (1983).
- Agarwal, N., Sun, Q., Wang, S. Y., Wang, J. L. Carbohydrate-binding protein 35. I. Properties of the recombinant polypeptide and the individuality of the domains. Journal of Biological Chemistry. 268, 14932 (1993).
- Zillmann, M., Zapp, M. I., Berget, S. M. Gel electrophoretic isolation of splicing complexes containing U1 small nuclear ribonucleoprotein particles. Molecular and Cellular Biology. 8, 814-821 (1988).
- Barondes, S. H., et al. Galectins: a family of animal β-galactoside-binding proteins. Cell. 76, 597-598 (1994).
- Laing, J. G., Wang, J. L. Identification of carbohydrate binding protein 35 in heterogeneous nuclear ribonucleoprotein complex. Biochemistry. 27, 5329-5334 (1988).
- Vyakarnam, A., Lenneman, A. J., Lakkides, K. M., Patterson, R. J., Wang, J. L. A comparative nuclear localization study of galectin-1 with other splicing components. Experimental Cell Research. 242, 419-428 (1998).
- Michaud, S., Reed, R. An ATP-independent complex commits pre-mRNA to the mammalian spliceosome assembly pathway. Genes & Development. 5, 2534-2546 (1991).
- Chiu, Y. -. F., et al. Cwc25 is a novel splicing factor required after Prp2 and Yju2 to facilitate the first catalytic reaction. Molecular and Cellular Biology. 29, 5671-5678 (2009).
- Krishnan, R., et al. Biased Brownian ratcheting leads to pre-mRNA remodeling and capture prior to first-step splicing. Nature Structural and Molecular Biology. 20, 1450-1457 (2013).
- Gray, R. M., et al. Distinct effects on splicing of two monoclonal antibodies directed against the amino-terminal domain of galectin-3. Archives of Biochemistry and Biophysics. 475, 100-108 (2008).

