微生物化学およびタンパク質生産のための光制御発酵
Summary
微生物代謝の光遺伝学的制御は、発酵プロセスに対する柔軟な動的制御を提供する。ここでのプロトコルは、異なる体積スケールでの化学およびタンパク質生産のための青色光調節発酵を設定する方法を示しています。
Abstract
微生物細胞工場は、再生可能な原料から化学物質や組換えタンパク質を生産するための持続可能な代替手段を提供します。しかし、微生物に遺伝子組み換えを過剰に負担をかけると、宿主の適応度と生産性が低下する可能性があります。この問題は、動的制御(酵素および経路の誘導性発現、典型的には化学的または栄養ベースの添加剤を使用して、細胞の成長および産生のバランスをとることによって克服することができる。光遺伝学は、遺伝子発現を動的に調節する非侵襲的、高度に調整可能、かつ可逆的な方法を提供する。ここでは、化学物質または組換えタンパク質の生産のために、操作された 大腸菌 および サッカロミセス・セレビシエ の光制御発酵を設定する方法について説明します。私たちは、発酵制御と生産性を向上させるために微生物の増殖と生産を切り離すために、選択された時間と投与量で光を適用する方法、および最良の結果を得るための主要な最適化考慮事項について説明します。さらに、ラボ規模のバイオリアクター実験のための光制御を実装する方法について説明します。これらのプロトコルは、発酵性能を向上させるために、操作された微生物における光遺伝学的制御の採用を容易にする。
Introduction
光応答性タンパク質による生物学的プロセスの制御であるオプトジェネティクスは、化学的およびタンパク質生産のための微生物発酵を動的に制御する新しい戦略を提供します1,2。操作された代謝経路の負担と、一部の中間体および生成物の毒性は、しばしば細胞増殖を損なう3。このようなストレスは、バイオマスの蓄積不良や生産性の低下につながる可能性があります3。この課題は、発酵を時間的に成長段階と生産段階に分け、代謝資源をバイオマス蓄積または製品合成にそれぞれ捧げることによって対処することができます4。我々は最近、この二相発酵における成長から生産への移行が、照明条件の変化によって誘発され得ることを示した5,6,7。光入力の高い同調性、可逆性、直交性8は、従来の二相発酵の動的制御に使用される化学誘導物質では複製が困難または不可能な光制御発酵に独自の利点を提供します4,9,10,11。
エリスロバクター・リトラリス由来の青色光応答性EL222タンパク質は、サッカロミセス・セレビシエ5,7,12,13における代謝工学のためのいくつかの光遺伝学的回路を開発するために使用されている。EL222は、青色光活性化(465nm)時に立体構造シフトを受ける光酸素電圧センサ(LOV)ドメインを含み、これにより、同族DNA配列(C120)13に結合することができる。EL222をウイルスVP16活性化ドメイン(VP16-EL222)に融合させることで、合成プロモーターPC120からS. cerevisiae7および他の生物14における遺伝子発現を可逆的に活性化することができる青色光応答性転写因子が得られる。EL222に基づくいくつかの回路が開発され、目的の遺伝子がPC120から直接発現される基本的な光活性化OptoEXPシステム7など、S. cerevisiaeの化学生産に使用されています(図1A)。しかし、発酵の生産段階で典型的に遭遇する高い細胞密度での光の浸透の懸念は、OptoINVRTおよびOptoQ-INVRT回路(図1B)5、7、13のような暗闇で誘導される反転回路を開発する動機となった。これらの系は、それぞれセレビシエ菌およびクラッサ菌由来のガラクトース(GAL)またはキナ酸(Q)レギュロンを利用し、対応するリプレッサー(GAL80およびQS)をVP16-EL222で制御して、光では遺伝子発現を抑制し、暗闇では強く誘導します。OptoEXP回路とOptoINVRT回路を組み合わせることで、遺伝子発現の双方向制御が可能になり、成長期が青色光で誘導され、生産期が暗さで誘導される2相発酵が可能になります(図2A)5,7。
生産段階で遺伝子発現を誘導するために暗闇の代わりに光を使用することは、光遺伝学的制御の能力を大幅に拡大するが、発酵のこの段階で典型的に遭遇する高い細胞密度の光浸透制限を克服することも必要である。この目的のために、我々は、青色光刺激に対する転写応答を増幅するOptoAMPおよびOptoQ-AMPとして知られる回路を開発しました。これらの回路は、VP16-EL222の野生型または過敏性変異体を使用して、GALまたはQレギュロンの転写活性化剤Gal4pまたはQF2の産生をそれぞれ制御し、光による感度の向上とより強い遺伝子発現を達成します12,13(図1C)。OptoAMP回路は、光学密度(600nmで測定)で5Lバイオリアクターで完全かつ均質な光誘導を達成することができます。OD600)は、照明のわずか〜0.35%(バルク表面の〜7%のみに5%の光線量)を有する少なくとも40の値である。これは、100%に近い照明を必要とするOptoEXPと比較して、より高い感度を示しています12。高い細胞密度で光で遺伝子発現を効果的に誘導する能力は、発酵の動的制御のための新しい機会を開きます。これには、化学生産を最適化するために独自の光スケジュールで成長、誘導、生産段階が確立される三相発酵など、2つ以上の時間的段階における発酵の操作が含まれます(図2B)12。
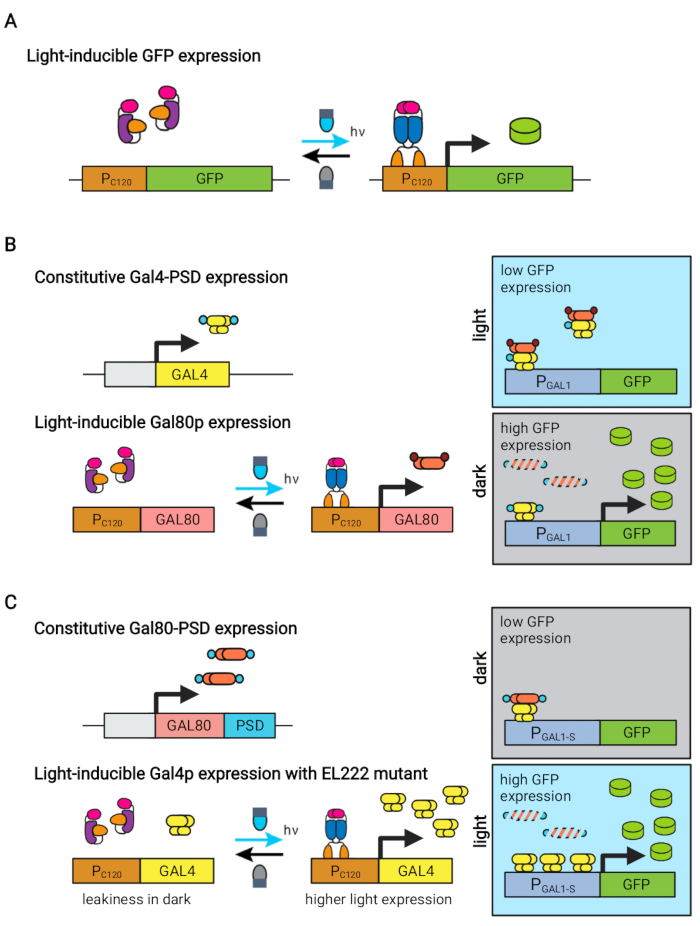
図1:セレビシエ菌の動的制御のための光遺伝学的回路 OptoEXP、OptoINVRT、およびOptoAMP回路は、光に敏感なVP16-EL222システムに基づいています。(A)OptoEXP回路では、青色光への曝露はVP16-EL222の立体構造変化および二量体化を引き起こし、DNA結合ドメインを露出させ、PC120からの転写を可能にする。この図はZhao et al.7から修正されたものである。(B)OptoINVRT回路は、GALまたはQレギュロンを利用して、暗闇での発現を誘導する。GALベースの回路では、VP16-EL222およびGAL4が恒常的に発現され、PC120はGAL80リプレッサーの発現を駆動する(Qベースの回路では、GAL4およびGAL80はそれぞれQF2およびQSに置き換えられ、GALプロモーターの代わりに合成QUAS含有プロモーターが使用される)。光の中で、Gal80pはPGAL1からの目的遺伝子の活性化を妨げる。暗闇では、GAL80は発現されず、構成的デグロンドメイン(小さな茶色のドメイン)に融合することによって急速に分解され、Gal4pによるPGAL1の活性化を可能にする。この図はZhao et al.5から修正されています。(C) OptoAMP回路はVP16-EL222を使用してGALまたはQレギュロンも制御します。これらの回路では、GAL80リプレッサー(またはQS)が恒常的に表現され、光感受性デグロン(小さな青色領域)に融合され、暗闇でのタイトな抑制を保証します。PC120および過敏性VP16−EL222変異体は、光でGAL4(またはQF2)の発現を制御し、光中でPGAL1(またはQUAS含有プロモーター)を強く活性化する。GAL由来の回路は、活性が増大したPGAL1-MやPGAL1-Sなどの操作型PGAL1や、GALレギュロン(PGAL1、PGAL10、PGAL2、PGAL7)によって制御される野生型プロモーターを使用することができる。この図はZhao et al.12から修正されています。この図の拡大版を見るにはここをクリックしてください。
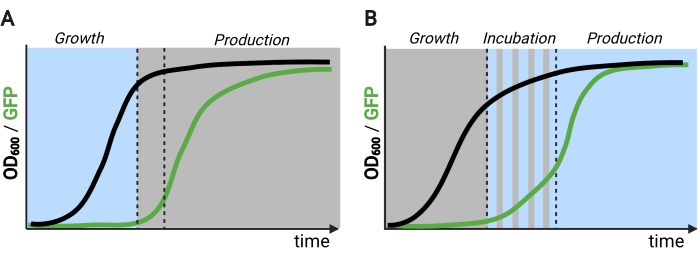
図2:時間の経過に伴う 二相発酵と三相発酵。(A)逆回路で操作される二相発酵は、光駆動の成長段階と暗黒生産段階からなる。成長段階では、生産経路が抑制されたままであるため、バイオマスは蓄積します。所望のOD600に達すると、細胞は、産生段階のために新鮮な培地に再懸濁される前に代謝的に調整するために暗所にシフトされる。(B)三相プロセスにおいて、成長、インキュベーション、および生産段階は、暗い成長期間、パルスインキュベーション、および完全に照らされた生産段階からなることができる固有の光スケジュールによって定義される。バイオレンダーで作成した図。 この図の拡大版を表示するには、ここをクリックしてください。
光遺伝学的回路は、大腸菌における化学的およびタンパク質産生の動的制御のためにも開発されている。OptoLAC回路は、YF1/FixJ 2成分系をベースとする光応答性pDawn回路を使用して、細菌のLacIリプレッサを制御します6(図3)。OptoINVRT5と同様に、OptoLAC回路は、光の中で遺伝子発現を抑制し、暗闇の中でそれを誘導するように設計されています。OptoLAC回路を用いた発現レベルは、標準的なイソプロピルβ-d-1-チオガラクトピラノシド(IPTG)誘導で達成された発現レベルに匹敵するか、それを超えることができるため、化学的誘導の強度を維持しながら、同調性と可逆性を高めます6。したがって、OptoLAC回路は、大腸菌における代謝工学のための効果的な光遺伝学的制御を可能にする。
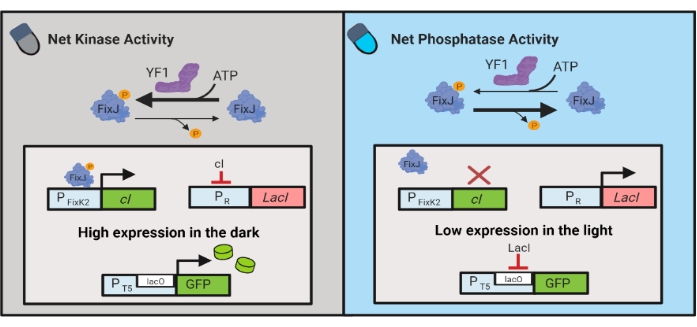
図3:大腸菌の動的制御のためのOptoLAC回路。 OptoLAC回路は、pDawnシステムとlacオペロンを適応させて、暗闇の中での活性化と光での抑圧を実現します。暗所では、YF1はFixJをリン酸化し、PFixK2プロモーターを活性化してcIリプレッサーを発現させる。cIリプレッサーは、PRプロモーターからのlacIリプレッサーの発現を防止し、lacO含有プロモーターからの目的遺伝子の転写を可能にする。逆に、青色光はYF1ネットキナーゼ活性を低下させ、FixJリン酸化を逆転させ、したがってcI発現を逆転させ、lacIの発現を抑制し、lacO含有プロモーターからの発現を妨げる。この図はLalwani et al.6から修正されたものである。この図の拡大版を表示するには、ここをクリックしてください。
ここでは、化学またはタンパク質生産のための セレビシエ 菌および 大腸菌 の光制御発酵のための基本的なプロトコルについて説明します。酵母とバクテリアの両方について、まず、光駆動の増殖段階と、OptoINVRTおよびOptoLAC回路によって可能になる暗闇誘発生産段階を有する発酵に焦点を当てています。続いて、OptoAMP回路によって可能になる三相(成長、誘導、生産)光制御発酵のプロトコルについて説明します。さらに、光遺伝学的に制御された発酵をマイクロプレートからラボ規模のバイオリアクターにスケールアップする方法についても説明します。このプロトコルにより、化学的またはタンパク質生産のための光制御発酵を実行するための完全で再現性の高いガイドを提供することを目指しています。
Protocol
Representative Results
Discussion
動的制御は、代謝工学と組換えタンパク質生産の収量を改善するために長い間適用されてきました4。酵素発現の変化は、IPTG21、ガラクトース22、およびテトラサイクリン23などの化学誘導物質を用いて最も典型的には実施されるが、温度およびpHなどのプロセス条件を用いて媒介される。遺伝子発現の光遺伝学的制御によ…
Disclosures
The authors have nothing to disclose.
Acknowledgements
この研究は、米国エネルギー省科学局、生物環境研究局賞番号DE-SC0019363、NSF CAREER賞CBET-1751840、ピュー慈善信託、およびカミーユドレフュス教師学者賞の支援を受けました。
Materials
| Light-controlled chemical production using S. cerevisiae | |||
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Agar powder | Thermo Fisher Scientific | 303991049 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| EZ-L439 OptoINVRT7 Plasmid | N/A | N/A | See Reference 1 |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| PmeI | New England Biolabs | R0560L | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| Replica-plating device | Thomas Scientific | F37848-0000 | |
| Replica-plating pads | Sunrise Science Products | 3005-012 | |
| SC-His powder | Sunrise Science Products | 1303-030 | |
| SC Complete powder | Sunrise Science Products | 1459-100 | |
| Sterile sealing film | Excel Scientific | STR-SEAL-PLT | |
| YPD agar plates | VWR | 100217-054 | |
| Zeocin | Thermo Fisher Scientific | R25005 | |
| Light-controlled protein production using E. coli | |||
| 6X SDS Sample Buffer | Cepham Life Sciences | 10502 | |
| 12% Acrylamide protein gels | Thermo Fisher Scientific | NP0341BOX | |
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| Coomassie Brilliant Blue G-250 | Thermo Fisher Scientific | 20279 | |
| Electrophoresis cell | Bio-Rad | 1658004 | |
| Electrophoresis power supply | Bio-Rad | 1645050 | |
| LB broth (Miller) | Fisher Scientific | BP97235 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NaCl | Thomas Scientific | SX0425-1 | |
| OptoLAC plasmids | N/A | N/A | See Reference 2 |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Thermomixer | Eppendorf | 5382000015 | |
| Tris base | Fisher Scientific | BP1521 | |
| Three-phase fermentation using S. cerevisiae | |||
| Same materials as "Light-controlled chemical production using S. cerevisiae" protocol plus the following: | |||
| EZ-L580 OptoAMP4 Plasmid | N/A | N/A | See Reference 10 |
| Chemical production in a light-controlled bioreactor | |||
| Aluminum foil | Reynolds | B004NG90YO | |
| Antifoam | Sigma-Aldrich | A8311 | |
| Bioreactor with control station | Eppendorf | B120110001 | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Bleach | VWR Scientific | 89501-620 (CS) | |
| Blue LED panel | HQRP | 884667106091218 | |
| BPT tubing | Fisher Scientific | 14-170-15 | |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Hydrochloric acid (HCl) | Fisher Scientific | 7647-01-0 | |
| M9 Minimal Salts | Thermo Fisher Scientific | A1374401 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NH4OH Solution | Sigma-Aldrich | I0503-1VL | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SC Complete powder | Sunrise Science Products | 1459-100 |
References
- Figueroa, D., Rojas, V., Romero, A., Larrondo, L. F., Salinas, F. The rise and shine of yeast optogenetics. Yeast. 38 (2), 131-146 (2021).
- Pouzet, S., et al. The promise of optogenetics for bioproduction: Dynamic control strategies and scale-up instruments. Bioengineering. 7 (4), 151 (2020).
- Venayak, N., Anesiadis, N., Cluett, W. R., Mahadevan, R. Engineering metabolism through dynamic control. Current Opinion in Biotechnology. 34, 142-152 (2015).
- Lalwani, M. A., Zhao, E. M., Avalos, J. L. Current and future modalities of dynamic control in metabolic engineering. Current Opinion in Biotechnology. 52, 56-65 (2018).
- Zhao, E. M., et al. Design and characterization of rapid optogenetic circuits for dynamic control in yeast metabolic engineering. ACS Synthetic Biology. 9 (12), 3254-3266 (2020).
- Lalwani, M. A., et al. Optogenetic control of the lac operon for bacterial chemical and protein production. Nature Chemical Biology. 17 (1), 71-79 (2021).
- Zhao, E. M., et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature. 555 (7698), 683-687 (2018).
- Baumschlager, A., Khammash, M. Synthetic biological approaches for optogenetics and tools for transcriptional light-control in bacteria. Advanced Biology. 5 (5), 2000256 (2021).
- Dvorak, P., et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway. Microbial Cell Factories. 14, 201 (2015).
- Hartline, C. J., Schmitz, A. C., Han, Y., Zhang, F. Dynamic control in metabolic engineering: Theories, tools, and applications. Metabolic Engineering. 63, 126-140 (2021).
- Ni, C., Dinh, C. V., Prather, K. L. J. Dynamic control of metabolism. Annual Review of Chemical and Biomolecular Engineering. 12, 519-560 (2021).
- Zhao, E. M., et al. Optogenetic amplification circuits for light-induced metabolic control. ACS Synthetic Biology. 10 (5), 1143-1154 (2021).
- Lalwani, M. A., Zhao, E. M., Wegner, S. A., Avalos, J. L. The Neurospora crassa Inducible Q System Enables Simultaneous Optogenetic Amplification and Inversion in Saccharomyces cerevisiae for Bidirectional Control of Gene Expression. ACS Synthetic Biology. 10 (8), 2060-2075 (2021).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Marx, H., Mecklenbräuker, A., Gasser, B., Sauer, M., Mattanovich, D. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Research. 9 (8), 1260-1270 (2009).
- Nordén, K., et al. Increasing gene dosage greatly enhances recombinant expression of aquaporins in Pichia pastoris. BMC Biotechnology. 11, 47 (2011).
- Zhao, E. M., et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nature Chemical Biology. 15 (6), 589-597 (2019).
- Dowee, W. J., Miller, J. F., Ragsdale, C. W. High efficiency transformation of E. coli by high voltage electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Zhou, K., Edgar, S., Stephanopoulos, G. Engineering microbes to synthesize plant isoprenoids. Methods in Enzymology. 575, 225-245 (2016).
- Arfman, N., Worrell, V., Ingram, L. O. Use of the tac promoter and lacI(q) for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis. Journal of Bacteriology. 174 (22), 7370-7378 (1992).
- Steen, E. J., et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microbial Cell Factories. 7 (1), 1-8 (2008).
- Tan, S. Z., Manchester, S., Prather, K. L. J. Controlling central carbon metabolism for improved pathway yields in Saccharomyces cerevisiae. ACS Synthetic Biology. 5 (2), 116-124 (2015).
- Jayaraman, P., et al. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research. 44 (14), 6994 (2016).
- Fernandez-Rodriguez, J., Moser, F., Song, M., Voigt, C. A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology. 13 (7), 706-708 (2017).
- Ding, Q., et al. Light-powered Escherichia coli cell division for chemical production. Nature Communications. 11 (1), 1-14 (2020).
- Senoo, S., Tandar, S. T., Kitamura, S., Toya, Y., Shimizu, H. Light-inducible flux control of triosephosphate isomerase on glycolysis in Escherichia coli. Biotechnology and Bioengineering. 116 (12), 3292-3300 (2019).
- Ramakrishnan, P., Tabor, J. J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. Coli. ACS Synthetic Biology. 5 (7), 733-740 (2016).
- Tabor, J. J., Levskaya, A., Voigt, C. A. Multichromatic control of gene expression in escherichia coli. Journal of Molecular Biology. 405 (2), 315-324 (2011).
- Stewart, C. J., McClean, M. N. Design and implementation of an automated illuminating, culturing, and sampling system for microbial optogenetic applications. Journal of Visualized Experiments:JoVE. (120), e54894 (2017).
- Grødem, E. O. S., Sweeney, K., McClean, M. N. Automated calibration of optoPlate LEDs to reduce light dose variation in optogenetic experiments. BioTechniques. 69 (4), 313-316 (2020).
- Gerhardt, K. P., et al. An open-hardware platform for optogenetics and photobiology. Scientific Reports. 6, (2016).
- Bugaj, L. J., Lim, W. A. High-throughput multicolor optogenetics in microwell plates. Nature Protocols. 14 (7), 2205-2228 (2019).
- Steel, H., Habgood, R., Kelly, C., Papachristodoulou, A. In situ characterisation and manipulation of biological systems with Chi.Bio. PLoS Biology. 18 (7), (2020).
- Carrasco-López, C., García-Echauri, S. A., Kichuk, T., Avalos, J. L. Optogenetics and biosensors set the stage for metabolic cybergenetics. Current Opinion in Biotechnology. 65, 296-309 (2020).
- Milias-Argeitis, A., Rullan, M., Aoki, S. K., Buchmann, P., Khammash, M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications. 7 (1), 1-11 (2016).
- Melendez, J., et al. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology: Quantitative Biosciences From Nano to Macro. 6 (3), 366-372 (2014).
- Milias-Argeitis, A., et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology. 29 (12), 1114-1116 (2011).
- Castillo-Hair, S. M., Baerman, E. A., Fujita, M., Igoshin, O. A., Tabor, J. J. Optogenetic control of Bacillus subtilis gene expression. Nature Communications. 10 (1), 1-11 (2019).
- Xia, A., et al. Optogenetic modification of pseudomonas aeruginosa enables controllable twitching motility and host infection. ACS Synthetic Biology. 10 (3), 531-541 (2021).
- Pu, L., Yang, S., Xia, A., Jin, F. Optogenetics manipulation enables prevention of biofilm formation of engineered pseudomonas aeruginosa on surfaces. ACS Synthetic Biology. 7 (1), 200-208 (2018).

