Fermentaciones controladas por la luz para la producción química microbiana y de proteínas
Summary
El control optogenético del metabolismo microbiano ofrece un control dinámico flexible sobre los procesos de fermentación. El protocolo aquí muestra cómo configurar fermentaciones reguladas por luz azul para la producción química y de proteínas a diferentes escalas volumétricas.
Abstract
Las fábricas de células microbianas ofrecen una alternativa sostenible para producir productos químicos y proteínas recombinantes a partir de materias primas renovables. Sin embargo, sobrecargar un microorganismo con modificaciones genéticas puede reducir la aptitud y la productividad del huésped. Este problema se puede superar mediante el uso de control dinámico: expresión inducible de enzimas y vías, típicamente utilizando aditivos químicos o a base de nutrientes, para equilibrar el crecimiento y la producción celular. Optogenetics ofrece un método no invasivo, altamente ajustable y reversible para regular dinámicamente la expresión génica. Aquí, describimos cómo configurar fermentaciones controladas por la luz de Escherichia coli y Saccharomyces cerevisiae modificadas para la producción de productos químicos o proteínas recombinantes. Discutimos cómo aplicar luz en momentos y dosis seleccionados para desacoplar el crecimiento microbiano y la producción para mejorar el control de la fermentación y la productividad, así como las consideraciones clave de optimización para obtener los mejores resultados. Además, describimos cómo implementar controles de luz para experimentos de biorreactores a escala de laboratorio. Estos protocolos facilitan la adopción de controles optogenéticos en microorganismos diseñados para mejorar el rendimiento de la fermentación.
Introduction
La optogenética, el control de procesos biológicos con proteínas sensibles a la luz, ofrece una nueva estrategia para controlar dinámicamente las fermentaciones microbianas para la producción química y proteica1,2. La carga de las vías metabólicas modificadas y la toxicidad de algunos productos intermedios y productos a menudo perjudican el crecimiento celular3. Tales tensiones pueden conducir a una acumulación deficiente de biomasa y a una reducción de la productividad3. Este desafío puede abordarse dividiendo temporalmente las fermentaciones en una fase de crecimiento y producción, que dedican recursos metabólicos a la acumulación de biomasa o a la síntesis de productos, respectivamente4. Recientemente demostramos que la transición del crecimiento a la producción en esta fermentación bifásica puede ser inducida con cambios en las condiciones de iluminación5,6,7. La alta capacidad de ajuste, reversibilidad y ortogonalidad de las entradas de luz8 ofrece ventajas únicas a las fermentaciones controladas por la luz que son difíciles o imposibles de replicar con inductores químicos utilizados en el control dinámico de fermentaciones bifásicas convencionales4,9,10,11.
La proteína EL222 sensible a la luz azul derivada de Erythrobacter litoralis se ha utilizado para desarrollar varios circuitos optogenéticos para la ingeniería metabólica en Saccharomyces cerevisiae5,7,12,13. EL222 contiene un dominio sensor de voltaje de luz-oxígeno (LOV) que sufre un cambio conformacional tras la activación de la luz azul (465 nm), lo que le permite unirse a su secuencia de ADN cognado (C120)13. La fusión de EL222 al dominio de activación viral VP16 (VP16-EL222) da como resultado un factor de transcripción sensible a la luz azul que puede activar reversiblemente la expresión génica en S. cerevisiae7 y otros organismos14 a partir del promotor sintético PC120. Varios circuitos basados en EL222 han sido desarrollados y utilizados para la producción química en S. cerevisiae, como el sistema básico OptoEXP activado por luz7, en el que el gen de interés se expresa directamente desde PC120 (Figura 1A). Sin embargo, las preocupaciones de penetración de la luz en las altas densidades celulares típicamente encontradas en la fase de producción de las fermentaciones nos motivaron a desarrollar circuitos invertidos que se inducen en la oscuridad, como los circuitos OptoINVRT y OptoQ-INVRT (Figura 1B)5,7,13. Estos sistemas aprovechan los reguleones de galactosa (GAL) o ácido quínico (Q) de S. cerevisiae y N. crassa, respectivamente, controlando sus correspondientes represores (GAL80 y QS) con VP16-EL222, para reprimir la expresión génica a la luz e inducirla fuertemente en la oscuridad. La combinación de los circuitos OptoEXP y OptoINVRT da como resultado un control bidireccional de la expresión génica, permitiendo fermentaciones bifásicas en las que la fase de crecimiento se induce con luz azul y la fase de producción con oscuridad (Figura 2A)5,7.
El uso de la luz en lugar de la oscuridad para inducir la expresión génica durante la fase de producción ampliaría en gran medida las capacidades de los controles optogenéticos, pero también requeriría superar las limitaciones de penetración de la luz de las altas densidades celulares que se encuentran típicamente en esta fase de fermentación. Para ello, hemos desarrollado circuitos, conocidos como OptoAMP y OptoQ-AMP, que amplifican la respuesta transcripcional a la estimulación de la luz azul. Estos circuitos utilizan mutantes de tipo salvaje o hipersensibles de VP16-EL222 para controlar la producción de los activadores transcripcionales Gal4p o QF2 de los reguleones GAL o Q, respectivamente, logrando una mayor sensibilidad y una expresión génica más fuerte con luz12,13 (Figura 1C). Los circuitos OptoAMP pueden lograr una inducción de luz completa y homogénea en biorreactores de 5 L a una densidad óptica (medida a 600 nm; OD600) valores de al menos 40 con solo ~ 0.35% de iluminación (dosis de luz del 5% en solo ~ 7% de la superficie a granel). Esto demuestra un mayor grado de sensibilidad en comparación con OptoEXP, que requiere cerca del 100% de iluminación12. La capacidad de inducir eficazmente la expresión génica con luz a altas densidades celulares abre nuevas oportunidades para el control dinámico de las fermentaciones. Esto incluye fermentaciones operativas en más de dos fases temporales, como las fermentaciones trifásicas, en las que se establecen fases de crecimiento, inducción y producción con programas de luz únicos para optimizar la producción química (Figura 2B)12.
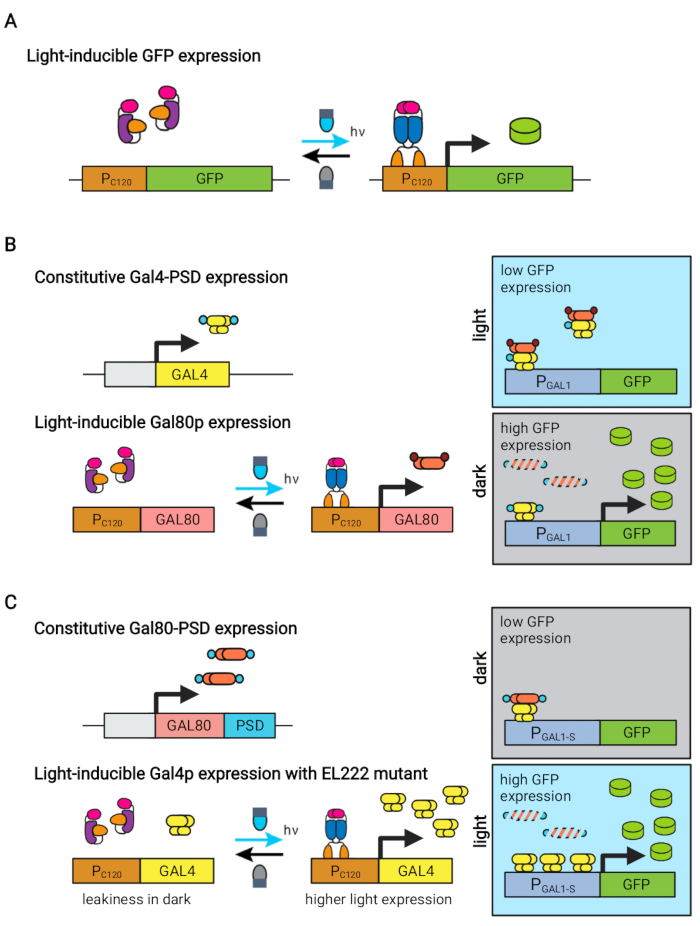
Figura 1: Circuitos optogenéticos para el control dinámico de S. cerevisiae. Los circuitos OptoEXP, OptoINVRT y OptoAMP se basan en el sistema VP16-EL222 sensible a la luz. (A) En el circuito OptoEXP, la exposición a la luz azul causa un cambio conformacional y dimerización de VP16-EL222, que expone un dominio de unión al ADN y permite la transcripción desde PC120. La cifra ha sido modificada a partir de Zhao et al.7. (B) Los circuitos OptoINVRT aprovechan los regulones GAL (mostrados) o Q para inducir la expresión en la oscuridad. En los circuitos basados en GAL, VP16-EL222 y GAL4 se expresan constitutivamente, mientras que PC120 impulsa la expresión del represor GAL80 (en los circuitos basados en Q, GAL4 y GAL80 se reemplazan por QF2 y QS, respectivamente, y se utiliza un promotor sintético que contiene QUAS en lugar de un promotor GAL). A la luz, Gal80p previene la activación del gen de interés de PGAL1. En la oscuridad, GAL80 no se expresa y se degrada rápidamente fusionándolo a un dominio degron constitutivo (pequeño dominio marrón), que permite la activación de PGAL1 por Gal4p. La cifra ha sido modificada a partir de Zhao et al.5. (C) Los circuitos OptoAMP también utilizan VP16-EL222 para controlar los reguleones GAL (mostrados) o Q. En estos circuitos, el represor GAL80 (o QS) se expresa constitutivamente y se fusiona con un degron fotosensible (pequeño dominio azul) que garantiza una represión estricta en la oscuridad. PC120 y una expresión de control mutante hipersensible VP16-EL222 de GAL4 (o QF2) con luz, que activa fuertemente PGAL1 (o un promotor que contiene QUAS) en la luz. Los circuitos derivados de GAL pueden utilizar formas de ingeniería de PGAL1, como PGAL1-M o PGAL1-S, que tienen una mayor actividad, así como promotores de tipo salvaje controlados por el regulón GAL (PGAL1, PGAL10, PGAL2, PGAL7). La figura ha sido modificada de Zhao et al.12. Haga clic aquí para ver una versión más grande de esta figura.
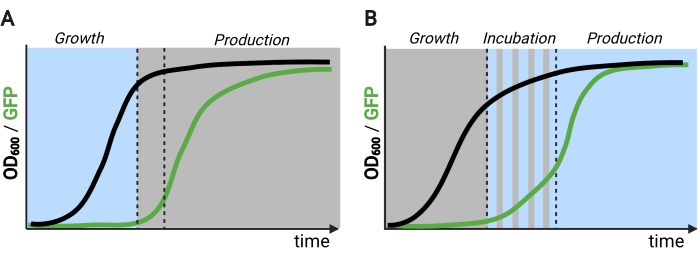
Figura 2: Fermentaciones bifásicas y trifásicas a través del tiempo. (A) Las fermentaciones bifásicas operadas con circuitos invertidos consisten en una fase de crecimiento impulsada por la luz y una fase de producción oscura. En la fase de crecimiento, la biomasa se acumula a medida que la vía de producción permanece reprimida. Al alcanzar el OD600 deseado, las células se desplazan a la oscuridad para ajustarse metabólicamente antes de ser resuspendidas en medios frescos para la fase de producción. (B) En un proceso de tres fases, las fases de crecimiento, incubación y producción están definidas por programas de luz únicos, que pueden consistir en un período de crecimiento oscuro, incubación pulsada y fase de producción completamente iluminada. Figura creada con Biorender. Haga clic aquí para ver una versión más grande de esta figura.
También se han desarrollado circuitos optogenéticos para el control dinámico de la producción química y proteica en E. coli. Los circuitos OptoLAC controlan el represor bacteriano LacI utilizando el circuito pDawn sensible a la luz, que se basa en el sistema de dos componentes YF1/FixJ6 (Figura 3). Al igual que OptoINVRT5, los circuitos OptoLAC están diseñados para reprimir la expresión génica en la luz e inducirla en la oscuridad. Los niveles de expresión que utilizan circuitos OptoLAC pueden igualar o superar los alcanzados con la inducción estándar de isopropilo β-d-1-tiogalactopiranósido (IPTG), manteniendo así la fuerza de la inducción química al tiempo que ofrece una mayor capacidad de ajuste y reversibilidad6. Por lo tanto, los circuitos OptoLAC permiten un control optogenético efectivo para la ingeniería metabólica en E. coli.
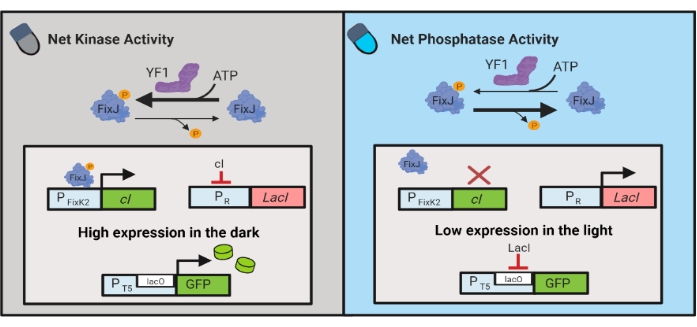
Figura 3: Circuitos OptoLAC para el control dinámico de E. coli. Los circuitos OptoLAC adaptan el sistema pDawn y el operón lac para lograr la activación en la oscuridad y la represión en la luz. En la oscuridad, YF1 fosforila FixJ, que luego activa el promotor PFixK2 para expresar el represor cI . El represor cI impide la expresión del represor lacI del promotor PR , lo que permite la transcripción del gen de interés a partir de un promotor que contiene lacO. Por el contrario, la luz azul reduce la actividad de la quinasa neta YF1, revirtiendo la fosforilación de FixJ y, por lo tanto, la expresión de cI , lo que desreprime la expresión de lacI e impide la expresión del promotor que contiene lacO. La figura ha sido modificada a partir de Lalwani et al.6. Haga clic aquí para ver una versión más grande de esta figura.
Describimos aquí los protocolos básicos para fermentaciones controladas por la luz de S. cerevisiae y E. coli para la producción química o proteica. Tanto para levaduras como para bacterias, primero nos centramos en fermentaciones con una fase de crecimiento impulsada por la luz y una fase de producción inducida por la oscuridad habilitada por los circuitos OptoINVRT y OptoLAC. Posteriormente, describimos un protocolo para una fermentación trifásica (crecimiento, inducción, producción) controlada por la luz habilitada por los circuitos OptoAMP. Además, describimos cómo escalar fermentaciones controladas optogenéticamente desde microplacas hasta biorreactores a escala de laboratorio. Con este protocolo, nuestro objetivo es proporcionar una guía completa y fácilmente reproducible para la realización de fermentaciones controladas por la luz para la producción química o proteica.
Protocol
Representative Results
Discussion
El control dinámico se ha aplicado durante mucho tiempo para mejorar los rendimientos de la ingeniería metabólica y la producción de proteínas recombinantes4. Los cambios en la expresión enzimática se implementan más típicamente utilizando inductores químicos como IPTG21, galactosa22 y tetraciclina23, pero también se han mediado utilizando condiciones de proceso como la temperatura y el pH. El control optogenético …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Esta investigación fue apoyada por el Departamento de Energía de los Estados Unidos, la Oficina de Ciencias, el Premio de la Oficina de Investigación Biológica y Ambiental Número DE-SC0019363, el Premio NSF CAREER CBET-1751840, The Pew Charitable Trusts y el Premio Camille Dreyfus Teacher-Scholar.
Materials
| Light-controlled chemical production using S. cerevisiae | |||
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Agar powder | Thermo Fisher Scientific | 303991049 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| EZ-L439 OptoINVRT7 Plasmid | N/A | N/A | See Reference 1 |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| PmeI | New England Biolabs | R0560L | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| Replica-plating device | Thomas Scientific | F37848-0000 | |
| Replica-plating pads | Sunrise Science Products | 3005-012 | |
| SC-His powder | Sunrise Science Products | 1303-030 | |
| SC Complete powder | Sunrise Science Products | 1459-100 | |
| Sterile sealing film | Excel Scientific | STR-SEAL-PLT | |
| YPD agar plates | VWR | 100217-054 | |
| Zeocin | Thermo Fisher Scientific | R25005 | |
| Light-controlled protein production using E. coli | |||
| 6X SDS Sample Buffer | Cepham Life Sciences | 10502 | |
| 12% Acrylamide protein gels | Thermo Fisher Scientific | NP0341BOX | |
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| Coomassie Brilliant Blue G-250 | Thermo Fisher Scientific | 20279 | |
| Electrophoresis cell | Bio-Rad | 1658004 | |
| Electrophoresis power supply | Bio-Rad | 1645050 | |
| LB broth (Miller) | Fisher Scientific | BP97235 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NaCl | Thomas Scientific | SX0425-1 | |
| OptoLAC plasmids | N/A | N/A | See Reference 2 |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Thermomixer | Eppendorf | 5382000015 | |
| Tris base | Fisher Scientific | BP1521 | |
| Three-phase fermentation using S. cerevisiae | |||
| Same materials as "Light-controlled chemical production using S. cerevisiae" protocol plus the following: | |||
| EZ-L580 OptoAMP4 Plasmid | N/A | N/A | See Reference 10 |
| Chemical production in a light-controlled bioreactor | |||
| Aluminum foil | Reynolds | B004NG90YO | |
| Antifoam | Sigma-Aldrich | A8311 | |
| Bioreactor with control station | Eppendorf | B120110001 | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Bleach | VWR Scientific | 89501-620 (CS) | |
| Blue LED panel | HQRP | 884667106091218 | |
| BPT tubing | Fisher Scientific | 14-170-15 | |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Hydrochloric acid (HCl) | Fisher Scientific | 7647-01-0 | |
| M9 Minimal Salts | Thermo Fisher Scientific | A1374401 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NH4OH Solution | Sigma-Aldrich | I0503-1VL | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SC Complete powder | Sunrise Science Products | 1459-100 |
References
- Figueroa, D., Rojas, V., Romero, A., Larrondo, L. F., Salinas, F. The rise and shine of yeast optogenetics. Yeast. 38 (2), 131-146 (2021).
- Pouzet, S., et al. The promise of optogenetics for bioproduction: Dynamic control strategies and scale-up instruments. Bioengineering. 7 (4), 151 (2020).
- Venayak, N., Anesiadis, N., Cluett, W. R., Mahadevan, R. Engineering metabolism through dynamic control. Current Opinion in Biotechnology. 34, 142-152 (2015).
- Lalwani, M. A., Zhao, E. M., Avalos, J. L. Current and future modalities of dynamic control in metabolic engineering. Current Opinion in Biotechnology. 52, 56-65 (2018).
- Zhao, E. M., et al. Design and characterization of rapid optogenetic circuits for dynamic control in yeast metabolic engineering. ACS Synthetic Biology. 9 (12), 3254-3266 (2020).
- Lalwani, M. A., et al. Optogenetic control of the lac operon for bacterial chemical and protein production. Nature Chemical Biology. 17 (1), 71-79 (2021).
- Zhao, E. M., et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature. 555 (7698), 683-687 (2018).
- Baumschlager, A., Khammash, M. Synthetic biological approaches for optogenetics and tools for transcriptional light-control in bacteria. Advanced Biology. 5 (5), 2000256 (2021).
- Dvorak, P., et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway. Microbial Cell Factories. 14, 201 (2015).
- Hartline, C. J., Schmitz, A. C., Han, Y., Zhang, F. Dynamic control in metabolic engineering: Theories, tools, and applications. Metabolic Engineering. 63, 126-140 (2021).
- Ni, C., Dinh, C. V., Prather, K. L. J. Dynamic control of metabolism. Annual Review of Chemical and Biomolecular Engineering. 12, 519-560 (2021).
- Zhao, E. M., et al. Optogenetic amplification circuits for light-induced metabolic control. ACS Synthetic Biology. 10 (5), 1143-1154 (2021).
- Lalwani, M. A., Zhao, E. M., Wegner, S. A., Avalos, J. L. The Neurospora crassa Inducible Q System Enables Simultaneous Optogenetic Amplification and Inversion in Saccharomyces cerevisiae for Bidirectional Control of Gene Expression. ACS Synthetic Biology. 10 (8), 2060-2075 (2021).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Marx, H., Mecklenbräuker, A., Gasser, B., Sauer, M., Mattanovich, D. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Research. 9 (8), 1260-1270 (2009).
- Nordén, K., et al. Increasing gene dosage greatly enhances recombinant expression of aquaporins in Pichia pastoris. BMC Biotechnology. 11, 47 (2011).
- Zhao, E. M., et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nature Chemical Biology. 15 (6), 589-597 (2019).
- Dowee, W. J., Miller, J. F., Ragsdale, C. W. High efficiency transformation of E. coli by high voltage electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Zhou, K., Edgar, S., Stephanopoulos, G. Engineering microbes to synthesize plant isoprenoids. Methods in Enzymology. 575, 225-245 (2016).
- Arfman, N., Worrell, V., Ingram, L. O. Use of the tac promoter and lacI(q) for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis. Journal of Bacteriology. 174 (22), 7370-7378 (1992).
- Steen, E. J., et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microbial Cell Factories. 7 (1), 1-8 (2008).
- Tan, S. Z., Manchester, S., Prather, K. L. J. Controlling central carbon metabolism for improved pathway yields in Saccharomyces cerevisiae. ACS Synthetic Biology. 5 (2), 116-124 (2015).
- Jayaraman, P., et al. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research. 44 (14), 6994 (2016).
- Fernandez-Rodriguez, J., Moser, F., Song, M., Voigt, C. A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology. 13 (7), 706-708 (2017).
- Ding, Q., et al. Light-powered Escherichia coli cell division for chemical production. Nature Communications. 11 (1), 1-14 (2020).
- Senoo, S., Tandar, S. T., Kitamura, S., Toya, Y., Shimizu, H. Light-inducible flux control of triosephosphate isomerase on glycolysis in Escherichia coli. Biotechnology and Bioengineering. 116 (12), 3292-3300 (2019).
- Ramakrishnan, P., Tabor, J. J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. Coli. ACS Synthetic Biology. 5 (7), 733-740 (2016).
- Tabor, J. J., Levskaya, A., Voigt, C. A. Multichromatic control of gene expression in escherichia coli. Journal of Molecular Biology. 405 (2), 315-324 (2011).
- Stewart, C. J., McClean, M. N. Design and implementation of an automated illuminating, culturing, and sampling system for microbial optogenetic applications. Journal of Visualized Experiments:JoVE. (120), e54894 (2017).
- Grødem, E. O. S., Sweeney, K., McClean, M. N. Automated calibration of optoPlate LEDs to reduce light dose variation in optogenetic experiments. BioTechniques. 69 (4), 313-316 (2020).
- Gerhardt, K. P., et al. An open-hardware platform for optogenetics and photobiology. Scientific Reports. 6, (2016).
- Bugaj, L. J., Lim, W. A. High-throughput multicolor optogenetics in microwell plates. Nature Protocols. 14 (7), 2205-2228 (2019).
- Steel, H., Habgood, R., Kelly, C., Papachristodoulou, A. In situ characterisation and manipulation of biological systems with Chi.Bio. PLoS Biology. 18 (7), (2020).
- Carrasco-López, C., García-Echauri, S. A., Kichuk, T., Avalos, J. L. Optogenetics and biosensors set the stage for metabolic cybergenetics. Current Opinion in Biotechnology. 65, 296-309 (2020).
- Milias-Argeitis, A., Rullan, M., Aoki, S. K., Buchmann, P., Khammash, M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications. 7 (1), 1-11 (2016).
- Melendez, J., et al. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology: Quantitative Biosciences From Nano to Macro. 6 (3), 366-372 (2014).
- Milias-Argeitis, A., et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology. 29 (12), 1114-1116 (2011).
- Castillo-Hair, S. M., Baerman, E. A., Fujita, M., Igoshin, O. A., Tabor, J. J. Optogenetic control of Bacillus subtilis gene expression. Nature Communications. 10 (1), 1-11 (2019).
- Xia, A., et al. Optogenetic modification of pseudomonas aeruginosa enables controllable twitching motility and host infection. ACS Synthetic Biology. 10 (3), 531-541 (2021).
- Pu, L., Yang, S., Xia, A., Jin, F. Optogenetics manipulation enables prevention of biofilm formation of engineered pseudomonas aeruginosa on surfaces. ACS Synthetic Biology. 7 (1), 200-208 (2018).

