Imaging ad autofluorescenza per valutare il metabolismo cellulare
Summary
Questo protocollo descrive l’imaging a fluorescenza e l’analisi dei coenzimi metabolici endogeni, della nicotinamide adenina ridotta (fosfato), del dinucleotide (NAD(P)H) e dell’adenina dinucleotide della flavina ossidata (FAD). L’imaging a autofluorescenza di NAD(P)H e FAD fornisce un metodo non distruttivo privo di etichette per valutare il metabolismo cellulare.
Abstract
Il metabolismo cellulare è il processo attraverso il quale le cellule generano energia e molte malattie, incluso il cancro, sono caratterizzate da un metabolismo anormale. Nicotinamide adenina (fosfato) dinucleotide (NAD(P)H) ridotta e flavina adenina dinucleotide ossidata (FAD) sono coenzimi delle reazioni metaboliche. NAD(P)H e FAD mostrano autofluorescenza e possono essere isolati spettralmente da lunghezze d’onda di eccitazione ed emissione. Entrambi i coenzimi, NAD(P)H e FAD, possono esistere in una configurazione libera o legata alle proteine, ognuno dei quali ha una durata di fluorescenza distinta, il tempo per il quale il fluoroforo rimane nello stato eccitato. L’imaging a vita di fluorescenza (FLIM) consente di quantificare l’intensità di fluorescenza e la durata di NAD(P)H e FAD per l’analisi senza etichetta del metabolismo cellulare. I microscopi a intensità di fluorescenza e durata possono essere ottimizzati per l’imaging di NAD(P)H e FAD selezionando le lunghezze d’onda di eccitazione ed emissione appropriate. Le perturbazioni metaboliche da parte del cianuro verificano i protocolli di imaging ad autofluorescenza per rilevare i cambiamenti metabolici all’interno delle cellule. Questo articolo dimostrerà la tecnica di imaging ad autofluorescenza di NAD(P)H e FAD per misurare il metabolismo cellulare.
Introduction
Il metabolismo è il processo cellulare di produzione di energia. Il metabolismo cellulare comprende più vie, tra cui glicolisi, fosforilazione ossidativa e glutaminolisi. Le cellule sane utilizzano queste vie metaboliche per generare energia per la proliferazione e la funzione, come la produzione di citochine da parte delle cellule immunitarie. Molte malattie, tra cui i disturbi metabolici, il cancro e la neurodegenerazione, sono caratterizzate da un metabolismo cellulare alterato1. Ad esempio, alcuni tipi di cellule tumorali hanno tassi elevati di glicolisi, anche in presenza di ossigeno, per generare molecole per la sintesi di acidi nucleici, proteine e lipidi2,3. Questo fenomeno, noto come effetto Warburg, è un segno distintivo di molti tipi di cancro, tra cui il cancro al seno, il cancro ai polmoni e i glioblastomi4. A causa delle alterazioni del metabolismo cellulare associate alla progressione del cancro, il metabolismo cellulare può essere un biomarcatore surrogato per la risposta ai farmaci5,6. Inoltre, comprendere l’efficacia del farmaco a livello cellulare è fondamentale in quanto l’eterogeneità cellulare può portare a diverse risposte farmacologiche negli individui7,8.
Le tecnologie che identificano e quantificano i cambiamenti nel metabolismo cellulare sono essenziali per gli studi sul cancro e sulla risposta ai farmaci. Le analisi chimiche e proteiche vengono utilizzate per valutare il metabolismo delle cellule o dei tessuti, ma mancano di risoluzione unicellulare e di informazioni spaziali. I saggi basati sul lettore di piastre metaboliche possono misurare il pH e il consumo di ossigeno nel campione nel tempo e la successiva perturbazione metabolica da parte di sostanze chimiche. Il pH può essere utilizzato per calcolare il tasso di acidificazione extracellulare (ECAR), che fornisce una panoramica dell’attività glicolitica delle cellule9. I metodi di imaging di tutto il corpo, tra cui la tomografia a emissione di positroni fluoro-D-glucosio 2-[fluoro-18] (FDG PET) e la spettroscopia di risonanza magnetica (MRS), sono modalità di imaging non invasive utilizzate clinicamente per identificare la recidiva del tumore e l’efficacia del farmaco attraverso misurazioni metaboliche10,11,12,13,14.
FDG-PET visualizza l’assorbimento tissutale di FDG, un analogo del glucosio radiomarcato. L’aumento dell’assorbimento di FDG-PET da parte dei tumori rispetto al tessuto circostante è dovuto all’effetto Warburg12,13. MRS visualizza nuclei comuni di molecole utilizzate per il metabolismo, come 13C e 31P, e può ottenere informazioni dinamiche su come il metabolismo cambia in risposta a stimoli, come l’esercizio fisico o il mangiare14. Sebbene FDG-PET e MRS possano essere utilizzati clinicamente, queste tecnologie mancano della risoluzione spaziale per risolvere l’eterogeneità intratumorale. Allo stesso modo, le misurazioni del consumo di ossigeno vengono effettuate su una popolazione di massa di cellule. L’imaging a autofluorescenza supera l’ostacolo della risoluzione spaziale di queste tecnologie e fornisce un metodo non invasivo per quantificare il metabolismo cellulare.
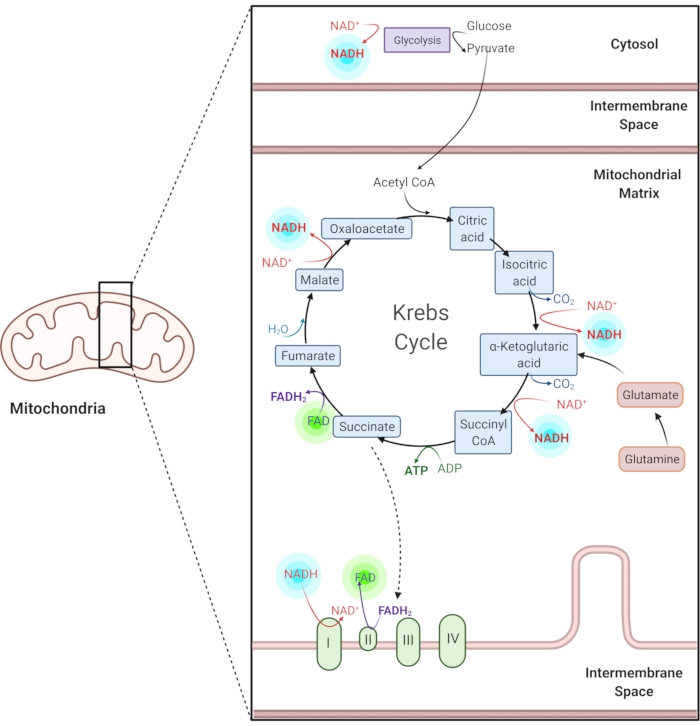
Figura 1: NADH e FAD nelle vie metaboliche comuni. NADH e FAD sono coenzimi utilizzati nella glicolisi, nel ciclo di Krebs e nella catena di trasporto degli elettroni. L’imaging a autofluorescenza di queste molecole fornisce informazioni sul metabolismo cellulare. Fare clic qui per visualizzare una versione più grande di questa figura.
La nicotinamide adenina (fosfato) dinucleotide (NAD(P)H) e la flavina adenina dinucleotide ossidata (FAD) sono coenzimi delle reazioni metaboliche, tra cui glicolisi, fosforilazione ossidativa e glutaminolisi (Figura 1). Sia NAD(P)H che FAD sono autofluorescenti e forniscono un contrasto endogeno per l’imaging a fluorescenza1,15. NADPH ha proprietà fluorescenti simili a NADH. Per questo motivo, NAD(P)H è spesso usato per rappresentare il segnale combinato di NADH e NADPH2,16.
L’imaging a vita di fluorescenza (FLIM) quantifica la durata della fluorescenza o il tempo per il quale un fluoroforo è nello stato eccitato. Le durate di fluorescenza rispondono al microambiente dei fluorofori e forniscono informazioni sul metabolismo cellulare17. NAD(P)H e FAD possono esistere all’interno delle cellule in conformazioni legate a proteine o libere, ognuna delle quali ha una vita diversa. Il NAD(P)H libero ha una vita più breve rispetto al NAD(P)H legato alle proteine; al contrario, la FAD libera ha una durata maggiore rispetto alla FAD18,19 legata. Le durate e i pesi dei componenti a vita possono essere quantificati dai dati di decadimento della durata della fluorescenza tramite Eq. (1)20:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq (1) rappresenta l’intensità di fluorescenza normalizzata in funzione del tempo. Le α 1 e α 2 in questa equazione rappresentano le componenti proporzionali di vita breve e lunga (α 1+ α 2=1), rispettivamente, τ1 e τ2 rappresentano rispettivamente la vita breve e lunga, e C rappresenta la luce di fondo7,20. La durata ponderata in ampiezza, qui rappresentata come τm, è calcolata utilizzando Eq. (2).
τm= α 1τ1+ α 2τ2 (2)
Una vita media può essere calcolata facendo la media di “t” sul decadimento di intensità del fluoroforo, che per un decadimento bieculente è mostrato da Eq. (3)17,21.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
Un’immagine di intensità di fluorescenza può essere calcolata dall’immagine a vita integrando il decadimento della durata della fluorescenza. L’imaging a autofluorescenza è un metodo non distruttivo e privo di etichette che può essere utilizzato per caratterizzare il metabolismo delle cellule vive a una risoluzione subcellulare. Il rapporto redox ottico fornisce una metrica analogica ottica dello stato redox chimico della cellula ed è calcolato come il rapporto tra le intensità NAD(P)H e FAD. Sebbene la formula per il calcolo del rapporto redox ottico non sia standardizzata22,23,24,25, è qui definita come l’intensità di FAD sulle intensità combinate di NAD(P)H e FAD. Questa definizione viene utilizzata perché l’intensità sommata nel denominatore normalizza la metrica tra 0 e 1 e il risultato atteso dell’inibizione del cianuro è una diminuzione del rapporto redox. Le durate di fluorescenza di NAD(P)H e FAD liberi forniscono informazioni sui cambiamenti nel microambiente del solvente metabolico, tra cui pH, temperatura, vicinanza all’ossigeno e osmolarità17.
I cambiamenti nella durata di fluorescenza delle frazioni legate di NAD(P)H e FAD possono indicare l’utilizzo della via metabolica e il metabolismo specifico del substrato26. I pesi dei componenti possono essere interpretati per cambiamenti nella frazione libera al legato dei coenzimi18,19. Complessivamente, queste metriche quantitative di durata dell’autofluorescenza consentono l’analisi del metabolismo cellulare e l’imaging ad autofluorescenza è stato utilizzato per identificare neoplasie da tessuti normali27,28, caratterizzare le cellule staminali29,30, valutare la funzione delle cellule immunitarie31,32,33,34,35, misurare l’attività neurologica36, 37,38 e comprensione dell’efficacia dei farmaci in tipi di cancro come il cancro al seno e il cancro della testa e del collo21,39,40,41,42. L’imaging ad autofluorescenza ad alta risoluzione può essere combinato con la segmentazione dell’immagine per l’analisi di singole cellule e la quantificazione dell’eterogeneità intrapopolazione43,44,45,46,47.
NAD(P)H e FAD possono essere ripresi su microscopi a fluorescenza a fotone singolo o multifotone configurati per l’imaging di intensità o durata. Per i microscopi a singolo fotone, NAD(P)H e FAD sono tipicamente eccitati a lunghezze d’onda di 375-405 nm e 488 nm, rispettivamente, a causa di sorgenti laser comuni a queste lunghezze d’onda48. Nell’eccitazione a fluorescenza a due fotoni, NAD(P)H e FAD ecciteranno a lunghezze d’onda di circa 700-750 nm e da 700 a 900 nm, rispettivamente15,49. Una volta che i fluorofori sono eccitati, NAD(P)H e FAD emettono fotoni a lunghezze d’onda comprese tra ~ 410 nm a ~ 490 nm e ~ 510 nm a ~ 640 nm, rispettivamente15. Le lunghezze d’onda massime di emissione NAD(P)H e FAD sono rispettivamente di circa 450 nm e 535 nm48.
A causa delle loro diverse lunghezze d’onda di eccitazione ed emissione, la fluorescenza dei due coenzimi metabolici può essere isolata spettralmente. Una comprensione delle caratteristiche spettrali di NAD(P)H e FAD è necessaria per la progettazione e l’ottimizzazione dei protocolli di imaging ad autofluorescenza. Il cianuro è un inibitore iv del complesso della catena di trasporto degli elettroni (ETC). Gli effetti del cianuro sul metabolismo cellulare e le intensità di autofluorescenza e la durata di vita di NAD(P)H e FAD all’interno delle cellule sono ben caratterizzati27,40. Pertanto, un esperimento di perturbazione al cianuro è un mezzo efficace per convalidare i protocolli di imaging NAD(P)H e FAD. Un esperimento di cianuro di successo fornisce la certezza che il protocollo di imaging NAD(P)H e FAD può essere utilizzato per valutare il metabolismo di gruppi sconosciuti o perturbazioni.
Protocol
Representative Results
Discussion
L’intensità di autofluorescenza e l’imaging a vita sono stati ampiamente utilizzati per valutare il metabolismo nelle cellule21,55. FLIM è ad alta risoluzione e quindi risolve singole cellule, che è importante per gli studi sul cancro perché l’eterogeneità cellulare contribuisce all’aggressività tumorale e alla resistenza ai farmaci7,39,41,44,45,46,58.<s…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Le fonti di finanziamento includono il Cancer Prevention and Research Institute of Texas (CPRIT RP200668) e la Texas A & M University. La Figura 1 è stata creata con BioRender.com.
Materials
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25×36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25×36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
References
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532 (2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500 (2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271 (2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Research. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456 (2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068 (2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer’s disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008 (2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104 (2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. . Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792 (2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007)
- . CellProfiler Available from: https://cellprofiler.org/releases (2007)
- Autofluorescence Imaging. GitHub Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021)
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. Cancer Research. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850 (2018).
- . The bh TCSPC Handbook Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021)
- Gadella, T. W. J., Mason, W. T. . Fluorescent and Luminescent Probes for Biological Activity. 34, 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).

