Автофлуоресцентная визуализация для оценки клеточного метаболизма
Summary
Этот протокол описывает флуоресцентную визуализацию и анализ эндогенных метаболических коферментов, восстановленного динуклеотида никотинамидаденина (фосфата) (NAD(P)H) и окисленного флавинадениндинуклеотида (FAD). Автофлуоресцентная визуализация NAD(P)H и FAD обеспечивает безмечаночный, неразрушающий метод оценки клеточного метаболизма.
Abstract
Клеточный метаболизм — это процесс, посредством которого клетки вырабатывают энергию, и многие заболевания, включая рак, характеризуются аномальным метаболизмом. Восстановленный никотинамид аденин (фосфат) динуклеотид (NAD(P)H) и окисленный флавинадениндинуклеотид (FAD) являются коферментами метаболических реакций. NAD(P)H и FAD проявляют автофлуоресценцию и могут быть спектрально изолированы волнами возбуждения и излучения. Оба кофермента, NAD(P)H и FAD, могут существовать либо в свободной, либо в связанной с белком конфигурации, каждая из которых имеет различное время жизни флуоресценции – время, в течение которого флуорофор остается в возбужденном состоянии. Флуоресцентная визуализация времени жизни (FLIM) позволяет количественно оценить интенсивность флуоресценции и время жизни NAD(P)H и FAD для анализа клеточного метаболизма без меток. Микроскопы с интенсивностью флуоресценции и сроком службы могут быть оптимизированы для визуализации NAD(P)H и FAD путем выбора соответствующих длин волн возбуждения и излучения. Метаболические возмущения цианидом проверяют протоколы автофлуоресцентной визуализации для обнаружения метаболических изменений в клетках. В данной статье будет продемонстрирована методика автофлуоресцентной визуализации NAD(P)H и FAD для измерения клеточного метаболизма.
Introduction
Метаболизм – это клеточный процесс производства энергии. Клеточный метаболизм охватывает несколько путей, включая гликолиз, окислительное фосфорилирование и глутаминолиз. Здоровые клетки используют эти метаболические пути для выработки энергии для пролиферации и функционирования, такой как производство цитокинов иммунными клетками. Многие заболевания, включая нарушения обмена веществ, рак и нейродегенерацию, характеризуются измененным клеточным метаболизмом1. Например, некоторые типы раковых клеток имеют повышенные скорости гликолиза, даже в присутствии кислорода, для генерации молекул для синтеза нуклеиновых кислот, белков и липидов2,3. Это явление, известное как эффект Варбурга, является отличительной чертой многих типов рака, включая рак молочной железы, рак легких и глиобластомы4. Из-за изменений клеточного метаболизма, связанных с прогрессированием рака, клеточный метаболизм может быть суррогатным биомаркером лекарственного ответа5,6. Кроме того, понимание эффективности лекарств на клеточном уровне имеет решающее значение, поскольку гетерогенность клеток может привести к различным реакциям на лекарства у людей7,8.
Технологии, которые идентифицируют и количественно оценивают изменения в клеточном метаболизме, необходимы для исследований рака и лекарственного ответа. Химические и белковые анализы используются для оценки метаболизма клеток или тканей, но им не хватает одноклеточного разрешения и пространственной информации. Анализы на основе считывателя метаболических пластин могут измерять рН и потребление кислорода в образце с течением времени и последующее метаболическое возмущение химическими веществами. pH может быть использован для расчета скорости внеклеточного подкисления (ECAR), что дает представление о гликолитической активности клеток9. Методы визуализации всего тела, включая 2-[фтор-18] фтор-D-глюкозо-позитронно-эмиссионную томографию (FDG PET) и магнитно-резонансную спектроскопию (MRS), являются неинвазивными методами визуализации, используемыми клинически для выявления рецидива опухоли и эффективности лекарств с помощью метаболических измерений10,11,12,13,14.
FDG-PET изображает поглощение тканями FDG, радиомаркированного аналога глюкозы. Повышенное поглощение ФДГ-ПЭТ опухолями относительно окружающих тканей обусловлено эффектом Варбурга12,13. MRS изображает общие ядра молекул, используемых для метаболизма, таких как 13C и 31P, и может получать динамическую информацию о том, как метаболизм изменяется в ответ на стимулы, такие как физические упражнения или еда14. Хотя ФДГ-ПЭТ и МРС могут использоваться клинически, этим технологиям не хватает пространственного разрешения для разрешения внутриопухолевой гетерогенности. Аналогичным образом, измерения потребления кислорода производятся на массовой популяции клеток. Автофлуоресцентная визуализация преодолевает препятствие пространственного разрешения этих технологий и обеспечивает неинвазивный метод количественной оценки клеточного метаболизма.
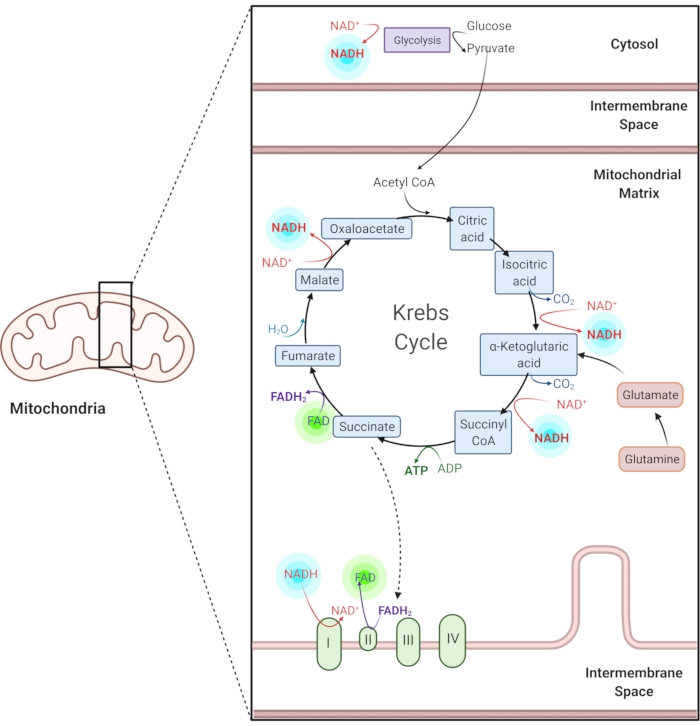
Рисунок 1: NADH и FAD в общих метаболических путях. NADH и FAD являются коферментами, используемыми в гликолизе, цикле Кребса и цепи переноса электронов. Автофлуоресцентная визуализация этих молекул предоставляет информацию о клеточном метаболизме. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Восстановленный никотинамид аденин (фосфат) динуклеотид (NAD(P)H) и окисленный флавинадениндинуклеотид (FAD) являются коферментами метаболических реакций, включая гликолиз, окислительное фосфорилирование и глутаминолиз (рисунок 1). Как NAD(P)H, так и FAD являются автофлуоресцентными и обеспечивают эндогенный контраст для флуоресцентной визуализации1,15. NADPH обладает свойствами, аналогичными свойствам NADH. Из-за этого NAD(P)H часто используется для представления комбинированного сигнала NADH и NADPH2,16.
Флуоресцентная визуализация времени жизни (FLIM) количественно определяет время жизни флуоресценции или время, в течение которого флуорофор находится в возбужденном состоянии. Время жизни флуоресценции реагирует на микроокружение флуорофоров и предоставляет информацию о клеточном метаболизме17. NAD(P)H и FAD могут существовать в клетках в связанных с белком или свободных конформациях, каждая из которых имеет различное время жизни. Свободный NAD(P)H имеет более короткий срок службы, чем связанный с белком NAD(P)H; и наоборот, свободный FAD имеет более длительный срок службы, чем связанный FAD18,19. Время жизни и вес компонентов в течение срока службы могут быть количественно определены на основе данных о распаде срока службы флуоресценции с помощью экв. (1)20:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq (1) представляет собой нормализованную интенсивность флуоресценции как функцию времени. Α 1 и α 2 в этом уравнении представляют пропорциональные компоненты короткого и длительного времени жизни (α 1+ α 2 = 1), соответственно, τ1 и τ2 представляют собой короткое и долгое время жизни соответственно, а C учитывает фоновое освещение7,20. Амплитудно-взвешенное время жизни, представленное здесь как τm, вычисляется с использованием Eq. (2).
τm= α 1τ1+ α 2τ2 (2)
Среднее время жизни может быть вычислено путем усреднения «t» по интенсивности распада флуорофора, что для двухэкспоненциального распада показано экв. (3)17,21.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
Изображение интенсивности флуоресценции может быть вычислено на основе изображения времени жизни путем интеграции распада времени жизни флуоресценции. Автофлуоресцентная визуализация является неразрушающим и безметочным методом, который может быть использован для характеристики метаболизма живых клеток с субклеточным разрешением. Оптический окислительно-восстановительный коэффициент обеспечивает оптическую аналоговую метрику химического окислительно-восстановительного состояния клетки и рассчитывается как отношение интенсивностей NAD(P)H и FAD. Хотя формула для расчета оптического окислительно-восстановительного соотношения не стандартизирована22,23,24,25, она определяется здесь как интенсивность FAD по комбинированным интенсивностям NAD(P)H и FAD. Это определение используется потому, что суммированная интенсивность в знаменателе нормализует метрику между 0 и 1, а ожидаемым результатом ингибирования цианида является снижение окислительно-восстановительного соотношения. Время жизни флуоресценции свободных NAD(P)H и FAD дает представление об изменениях в микроокружении метаболического растворителя, включая рН, температуру, близость к кислороду и осмолярность17.
Изменения в времени жизни флуоресценции связанных фракций NAD(P)H и FAD могут указывать на использование метаболического пути и субстрат-специфический метаболизм26. Массы компонентов могут быть интерпретированы для изменения свободной до связанной фракции коэнзимов18,19. В целом, эти количественные показатели времени аутофлуоресценции позволяют анализировать клеточный метаболизм, а аутофлуоресцентная визуализация была использована для идентификации новообразований из нормальных тканей27,28, характеризующих стволовые клетки29,30, оценивающих функцию иммунных клеток31,32,33,34,35, измеряющих неврологическую активность36, 37,38, и понимание эффективности лекарств при таких типах рака, как рак молочной железы и рак головы и шеи21,39,40,41,42. Автофлуоресцентная визуализация высокого разрешения может быть объединена с сегментацией изображения для одноклеточного анализа и количественной оценки гетерогенности интрапопуляции43,44,45,46,47.
NAD(P)H и FAD могут быть визуализированы на однофотонных или многофотонных флуоресцентных микроскопах, сконфигурированных для интенсивной или пожизненной визуализации. Для однофотонных микроскопов NAD(P)H и FAD обычно возбуждаются на длинах волн 375-405 нм и 488 нм соответственно из-за общих лазерных источников на этих длинах волн48. При двухфотонном флуоресцентном возбуждении NAD(P)H и FAD будут возбуждаться на длинах волн приблизительно от 700 до 750 нм и от 700 до 900 нм соответственно15,49. Как только флуорофоры возбуждаются, NAD(P)H и FAD испускают фотоны на длинах волн от ~410 нм до ~490 нм и ~510 нм до ~640 нм, соответственно15. Длины волн максимального излучения NAD(P)H и FAD составляют приблизительно 450 нм и 535 нм соответственно48.
Из-за их различных длин волн возбуждения и излучения флуоресценция двух метаболических коферментов может быть спектрально изолирована. Понимание спектральных характеристик NAD(P)H и FAD необходимо для проектирования и оптимизации протоколов автофлуоресцентной визуализации. Цианид является ингибитором комплекса IV цепи переноса электронов (ETC). Влияние цианида на клеточный метаболизм и интенсивность аутофлуоресценции и время жизни NAD(P)H и FAD в клетках хорошо охарактеризованы27,40. Таким образом, эксперимент с возмущением цианида является эффективным средством проверки протоколов визуализации NAD(P)H и FAD. Успешный эксперимент с цианидом дает уверенность в том, что протокол визуализации NAD(P)H и FAD может быть использован для оценки метаболизма неизвестных групп или возмущений.
Protocol
Representative Results
Discussion
Интенсивность автофлуоресценции и пожизненная визуализация широко использовались для оценки метаболизма в клетках21,55. FLIM имеет высокое разрешение и, следовательно, разрешает одиночные клетки, что важно для исследований рака, поскольку клеточная гетер?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Источники финансирования включают Институт профилактики рака и исследований Техаса (CPRIT RP200668) и Техасский университет A & M. Рисунок 1 был создан с помощью BioRender.com.
Materials
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25×36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25×36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
References
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532 (2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500 (2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271 (2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Research. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456 (2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068 (2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer’s disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008 (2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104 (2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. . Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792 (2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007)
- . CellProfiler Available from: https://cellprofiler.org/releases (2007)
- Autofluorescence Imaging. GitHub Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021)
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. Cancer Research. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850 (2018).
- . The bh TCSPC Handbook Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021)
- Gadella, T. W. J., Mason, W. T. . Fluorescent and Luminescent Probes for Biological Activity. 34, 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).

