Eine Spin-Tip-Anreicherungsstrategie zur simultanen Analyse von N-Glykopeptiden und Phosphopeptiden aus menschlichem Bauchspeicheldrüsengewebe
Summary
Posttranslationale Modifikationen (PTMs) verändern Proteinstrukturen und -funktionen. Methoden zur gleichzeitigen Anreicherung mehrerer PTM-Typen können die Abdeckung in Analysen maximieren. Wir präsentieren ein Protokoll mit dual-funktioneller Ti(IV)-immobilisierter Metallaffinitätschromatographie, gefolgt von Massenspektrometrie zur gleichzeitigen Anreicherung und Analyse der Protein-N-Glykosylierung und Phosphorylierung in Pankreasgeweben.
Abstract
Die Massenspektrometrie kann eine tiefe Abdeckung der posttranslationalen Modifikationen (PTMs) liefern, obwohl eine Anreicherung dieser Modifikationen aus komplexen biologischen Matrizen aufgrund ihrer geringen Stöchiometrie im Vergleich zu nicht modifizierten Analyten oft erforderlich ist. Die meisten Anreicherungs-Workflows von PTMs auf Peptiden in Bottom-up-Proteomik-Workflows, bei denen Proteine enzymatisch verdaut werden, bevor die resultierenden Peptide analysiert werden, reichern nur eine Art von Modifikation an. Es ist jedoch das gesamte Komplement von PTMs, das zu biologischen Funktionen führt, und die Anreicherung einer einzigen Art von PTM kann ein solches Übersprechen von PTMs übersehen. PTM-Übersprechen wurde zwischen Proteinglykosylierung und Phosphorylierung beobachtet, den beiden häufigsten PTMs in menschlichen Proteinen und auch den beiden am meisten untersuchten PTMs unter Verwendung von Massenspektrometrie-Workflows. Unter Verwendung der hier beschriebenen simultanen Anreicherungsstrategie werden beide PTMs aus postmortalem menschlichem Pankreasgewebe, einer komplexen biologischen Matrix, angereichert. Die dual-funktionelle Ti(IV)-immobilisierte Metallaffinitätschromatographie wird verwendet, um verschiedene Formen der Glykosylierung und Phosphorylierung gleichzeitig in mehreren Fraktionen in einer bequemen Spinspitzen-basierten Methode zu trennen, die nachgelagerte Analysen potenzieller PTM-Crosstalk-Wechselwirkungen ermöglicht. Dieser Anreicherungs-Workflow für Glyko- und Phosphopeptide kann auf verschiedene Probentypen angewendet werden, um eine tiefe Profilierung mehrerer PTMs zu erreichen und potenzielle Zielmoleküle für zukünftige Studien zu identifizieren.
Introduction
Protein-posttranslationale Modifikationen (PTMs) spielen eine wichtige Rolle bei der Modulation von Proteinstrukturen und damit ihrer Funktionen und nachgelagerten biologischen Prozesse. Die Vielfalt des menschlichen Proteoms nimmt aufgrund der kombinatorischen Variabilität verschiedener PTMs exponentiell zu. Verschiedene Varianten von Proteinen aus ihren kanonischen Sequenzen, wie sie vom Genom vorhergesagt werden, werden als Proteoformen bezeichnet, und viele Proteoformen entstehen ausPTMs 1. Die Untersuchung der proteoformen Vielfalt in Gesundheit und Krankheit ist in den letzten Jahren zu einem Forschungsgebiet von großem Interessegeworden 2,3.
Das Studium von Proteoformen und insbesondere von PTMs mit großer Tiefe ist durch die Entwicklung von auf Massenspektrometrie (MS) basierenden Proteomikmethoden einfacher geworden. Mit MS werden Analyten ionisiert, fragmentiert und basierend auf dem m/z von Fragmenten identifiziert. Anreicherungsmethoden sind aufgrund der geringen relativen Häufigkeit von PTMs im Vergleich zu nicht modifizierten Formen von Proteinen oft notwendig. Obwohl die Analyse intakter Proteine und ihrer PTMs, die sogenannten Top-Down-Analysen, routinemäßiger geworden sind, ist die enzymatische Verdauung von Proteinen und die Analyse ihrer Komponentenpeptide in Bottom-up-Analysen immer noch der am weitesten verbreitete Weg für die PTM-Analyse. Die beiden am häufigsten untersuchten PTMs und die beiden häufigsten PTMs in vivo sind Glykosylierung und Phosphorylierung4. Diese beiden PTMs spielen eine wichtige Rolle bei der Zellsignalisierung und -erkennung und sind daher wichtige Modifikationen, die in der Krankheitsforschung charakterisiert werden müssen.
Die chemischen Eigenschaften verschiedener PTMs bieten oft Wege zur Anreicherung dieser PTMs auf Protein- und Peptidebene vor der Analyse. Die Glykosylierung ist ein hydrophiles PTM aufgrund der Häufigkeit von Hydroxylgruppen auf jedem Monosaccharid. Diese Eigenschaft kann verwendet werden, um Glykopeptide in der hydrophilen Interaktionschromatographie (HILIC) anzureichern, die mehr hydrophile Glykopeptide von den hydrophoben, nicht modifizierten Peptidentrennen können 5. Die Phosphorylierung fügt den Phosphatanteil hinzu, der außer bei saurem pH-Wert negativ geladen ist. Aufgrund dieser Ladung können verschiedene Metallkationen, einschließlich Titan, verwendet werden, um Phosphopeptide anzuziehen und zu binden, während nicht phosphorylierte Spezies weggespült werden. Dies ist das Prinzip der immobilisierten Metallaffinitätschromatographie (IMAC). Weitere Erörterungen dieser und anderer Anreicherungsstrategien für Glykosylierung und Phosphorylierung finden sich in den letzten Reviews 6,7.
Vergleichsweise große Mengen an Ausgangspeptidmaterial (0,5 mg oder mehr) werden aufgrund der geringen Stöchiometrie von PTMs an Peptiden häufig für Anreicherungsprotokolle benötigt. In Szenarien, in denen diese Probenmenge möglicherweise nicht leicht gewonnen werden kann, wie z. B. Tumorkernbiopsie oder Liquoranalysen, ist es vorteilhaft, einfache Arbeitsabläufe zu verwenden, die zu maximalen biomolekularen Informationen führen. Jüngste Strategien, die von unserem Labor und anderen entwickelt wurden, haben die gleichzeitige und parallele Analyse von Glykosylierung und Phosphorylierung unter Verwendung desselben PTM-Anreicherungsworkflows 8,9,10,11,12 hervorgehoben. Obwohl sich die chemischen Eigenschaften dieser beiden PTMs unterscheiden können, können diese PTMs aufgrund der innovativen Trenntechniken und verwendeten Materialien in mehreren Schritten analysiert werden. Zum Beispiel überlagert die elektrostatische Abstoßungs-hydrophile Wechselwirkungschromatographie (ERLIC) Trennungen basierend auf hydrophilen Wechselwirkungen zwischen Analyten und der mobilen Phase mit Ladungs-Ladungs-Wechselwirkungen zwischen Analyten und dem stationären Phasenmaterial13,14,15,16. Bei saurem pH-Wert kann die Anziehung phosphorylierter Peptide in die stationäre Phase ihre Retention und Trennung von nicht modifizierten Peptiden verbessern. Material, das aus Ti(IV) besteht, das auf hydrophilen Mikrosphären immobilisiert ist, kann für HILIC- und IMAC-basierte Elution verwendet werden, um Phosphopeptide und neutrale, saure und Mannose-6-phosphorylierte Glykopeptide17,18 zu trennen. Diese Strategie wird als dual-funktionales Ti(IV)-IMAC bezeichnet. Die Verwendung dieser Strategien zur Anreicherung mehrerer PTMs in einem einzigen Workflow kann Analysen potenzieller PTM-Crosstalk-Interaktionen zugänglicher machen. Darüber hinaus sind die Gesamtprobenmenge und der Zeitaufwand geringer als bei den herkömmlichen Anreicherungsmethoden, wenn sie parallel durchgeführt werden (d. h. HILIC und IMAC auf separaten Probenaliquoten).
Um die dual-funktionelle Ti(IV)-IMAC-Strategie für die gleichzeitige Analyse von Proteinglykosylierung und Phosphorylierung zu demonstrieren, haben wir sie zur Analyse von postmortalem menschlichem Pankreasgewebe angewendet. Die Bauchspeicheldrüse produziert sowohl Verdauungsenzyme als auch regulatorische Hormone, einschließlich Insulin und Glucagon. Die Pankreasfunktion ist bei Bauchspeicheldrüsenerkrankungen beeinträchtigt. Bei Diabetes ist die Regulierung des Blutzuckers betroffen, was zu höheren Glukosespiegeln im Blut führt. Bei Pankreatitis resultiert eine Entzündung aus der Autoverdauung desOrgans 3. Veränderungen der PTM-Profile, einschließlich Glykosylierung und Phosphorylierung, können, wie es oft der Fall ist, bei anderen Krankheiten auftreten.
Hier beschreiben wir ein Protokoll für eine Spin-Tip-basierte simultane Anreicherungsmethode, basierend auf einer dual-funktionellen Ti(IV)-IMAC-Strategie, für N-Glykopeptide und Phosphopeptide, die aus Proteinen gewonnen werden, die aus Pankreasgewebe extrahiert werden. Das Protokoll umfasst Proteinextraktion und -verdauung, Anreicherung, MS-Datenerfassung und Datenverarbeitung, wie in Abbildung 1 dargestellt. Repräsentative Daten aus dieser Studie sind über das ProteomeXchange-Konsortium mit der Kennung PXD033065 verfügbar.
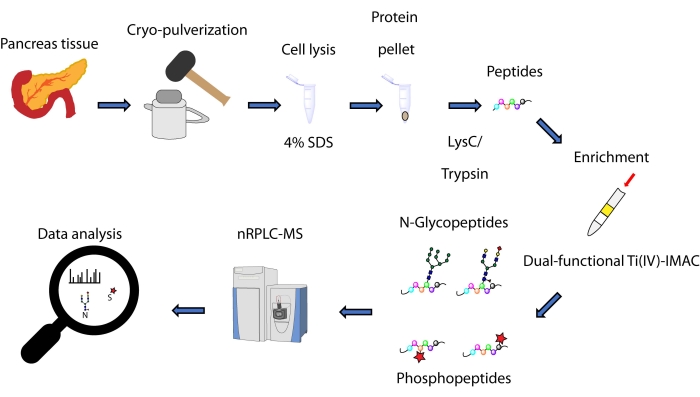
Abbildung 1: Workflow zur simultanen Analyse von N-Glykopeptiden und Phosphopeptiden aus humanem Pankreasgewebe. Gewebe werden zunächst vor der Proteinextraktion mit dem Waschmittel Natriumdodecylsulfat (SDS) zu einem feinen Pulver pulverisiert. Proteine werden dann einer enzymatischen Verdauung unterzogen. Die resultierenden Peptide werden vor der Anreicherung mit dual-funktionellem Ti(IV)-IMAC aliquotiert. Die Rohdaten werden mittels nanoskaliger Umkehrphasen-Flüssigkeitschromatographie-Massenspektrometrie (nRPLC-MS) gesammelt und mit Hilfe einer Datenbanksuchsoftware analysiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Dieses Protokoll soll PTM-Analysen zugänglicher machen und eine umfassendere Analyse mehrerer PTMs im selben Workflow ermöglichen. Dieses Protokoll kann auf andere komplexe biologische Matrizen, einschließlich Zellen und Bioflüssigkeiten, angewendet werden.
Protocol
Representative Results
Discussion
Die dual-funktionelle Ti(IV)-IMAC-Strategie ist nützlich für die gleichzeitige Analyse von N-Glykopeptiden und Phosphopeptiden aus derselben Probe in einem einzigen Probenvorbereitungs-Workflow. Es wurde auch gezeigt, dass ERLIC-basierte Methoden eine gleichzeitige Anreicherung von PTMs durchführen. Beide Strategien wurden zuvor für Deep Coverage in PTM-Analyseneingesetzt 14,18. Durch die Anpassung der dualen Ti-Methode an die Verkürzung der Probeninkubation…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Diese Forschung wurde zum Teil durch Zuschüsse des NIH (R01DK071801, RF1AG052324, P01CA250972 und R21AG065728) und der Juvenile Diabetes Research Foundation (1-PNF-2016-250-S-B und SRA-2016-168-S-B) unterstützt. Die hier vorgestellten Daten wurden zum Teil auch durch die Unterstützung eines NIH/NCATS UL1TR002373 Award durch das Institute for Clinical and Translational Research der University of Wisconsin gewonnen. Die Orbitrap-Instrumente wurden durch die Unterstützung eines NIH Shared Instrument Grant (NIH-NCRR S10RR029531) und des Office of the Vice Chancellor for Research and Graduate Education an der University of Wisconsin-Madison erworben. Wir möchten auch die großzügige Unterstützung der Organ- und Gewebespendeorganisation der University of Wisconsin anerkennen, die die menschliche Bauchspeicheldrüse für die Forschung zur Verfügung gestellt hat, und die Hilfe von Dan Tremmel, Dr. Sara D. Sackett und Prof. Jon Odorico für die Bereitstellung der Proben für unser Labor. Unser Forschungsteam möchte sich besonders bei den Familien bedanken, die für diese Studie Taschentücher gespendet haben. L.L. würdigt den NIH-Zuschuss S10OD025084, einen Pankreaskrebs-Pilotzuschuss des Carbone Cancer Center der University of Wisconsin (233-AAI9632) sowie eine Vilas Distinguished Achievement Professorship und die Charles Melbourne Johnson Distinguished Chair Professorship mit Mitteln der Wisconsin Alumni Research Foundation und der University of Wisconsin-Madison School of Pharmacy.
Materials
| Acetic Acid, Glacial (Certified ACS) | Fisher Scientific | A38S-500 | |
| Acetone (Certified ACS) | Fisher Scientific | A18-1 | |
| Acetonitrile, Optima LC/MS Grade | Fisher Scientific | A955-4 | |
| Ammonium Acetate (Crystalline/Certified ACS) | Fisher Scientific | A637-500 | |
| Ammonium Hydroxide (Certified ACS Plus) | Fisher Scientific | A669-212 | |
| Byonic software | Protein Metrics | n/a | Commercial software used for glycoproteomic analysis (https://proteinmetrics.com/byos/) |
| C18 BEH material | Waters | 186002353 | Material removed from column and used to pack nano capillaries (pulledto integrate tip used directly in line with instrument inlet) |
| CAE-Ti-IMAC, 100% | J&K Scientific | 2749380-1G | Material used for dual-functional Ti(IV)-IMAC; can also be used for conventional IMAC/conventional phosphopeptide enrichment |
| Cellcrusher kit | Cellcrusher | n/a | Used for grinding tissue samples into powder before extraction |
| Eppendorf 5424R Microcentrifuge | Fisher Scientific | 05-401-205 | For temperature-controlled centrifugation |
| cOmplete protease inhibitor cocktail tablets | Sigma | 11697498001 | |
| DTT, Molecular Grade (DL-Dithiothreitol) | Promega | V3151 | Protein reducing agent |
| Ethanol, 200 proof (100%), USP | Fisher | 22-032-601 | |
| Fisherbrand Analog Vortex Mixer | Fisher Scientific | 02-215-414 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (1.5 mL) | Fisher Scientific | 02-681-320 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (2 mL) | Fisher Scientific | 02-681-321 | |
| Fisherbrand Model 120 Sonic Dismembrator | Fisher Scientific | FB120110 | For sample lysis using ultrasonication |
| Formic Acid, 99.0+%, Optima LC/MS Grade | Fisher Scientific | A117-50 | |
| Fused silica capillary (75 μm inner diameter, 360 μm outer diameter) | Polymicro Technologies LLC | 100 m TSP075375 | For in-house pulled and packed columns with integrated emitter |
| Hydrofluoric acid (48 wt. % in H2O) | Sigma-Aldrich | 339261-100ML | Used for opening emitter of pulled capillary column |
| Iodoacetamide, BioUltra | Sigma | I1149-5G | Protein reducing reagent |
| MaxQuant software | n/a | n/a | Free software used for phosphoproteomic analysis (https://www.maxquant.org/) |
| Multi-therm Shaker with heating and cooling | Benchmark Scientific | H5000-HC | Heating block |
| Oasis HLB 1 cc Vac Cartridge, 10 mg Sorbent per Cartridge, 30 µm, 100/pk | Waters | 186000383 | Larger-scale cartridge desalting for tryptic digests (loading capacity approximately up to 1 mg each) |
| OMIX C18 pipette tips, 100 µL tip, 10 – 100 μL elution volume, 1 x 96 tips | Agilent | A57003100 | Smaller-scale packed pipette tip for desalting for enrichment elutions |
| P-2000 Micropipette Puller | Sutter Instrument Co. | P-2000/F | For pulling nano-capillary columns for LC-MS |
| PhosSTOP phosphatase inhibitor tablets | Sigma | 4906845001 | |
| Pierce BCA Protein Assay Kit | Thermo Fisher Scientific | 23225 | |
| Pierce Quantitative Colorimetric Peptide Assay | Thermo Fisher Scientific | 23275 | |
| PolySAX LP (12 μm, pore size 300 Å) | PolyLC | BMSX1203 | Material for strong anion-exchange chromatography used for ERLIC/conventional glycopeptide enrichment |
| Potassium Phosphate Monobasic (Crystalline/Certified ACS) | Fisher Scientific | P285-500 | |
| Pressure injection cell with integrated magnetic stirplate | Next Advance | PC77-MAG | For packing nano-capillary columns with stationary phase up to 2500 psi limit |
| Proteome Discoverer software | Thermo Fisher Scientific | n/a | Commercial software for proteomics anaysis (with integrated database searching software nodes) and data visualization (https://www.thermofisher.com/us/en/home/industrial/mass-spectrometry/liquid-chromatography-mass-spectrometry-lc-ms/lc-ms-software/multi-omics-data-analysis/proteome-discoverer-software.html) |
| SpeedVac SC110 Vacuum Concentrator Model SC110-120 | Savant | n/a | Centrifugal vacuum concentrator for drying samples (under heat) |
| SDS Solution, 10% Sodium Dodecyl Sulfate Solution, Molecular Biology/Electrophoresis | Fisher Scientific | BP2436200 | |
| Sequencing Grade Modified Trypsin | Promega | V5111 | |
| Sodium Chloride (Crystalline/Certified ACS) | Fisher Scientific | S271-500 | |
| TopTip, Empty, 10-200 µL, Pack of 96 | Glygen Corporation | TT2EMT.96 | Empty pipette tip with micron-sized hole used that can be used to pack chromatographic materials for enrichments, bundled with tube adapters |
| Triethylammonium bicarbonate buffer (TEAB, 1 M, pH 8.5 (volatile)) | Sigma | 90360-100ML | |
| Trifluoroacetic acid, Reagent Grade, 99% | Fisher Scientific | 60-017-61 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | Fisher Scientific | BP152-500 | |
| Trypsin/Lys-C Mix, Mass Spec Grade | Promega | V5071 | |
| Urea (Certified ACS) | Fisher Scientific | U15-500 | |
| Water, Optima LC/MS Grade | Fisher Scientific | W64 |
References
- Smith, L. M., Kelleher, N. L. Proteoform: a single term describing protein complexity. Nature Methods. 10 (3), 186-187 (2013).
- Pan, S., Brentnall, T. A., Chen, R. Glycoproteins and glycoproteomics in pancreatic cancer. World Journal of Gastroenterology. 22 (42), 9288-9299 (2016).
- Tabang, D. N., Ford, M., Li, L. Recent advances in mass spectrometry-based glycomic and glycoproteomic studies of pancreatic diseases. Frontiers in Chemistry. 9, 707387 (2021).
- Khoury, G. A., Baliban, R. C., Floudas, C. A. Proteome-wide post-translational modification statistics: frequency analysis and curation of the swiss-prot database. Scientific Reports. 1, 90 (2011).
- Alpert, A. J. Hydrophilic-interaction chromatography for the separation of peptides, nucleic acids and other polar compounds. Journal of Chromatography A. 499, 177-196 (1990).
- Riley, N. M., Bertozzi, C. R., Pitteri, S. J. A pragmatic guide to enrichment strategies for mass spectrometry-based glycoproteomics. Molecular & Cellular Proteomics. 20, 100029 (2020).
- Low, T. Y., et al. Widening the bottleneck of phosphoproteomics: Evolving strategies for phosphopeptide enrichment. Mass Spectrometry Reviews. 40 (4), 309-333 (2021).
- Cho, K. C., Chen, L., Hu, Y., Schnaubelt, M., Zhang, H. Developing workflow for simultaneous analyses of phosphopeptides and glycopeptides. ACS Chemical Biology. 14 (1), 58-66 (2019).
- Zhou, Y., et al. An integrated workflow for global, glyco-, and phospho-proteomic analysis of tumor tissues. Analytical Chemistry. 92 (2), 1842-1849 (2020).
- Tang, R., et al. Facile preparation of bifunctional adsorbents for efficiently enriching N-glycopeptides and phosphopeptides. Analytica Chimica Acta. 1144, 111-120 (2021).
- Wang, Z., Wang, J., Sun, N., Deng, C. A promising nanoprobe based on hydrophilic interaction liquid chromatography and immobilized metal affinity chromatography for capture of glycopeptides and phosphopeptides. Analytica Chimica Acta. 1067, 1-10 (2019).
- Glover, M. S., et al. Characterization of intact sialylated glycopeptides and phosphorylated glycopeptides from IMAC enriched samples by EThcD fragmentation: Toward combining phosphoproteomics and glycoproteomics. International Journal of Mass Spectrometry. 427, 35-42 (2018).
- Alpert, A. J. Electrostatic repulsion hydrophilic interaction chromatography for isocratic separation of charged solutes and selective isolation of phosphopeptides. Analytical Chemistry. 80 (1), 62-76 (2008).
- Cui, Y., et al. Counterion optimization dramatically improves selectivity for phosphopeptides and glycopeptides in electrostatic repulsion-hydrophilic interaction chromatography. Analytical Chemistry. 93 (22), 7908-7916 (2021).
- Cui, Y., et al. Finding the sweet spot in ERLIC mobile phase for simultaneous enrichment of N-Glyco and phosphopeptides. Journal of the American Society for Mass Spectrometry. 30 (12), 2491-2501 (2019).
- Tabang, D. N., et al. Analysis of pancreatic extracellular matrix protein post-translational modifications via electrostatic repulsion-hydrophilic interaction chromatography coupled with mass spectrometry. Molecular Omics. 17 (5), 652-664 (2021).
- Huang, J., et al. Dual-functional Titanium(IV) immobilized metal affinity chromatography approach for enabling large-scale profiling of protein Mannose-6-Phosphate glycosylation and revealing its predominant substrates. Analytical Chemistry. 91 (18), 11589-11597 (2019).
- Huang, J., et al. Dual-functional Ti(IV)-IMAC material enables simultaneous enrichment and separation of diverse glycopeptides and phosphopeptides. Analytical Chemistry. 93 (24), 8568-8576 (2021).
- Jami-Alahmadi, Y., Pandey, V., Mayank, A. K., Wohlschlegel, J. A. A robust method for packing high resolution C18 RP-nano-HPLC columns. Journal of Visualized Experiments: JoVE. (171), e62380 (2021).
- Bern, M., Kil, Y. J., Becker, C. Byonic: advanced peptide and protein identification software. Current Protocols in Bioinformatics. , (2012).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).
- Zhou, Y., et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets. Nature Communications. 10 (1), 1523 (2019).
- Zacharias, L. G., et al. HILIC and ERLIC enrichment of glycopeptides derived from breast and brain cancer cells. Journal of Proteome Research. 15 (10), 3624-3634 (2016).
- Yang, W., et al. Comparison of enrichment methods for intact N- and O-linked glycopeptides using strong anion exchange and hydrophilic interaction liquid chromatography. Analytical Chemistry. 89 (21), 11193-11197 (2017).
- Toghi Eshghi, S., Shah, P., Yang, W., Li, X., Zhang, H. GPQuest: A spectral library matching algorithm for site-specific assignment of tandem mass spectra to intact N-glycopeptides. Analytical Chemistry. 87 (10), 5181-5188 (2015).
- Liu, M. -. Q., et al. pGlyco 2.0 enables precision N-glycoproteomics with comprehensive quality control and one-step mass spectrometry for intact glycopeptide identification. Nature Communications. 8 (1), 438 (2017).
- Lu, L., Riley, N. M., Shortreed, M. R., Bertozzi, C. R., Smith, L. M. O-Pair Search with MetaMorpheus for O-glycopeptide characterization. Nature Methods. 17 (11), 1133-1138 (2020).
- Caval, T., Heck, A. J. R., Reiding, K. R. Meta-heterogeneity: Evaluating and describing the diversity in glycosylation between sites on the same glycoprotein. Molecular & Cellular Proteomics. 20, 100010 (2021).
- Lee, J. S., Smith, E., Shilatifard, A. The language of histone crosstalk. Cell. 142 (5), 682-685 (2010).
- Leutert, M., Entwisle, S. W., Villén, J. Decoding post-translational modification crosstalk with proteomics. Molecular & Cellular Proteomics. 20, 100129 (2021).
- Hart, G. W., Slawson, C., Ramirez-Correa, G., Lagerlof, O. Cross talk between O-GlcNAcylation and phosphorylation: Roles in signaling, transcription, and chronic disease. Annual Review of Biochemistry. 80 (1), 825-858 (2011).

