Plate-forme analytique multimodale sur une puce d’imagerie par résonance plasmonique de surface multiplexée pour l’analyse de sous-ensembles de vésicules extracellulaires
Summary
Cet article propose une nouvelle génération de plateformes analytiques multiparamétriques avec un débit accru pour la caractérisation des sous-ensembles de vésicules extracellulaires. La méthode est basée sur une combinaison de méthodes de biodétection multiplexées avec des analyses métrologiques et morphomécaniques par microscopie à force atomique, couplée à la spectroscopie Raman, pour qualifier des cibles vésiculaires piégées sur une biopuce de microréseau.
Abstract
Les vésicules extracellulaires (VE) sont de minuscules vésicules dérivées de la membrane produites par toutes les cellules qui vont de 50 à plusieurs centaines de nanomètres de diamètre et sont utilisées comme moyen de communication intercellulaire. Ils apparaissent comme des outils diagnostiques et thérapeutiques prometteurs pour diverses maladies. Il existe deux principaux processus de biogenèse utilisés par les cellules pour produire des VE avec des différences de taille, de composition et de contenu. En raison de leur grande complexité en taille, composition et origine cellulaire, leur caractérisation nécessite une combinaison de techniques analytiques. Ce projet comprend le développement d’une nouvelle génération de plateformes analytiques multiparamétriques avec un débit accru pour la caractérisation de sous-populations de VE. Pour atteindre cet objectif, les travaux partent de la plateforme nanobioanalytique (NBA) mise en place par le groupe, qui permet une étude originale des VE basée sur une combinaison de méthodes de biodétection multiplexées avec des analyses métrologiques et morphomécaniques par microscopie à force atomique (AFM) de cibles vésiculaires piégées sur une biopuce de microarray. L’objectif était de compléter cette étude EV par une analyse phénotypique et moléculaire par spectroscopie Raman. Ces développements permettent de proposer une solution analytique multimodale et facile à utiliser pour la discrimination des sous-ensembles de VE dans les fluides biologiques à potentiel clinique
Introduction
L’intérêt croissant pour la recherche sur les VE dans le diagnostic et la thérapeutique 1,2,3,4,5, combiné aux défis auxquels ce domaine est confronté, a entraîné le développement et la mise en œuvre d’une grande variété d’approches et de techniques pour quantifier ou caractériser ces vésicules. Les méthodes les plus largement utilisées pour l’identification des VE sont l’immunoblot et la protéomique spécifiques aux protéines pour confirmer l’origine des VE, la microscopie électronique à transmission (MET) pour confirmer leur structure et l’analyse de suivi des nanoparticules (NTA) pour quantifier leur nombre et leur distribution de taille dans un volume d’échantillon.
Cependant, aucune de ces techniques ne donne à elle seule toutes les informations nécessaires pour caractériser les sous-ensembles de véhicules électriques. L’hétérogénéité inhérente des VE due à la diversité de leurs propriétés biochimiques et physiques empêche les analyses globales fiables et reproductibles, en particulier pour les VE contenus dans un mélange (échantillon brut). Des méthodes de détection et de caractérisation sont donc nécessaires pour les véhicules électriques, à la fois individuellement et généralement pour compléter d’autres méthodes plus rapides mais non sélectives6.
L’imagerie haute résolution par TEM (ou cryoTEM) ou AFM permet de déterminer la morphologie et la métrologie des VE avec une résolution nanométrique 7,8,9,10,11,12. Cependant, la principale limitation de l’utilisation de la microscopie électronique pour les objets biologiques, tels que les véhicules électriques, est la nécessité d’un vide pour effectuer l’étude qui nécessite la fixation et la déshydratation de l’échantillon. Une telle préparation rend difficile la traduction des structures observées à la morphologie EV en solution. Pour éviter cette déshydratation de l’échantillon, la technique de cryoTEM est la plus adaptée à la caractérisation EV13. Il est largement utilisé pour déterminer l’ultrastructure des véhicules électriques. L’immunomarquage des vésicules par des nanoparticules d’or biofonctionnalisées permet également d’identifier des sous-populations spécifiques de VE et de les distinguer des autres particules présentes dans un échantillon biologique complexe. Cependant, en raison du faible nombre de VE analysés par microscopie électronique, il est souvent difficile d’effectuer une caractérisation représentative d’un échantillon complexe et hétérogène.
Pour révéler cette hétérogénéité de taille, l’International Society for Extracellular Vesicles (ISEV) suggère d’analyser un nombre suffisant d’images à grand champ, accompagnées d’images plus petites, pour révéler des VE individuels à haute résolution14. L’AFM est une alternative aux approches optiques et aux techniques de diffraction électronique pour l’étude des véhicules électriques. Cette technique utilise une pointe tranchante maintenue par un porte-à-faux flexible qui scanne l’échantillon déposé sur un support, ligne par ligne, et ajuste la distance entre la pointe et les éléments présents grâce à une boucle de rétroaction. Cela permet de caractériser la topographie de l’échantillon et de recueillir des informations morphomécaniques15,16,17,18. Les VE peuvent être scannés par AFM soit après avoir été déposés sur un substrat atomiquement plat, soit après avoir été capturés sur un substrat spécifique fonctionnalisé par des anticorps, des peptides ou des aptamères pour caractériser les différentes sous-populations18,19. En raison de sa capacité à quantifier et à sonder simultanément la structure, la biomécanique et le contenu biomoléculaire membraneux des VE dans des échantillons biologiques complexes sans avoir besoin de prétraitement, d’étiquetage ou de déshydratation, l’AFM est maintenant de plus en plus utilisé pour caractériser les VE de manière fine et multiparamétrique dans des conditions physiologiques de température et de milieu.
Cet article propose une méthodologie utilisant une biopuce d’or de base capable d’être (bio)chimiquement fonctionnalisée dans un format multiplexé. Ce substrat est la pierre angulaire d’une puissante plateforme analytique combinant la biodétection de sous-ensembles de VE par résonance plasmonique de surface, et une fois les VE adsorbés/greffés ou immunocapturés sur la puce, AFM permet la caractérisation métrologique et morphomécanique des VE. Couplée à la signature Raman des sous-ensembles EV capturés sur la puce, cette plateforme analytique permet la qualification des VE présents dans les échantillons biologiques de manière sans marquage et sans aucune étape préanalytique. Cet article montre que la combinaison de techniques puissantes, assistées par une méthodologie très rigoureuse dans la préparation du substrat et l’acquisition de données, rend l’analyse EV approfondie, définitive et robuste.
Le principe de l’approche proposée est de préparer un substrat aurifère, d’adsorber/greffer ou de capturer les sous-types de VE, et de les scanner par AFM pour estimer la taille et la morphologie de chaque sous-ensemble de VE. De plus, ces VE adsorbés sont analysés par spectroscopie Raman. Ce substrat peut, en effet, présenter trois types d’interfaces de plus en plus complexes : nues, fonctionnalisées chimiquement, ou puces à lignand. Avant de décrire les différentes étapes du protocole, les lecteurs sont invités à se reporter à la présentation schématique de l’approche de la plateforme nanobioanalytique (NBA) à la figure 1, combinant l’imagerie par résonance plasmonique de surface (SPRi), l’AFM et la spectroscopie.
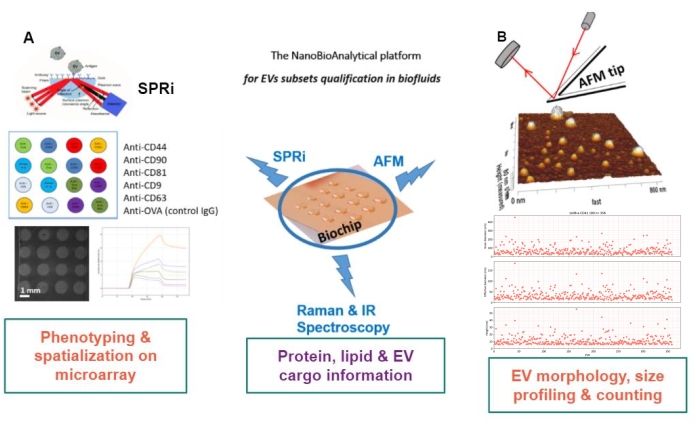
Figure 1 : La plateforme NanoBioAnalytical L’approche combine (A) l’imagerie par résonance plasmonique de surface, (B) la microscopie à force atomique et la (nano)spectroscopie infrarouge / Raman, toutes engagées sur le même substrat – une puce d’or multiplexée. Abréviations : NBA = NanoBioAnalytical platform; SPRi =imagerie par résonance plasmonique de surface; AFM = microscopie à force atomique; EV = vésicule extracellulaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La biopuce d’or de base constitue le cœur de la plateforme puisque toutes les techniques de caractérisation sans marquage sont réalisées sur cette biopuce. Selon les besoins de la caractérisation des VE (VE globaux/totaux ou sous-ensembles de VE) et les limites/exigences des méthodes utilisées, trois types de surfaces de biopuce d’or ont été développées : soit « nues », « C11/C16 » chimiquement fonctionnalisées ou biofonctionnalisées par ligand, appelées surface dorée « ligand ».
La biopuce nue, appelée « nue », permet l’adsorption simple de véhicules électriques sur l’or. Il est possible de sélectionner le tampon utilisé et de réaliser cette adsorption soit de manière passive (étapes d’incubation puis de rinçage), soit de la surveiller sous flux (en SPRi). De plus, cette adsorption passive peut être réalisée soit sur l’ensemble de la puce (sous forme de macroarray), soit localisée dans des microarrays à l’aide d’un observateur de micropipettes. La « procédure sous débit » permet aux enquêteurs de suivre la cinétique et le niveau d’adsorption de l’EV. Cette approche sur le substrat d’or nu est adoptée lorsque l’interface de la couche chimique peut interférer avec la méthode analytique (par exemple, pour la spectroscopie Raman).
La biopuce fonctionnalisée chimiquement, appelée « C11/C16 », est utilisée pour créer un « tapis » dense et robuste de véhicules électriques liés de manière covalente sur la surface de l’or en formant des liaisons amides primaires avec les thiolates lorsque l’objectif est d’avoir une vue globale de l’échantillon de VE. En effet, dans ce cas, l’or est fonctionnalisé par un mélange thiolate de mercapto-1-undécanol (11-MUOH : « C11 ») et d’acide mercapto-1-hexadécanoïque (16-MHA : « C16 »), et une fraction des thiolates est activée chimiquement pour établir une liaison covalente avec les cibles. Encore une fois, cette stratégie peut être réalisée soit passivement (étapes d’incubation puis de rinçage, soit dans un « macroarray » ou dans plusieurs microarrays à l’aide d’un observateur de micropipette) ou sous des débits (dans SPRi) pour suivre la cinétique et le niveau de greffage EV sur la surface de l’or.
La biopuce biofonctionnalisée par ligand, appelée « ligands », est activée chimiquement pour greffer par covalence différents ligands (p. ex. anticorps, récepteurs) afin de capturer sélectivement (avec affinité) différents sous-ensembles de VE qui coexistent dans l’échantillon biologique.
Protocol
Representative Results
Discussion
Les méthodes récentes d’identification des VE les plus largement utilisées sont l’immunoblot spécifique aux protéines pour confirmer l’origine des VE, la TEM pour confirmer leur structure et la NTA pour quantifier leur nombre et leur distribution granulométrique dans un échantillonde volume 3. Néanmoins, le grand intérêt pour les véhicules électriques dans la recherche (bio)médicale et les limites des outils analytiques existants ont incité la communauté scientifique à dével…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Kelly Aubertin et Fabien Picot de l’IVETh (Paris) sont reconnus pour les expériences d’imagerie Raman. Thierry Burnouf (Taipei Medical University, Taïwan) et Zuzana Krupova (d’Helincourt, France) sont reconnus pour avoir fourni les échantillons EV dérivés d’échantillons de plaquettes sanguines et de lait bovin, respectivement. Les travaux ont été soutenus par la région Bourgogne Franche-Comté et l’école doctorale EUR EIPHI (projet NOVICE, 2021-2024). Une partie de ce travail a été réalisée à l’aide de la plateforme CLIPP et dans les salles blanches RENATECH de FEMTO-ENGINEERING, pour lesquelles nous remercions Rabah Zeggari.
Materials
| CD41a antibody | Diaclone SAS (France) | 447528 | |
| CP920 | Microparticles GmbH, Germany | 448303 | |
| DXR3xi | Thermo Fisher Scientific | T1502 | |
| EDC | Sigma | A6272 | |
| Ethanolamine | Sigma | P5368-10PAK | |
| Evs derived from platelet concentrates | Collaboration : Pr T. Burnouf (TMU, Taipei) | S2889 | |
| Evs from bovine milk | Collaboration : Dr Z. Krupova (Excilone, Helincourt – France) | 3450 | |
| Glutaraldehyde | Sigma | 56845 | |
| Gwyddion | 853.223.020 | ||
| Magnetron sputtering | PLASSYS | SAB5300165 | |
| mercapto-1-hexadecanoic acid | Sigma | G5882 | |
| Mercapto-1-undecanol | Sigma | O8001 | |
| Mountains SPIP ones | Digital Surf | ||
| NanoWizard 3 Bioscience | Bruker-JPK | ||
| Octyl Glucoside (OG) | Sigma | ||
| Ovalbumine antibody | Sigma | ||
| Phosphate Buffer Saline (PBS) | Sigma | ||
| Rat Albumin Serum (RSA) | Sigma | ||
| Sodium acetate buffer | Sigma | ||
| SPR-Biacore 3000 | GE Healthcare/ Cytiva life sciences | ||
| SPRi Biochip | MIMENTO technology platform | The biochips were produced in-house in the clean room, Besancon | |
| SPRi Plex II | Horiba Scientific | ||
| Sulfo-NHS | Sigma |
References
- Silva, A. K. A., et al. Development of extracellular vesicle-based medicinal products: A position paper of the group "Extracellular Vesicle translatiOn to clinicaL perspectiVEs – EVOLVE France". Advanced Drug Delivery Reviews. 179, 114001 (2021).
- Xunian, Z., Kalluri, R. Biology and therapeutic potential of mesenchymal stem cell-derived exosomes. Cancer Science. 111 (9), 3100-3110 (2020).
- Hartjes, T. A., et al. Extracellular vesicle quantification and characterization: Common methods and emerging approaches. Bioengineering. 6 (1), 7 (2019).
- Xing, Y., et al. Analysis of extracellular vesicles as emerging theranostic nanoplatforms. Coordination Chemistry Reviews. 424, 213506 (2020).
- Wang, T., Xing, Y., Cheng, Z., Yu, F. Analysis of single extracellular vesicles for biomedical applications with especial emphasis on cancer investigations. Trends in Analytical Chemistry. 152, 116604 (2022).
- Boireau, W., Elie-Caille, C. Extracellular vesicles: Definition, isolation and characterization. Medecine Sciences: M/S. 37 (12), 1092-1100 (2021).
- Brisson, A. R., et al. Extracellular vesicles from activated platelets: A semiquantitative cryo-electron microscopy and immuno-gold labeling study. Platelets. 28 (3), 263-271 (2017).
- Yuana, Y., et al. Atomic force microscopy: A novel approach to the detection of nanosized blood microparticles. Journal of Thrombosis and Haemostasis. 8 (2), 315-323 (2010).
- Sebaihi, N., de Boeck, B., Yuana, Y., Nieuwland, R., Pétry, J. Dimensional characterization of extracellular vesicles using atomic force microscopy. Measurement Science and Technology. 28 (3), 034006 (2017).
- Beekman, P., et al. Immuno-capture of extracellular vesicles for individual multi-modal characterization using AFM, SEM and Raman spectroscopy. Lab on a Chip. 19 (15), 2526-2536 (2019).
- Malenica, M., et al. Perspectives of microscopy methods for morphology characterisation of extracellular vesicles from human biofluids. Biomedicines. 9 (6), 603 (2021).
- Verweij, F. J., et al. The power of imaging to understand extracellular vesicle biology in vivo. Nature Methods. 18 (9), 1013-1026 (2021).
- Théry, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): A position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. Journal of Extracellular Vesicles. 7 (1), 1535750 (2018).
- Obeid, S., et al. NanoBioAnalytical characterization of extracellular vesicles in 75-nm nanofiltered human plasma for transfusion: A tool to improve transfusion safety. Nanomedicine: Nanotechnology, Biology, and Medicine. 20, 101977 (2019).
- Obeid, S., et al. Development of a NanoBioAnalytical platform for «on-chip» qualification and quantification of platelet-derived microparticles. Biosensors and Bioelectronics. 93, 250-259 (2017).
- Ridolfi, A., et al. AFM-based high-throughput nanomechanical screening of single extracellular vesicles. Analytical Chemistry. 92 (15), 10274-10282 (2020).
- Vorselen, D., et al. The fluid membrane determines mechanics of erythrocyte extracellular vesicles and is softened in hereditary spherocytosis. Nature Communications. 9 (1), 4960 (2018).
- Hardij, J., et al. Characterisation of tissue factor bearing extracellular vesicles with AFM: Comparison of air-tapping-mode AFM and liquid Peak Force AFM. Journal of Extracellular Vesicles. 2, 21045 (2013).
- Jorgensen, M., et al. Extracellular Vesicle (EV) Array: Microarray capturing of exosomes and other extracellular vesicles for multiplexed phenotyping. Journal of Extracellular Vesicles. 2, 20920 (2013).
- Remy-Martin, F., et al. Surface plasmon resonance imaging in arrays coupled with mass spectrometry (SUPRA-MS): Proof of concept of on-chip characterization of a potential breast cancer marker in human plasma. Analytical and Bioanalytical Chemistry. 404 (2), 423-432 (2012).
- Czamara, K., et al. Raman spectroscopy of lipids: A review. Journal of Raman Spectroscopy. 46 (1), 4-20 (2015).
- Penders, J., et al. Single particle automated Raman trapping analysis of breast cancer cell-derived extracellular vesicles as cancer biomarkers. ACS Nano. 15 (11), 18192-18205 (2021).
- Baek, S. J., Park, A., Ahn, Y. J., Choo, J. Baseline correction using asymmetrically reweighted penalized least squares smoothing. Analyst. 140 (1), 250-257 (2015).
- Daaboul, G. G., et al. Digital detection of exosomes by interferometric imaging. Scientific Reports. 6, 37246 (2016).
- Ertsgaard, C. T., et al. Integrated nanogap platform for sub-volt dielectrophoretic trapping and real-time Raman imaging of biological nanoparticles. Nano Letters. 18 (9), 5946-5953 (2018).
- Maas, S. L., et al. Possibilities and limitations of current technologies for quantification of biological extracellular vesicles and synthetic mimics. Journal of Controlled Release. 200, 87-96 (2015).

