RNA تنبؤ هيكل الثانوية باستخدام الشكل عالي الإنتاجية
Summary
عالية الإنتاجية انتقائية 2 'أسيلة الهيدروكسيل تحليلها من قبل التمديد التمهيدي (SHAPE) تستخدم مادة كيميائية رواية التحقيق التكنولوجيا، النسخ العكسي، الكهربائي الشعري والبرمجيات التنبؤ هيكل ثانوي لتحديد هياكل الرنا من عدة مئات إلى عدة آلاف من النيوكليوتيدات في قرار النوكليوتيدات واحد.
Abstract
فهم وظيفة الحمض النووي الريبي المشاركة في العمليات البيولوجية تتطلب معرفة وافية من بنية الحمض النووي الريبي. وتحقيقا لهذه الغاية، ومنهجية يطلق عليها اسم "عالية الإنتاجية انتقائية 2 'أسيلة الهيدروكسيل تحليلها من قبل التمديد التمهيدي"، أو الشكل، يسمح التنبؤ من الحمض النووي الريبي هيكل الثانوي لقرار النوكليوتيدات واحد. هذا النهج يستخدم وكلاء التحقيق الكيميائية التي acylate تفضيلي المناطق الذين تقطعت بهم السبل أو مرنة واحدة من الحمض النووي الريبي في محلول مائي. تم الكشف عن مواقع التعديل الكيميائي بواسطة النسخ العكسي من الحمض النووي الريبي معدلة، والمنتجات من هذا التفاعل هي مجزأة من قبل الكهربائي الشعري الآلي (CE). منذ الناسخ العكسي مؤقتا في تلك النيوكليوتيدات RNA تعديلها من قبل الكواشف الشكل، مكتبة [كدنا] الناتجة خرائط غير مباشرة تلك ribonucleotides أن تقطعت بهم السبل واحدة في سياق RNA مطوية. باستخدام البرمجيات ShapeFinder، تتم معالجة electropherograms التي تنتجها الآلي CE وتحويلها إلى نوالجداول تفاعل cleotide التي يتم تحويلها إلى أنفسهم القيود الزائفة الطاقة المستخدمة في RNAStructure (V5.3) خوارزمية التنبؤ. وقد تم العثور على هياكل RNA ثنائي الأبعاد التي تم الحصول عليها من خلال الجمع بين SHAPE التحقيق مع الحمض النووي الريبي في سيليكون والتنبؤ هيكل ثانوي لتكون أكثر دقة من الهياكل التي تم الحصول عليها باستخدام أي من الطريقتين وحدها.
Introduction
لفهم وظائف الرنا الحفاز وغير الترميز المشاركة في تنظيم الربط والترجمة وتكاثر الفيروس وسرطان، لا بد من معرفة تفصيلية للبنية الحمض النووي الريبي 1،2. للأسف، تنبؤ دقيق من الحمض النووي الريبي للطي يمثل تحديا هائلا. وكلاء التحقيق الكلاسيكية تعاني من عيوب كثيرة مثل سمية، والتغطية النوكليوتيدات غير مكتملة و / أو إنتاجية محدودة إلى 100-150 النيوكليوتيدات في التجربة. خوارزميات التنبؤ المجردة هيكل الثانوي غير ملائم وبالمثل، نظرا لعدم الدقة الناجمة عن عدم قدرتها على التمييز بين فعالية هياكل مماثلة بقوة. الرنا كبيرة على وجه الخصوص هي أيضا في كثير من الأحيان إلى صهر أساليب تحديد هيكل 3D مثل البلورات بالأشعة السينية والرنين المغناطيسي النووي (NMR) التحليل الطيفي، وذلك بسبب مرونتها ومتعلق بتكوين كميات كبيرة من عينات نقية للغاية المطلوبة لهذه التقنيات.
HSHAPE IGH الإنتاجية يحل الكثير من هذه المشاكل عن طريق توفير نهج بسيطة فعالة ليحقق في هياكل الرنا كبير في قرار النوكليوتيدات واحد. وعلاوة على ذلك، والكواشف المستخدمة لشكل هي آمنة وسهلة للتعامل، وعلى النقيض من معظم الكواشف الكيميائية الأخرى التحقيق، تتفاعل مع جميع ribonucleotides أربعة. ويمكن لهذه الكواشف أيضا اختراق الأغشية الخلوية، مما يجعل من الممكن للتحقيق في الرنا في سياق الجسم الحي (ق) 3. وضعت أصلا في الأسابيع مختبر 4، وقد استخدمت SHAPE لتحليل مجموعة متنوعة واسعة من الرنا، والمثال أبرزها تقرير للهيكل الثانوي كاملة من ~ 9 KB HIV-1 الحمض النووي الريبي الجينوم 5. الإنجازات البارزة الأخرى باستخدام SHAPE تشمل توضيح لهياكل أشباه الفيروسات المعدية 6، الرناوات غير الترميز طويلة الإنسان 7، 8 ريبوسوم الخميرة، وriboswitches 9 وكذلك لتحديد مواقع الربط البروتين في الفيريون المرتبطة HIV-1 RNA 3. WHوقد نشرت إيل الاختلافات الأصلي وعالية الإنتاجية من البروتوكول SHAPE في أماكن أخرى 10-12، ويوفر هذا العمل وصفا مفصلا لتحديد الحمض النووي الريبي هيكل الثانوي بحلول SHAPE الإنتاجية العالية باستخدام [أليغنوكليوتيد الفلورسنت، وكولتر CEQ 8000 محلل الوراثية بيكمان، و SHAPEfinder وRNAStructure (V5.3) والبرمجيات. كما تشمل التفاصيل الفنية لم تنشر من قبل والمشورة استكشاف الأخطاء وإصلاحها.
الاختلافات من الشكل
جوهر الشكل وتنوعاتها هو التعرض من الحمض النووي الريبي في محلول مائي لالانهيدريدات بالإلكترونات التي acylate انتقائي 2'-الهيدروكسيل (OH-2') مجموعات الريبوز، وتنتج adducts ضخمة في مواقع التعديل. هذا التفاعل الكيميائي يخدم كوسيلة لاستجواب ديناميات RNA المحلية الهيكلي، كما النيوكليوتيدات الذين تقطعت بهم السبل واحد هم أكثر عرضة للاعتماد التشكل مواتية لهجوم من قبل بالإلكترونات هذه الكواشف، بينما قاعدة الاقتران أو للإنشاء معمارياالنيوكليوتيدات ained أقل أو يتفاعل 10. تم الكشف عن مواقع تشكيل معقد إضافي عن طريق النسخ العكسي بدء الاشعال من fluorescently أو رديولبلد المهجنة إلى موقع معين على الحمض النووي الريبي المحورة (و"(+)" رد فعل الإرشاد التمهيدي). عندما يفشل الناسخ العكسي (RT) لاجتياز ribonucleotides acylated، ويتم إنتاج مجموعة من المنتجات [كدنا] الذي يتزامن مع مواقع من تعديل أطوال. A السيطرة "(-)" التمهيدي التمديد يتم تنفيذ رد فعل باستخدام الحمض النووي الريبي التي لم تتعرض للكاشف أيضا بحيث الإنهاء المبكر من توليف الحمض النووي (أي "توقف") بسبب الحمض النووي الريبي هيكل، غير محدد RNA حبلا الكسر، وغيرها، قد. يمكن تمييزها عن التوقف التي تنتجها التعديل الكيميائي. وأخيرا، يتم استخدام اثنين من ردود الأفعال dideoxy التسلسل بدء من نفس الاشعال كعلامات لربط النيوكليوتيدات رد الفعل مع تسلسل الحمض النووي الريبي الأولية بعد الكهربائي.
في التطبيق الأصلي من حالات العسر الشديدويستخدم E، وهو نفس نهاية 32 ف المسمى التمهيدي ل(+)، (-)، واثنين من التفاعلات التسلسل. يتم تحميل المنتجات من هذه التفاعلات إلى الآبار المجاورة في 5-8٪ هلام بولي أكريلاميد بلاطة، ومجزأة عن طريق تغيير طبيعة بولي أكريلاميد الكهربائي للهلام (PAGE؛ الشكل 1). التحليل الكمي من الصور هلام التي تنتجها SHAPE التقليدية يمكن القيام بها باستخدام SAFA، شبه الآلي برامج التحليل footprinting 13.
في المقابل، شكل عالية الإنتاجية توظف الاشعال fluorescently المسمى والكهربائي الشعري الآلي. على وجه التحديد، لكل منطقة من الحمض النووي الريبي قيد التحقيق، مجموعة من أربعة الاشعال DNA وجود تسلسل مشتركة ولكن مختلفة 5 'يجب توليفها تسميات الفلورسنت أو شراؤها. هذه أليغنوكليوتيد] بشكل مختلف المسمى تخدم لرئيس اثنين من ردود الفعل SHAPE واثنين من ردود الفعل التسلسل، والمنتجات التي يتم تجميع ومجزأة / الكشف عنها من قبل الكهربائي الشعري الآلي (CE). وهرEAS الملف الشخصى التفاعل من 100-150 NT من الحمض النووي الريبي يمكن الحصول عليها من مجموعة من أربعة تفاعلات باستخدام النهج الأصلي، وشكل عالية الإنتاجية يسمح قرار من 300-600 NT من واحدة عينة مجمعة 3. ما يصل إلى 8 مجموعات من ردود الفعل قد تكون مجزأة في وقت واحد، في حين يمكن للما يصل الى 96 عينات أعدت للتجزئة على مدى 12 أشواط متتالية CE (الشكل 2). وعلاوة على ذلك، فإن البرنامج SHAPEfinder، وضعت لمعالجة البيانات الناشئة من CEQ وتحليل وراثية أخرى وتحليلها، هو أكثر الآلي وتتطلب تدخل المستخدم أقل بكثير من SAFA 13 أو حزم هلام التحليل الأخرى.
ظهرت منهجيات عالية الإنتاجية أكثر تقدما في الآونة الأخيرة مثل PARS (تحليل موازية للبنية الحمض النووي الريبي) 14 وفرج تسلسل (جزء التسلسل) 15، والتي تستخدم الإنزيمات بنية محددة بدلا من الكواشف الألكلة بالتزامن مع الجيل القادم التسلسل التقنيات للحصول على المعلومات!ن حول بنية الحمض النووي الريبي. وعلى الرغم من جاذبية هذه التقنيات، العديد من القيود الملازمة لنوكلياز التحقيق لا تزال 16. يمكن التحايل هذه المشاكل في التسلسل SHAPE (SHAPE تسلسل) 17 البروتوكول، حيث سبق التسلسل التالي جيل عن طريق التعديل الكيميائي والنسخ العكسي من الرنا بطريقة مماثلة لتلك التي يؤديها في الشكل التقليدي. في حين أن هذه الأساليب قد يمثلون مستقبل تحديد بنية الحمض النووي الريبي، من المهم أن نتذكر أن التسلسل التالي جيل مكلفة للغاية، ويبقى غير متاحة لكثير من المختبرات.
تحليل بيانات الشكل
يتم تقديم البيانات المنتجة في محلل الوراثية في شكل رحلاني، حيث كثافة مضان من العينة (ق) التي تتدفق من خلال كاشف الشعرية المرسومة ضد فهرس من وقت الهجرة. هذه المؤامرة تأخذ شكل آثار التداخل المقابلة لقناة مضان أربعةق المستخدمة للكشف عن fluorophores مختلفة، وحيث يتكون كل أثر للقمم المقابلة لمنتجات [كدنا] تسلسل أو الفردية. ويتم تصدير البيانات رحلاني من محلل الوراثية كملف نصي المفصول والمستوردة إلى التحول ShapeFinder وتحليل البرمجيات 18.
يستخدم ShapeFinder في البداية لتنفيذ سلسلة من التحولات رياضية على البيانات لضمان أن أوقات الهجرة وأحجام الذروة تعكس بدقة الهويات وكميات من منتجات التفاعل، على التوالي. ثم يتم محاذاة قمم ومتكاملة، ونتائج جدولتها مع تسلسل الحمض النووي الريبي الأولية. يتم الحصول على "لمحة تفاعل" للجزء ذات الصلة من الحمض النووي الريبي من خلال طرح القيم التحكم من (+) القيم المرتبطة مع بعضها RNA النوكليوتيدات، وتطبيع البيانات كما هو موضح أدناه. يتم استيراد هذا الملف إلى RNAstructure (V5.3) البرامج 19،20، والذي يحول فال تفاعل تطبيعUES إلى القيود الزائفة الطاقة التي يتم دمجها في بنية الحمض النووي الريبي الثانوية خوارزمية قابلة للطي. الجمع بين المواد الكيميائية التحقيق وقابلة للطي الخوارزميات في هذه الطريقة بشكل كبير على تحسين دقة التنبؤ بنية مقارنة إما طريقة وحدها 12،21. إخراج RNAstructure (V5.3) يتضمن صورا لأقل الطاقة RNA الهياكل الثانوية مرمزة مع ملف SHAPE تفاعل (ق)، وكذلك نفس الهياكل في تدوين النصوص الدوت قوس. قد في وقت لاحق يتم تصديرها هذا الأخير إلى برامج مخصصة لعرض رسومية من بنية الحمض النووي الريبي الثانوية مثل فارنا 22 و 23 PseudoViewer.
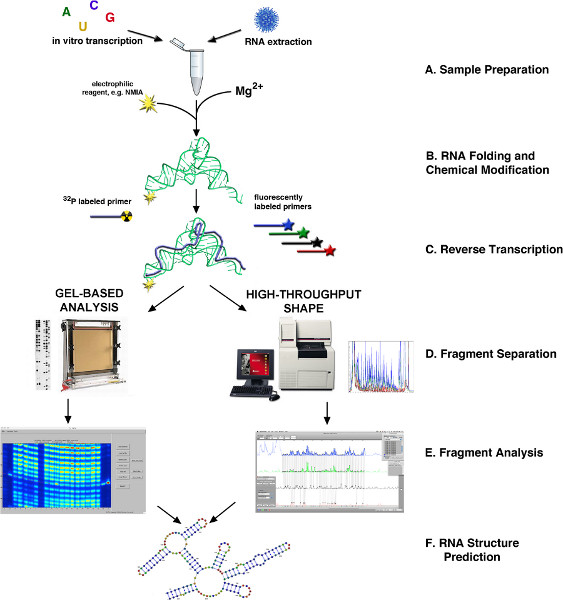
الشكل 1. انسيابي من عزم هيكل الحمض النووي الريبي عبر SHAPE (4،10). (A) RNA ميمكن الحصول على AY من العينات البيولوجية أو بواسطة نسخ في المختبر. (B) اعتمادا على مصدر، يتم طي RNA أو محضرة بطريقة أخرى وتعديلها مع كاشف الشكل. (C) النسخ العكسي باستخدام بادئات fluorescently المسمى أو بالإشعاع. (D) منتجات كدنا] هي مجزأة إما عن طريق الكهربائي الشعري أو لوح المستندة إلى هلام. (E) تحليل شظية. (F) التنبؤ بنية الحمض النووي الريبي. انقر هنا لعرض أكبر شخصية.
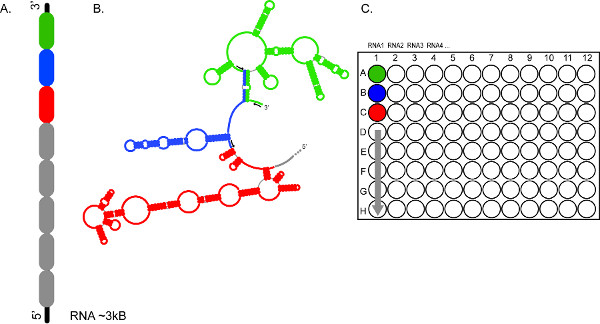
الشكل 2. الطابع الإنتاجية العالية من الشكل القائم على CE يسمح تحليل سريع من الرنا متعددة، و / أو شرائح متعددة من نفس الحمض النووي الريبي. (A) </stرونغ> يمثل كيف يمكن تقسيم على الحمض النووي الريبي إلى 300-600 أقسام NT (ونا مميزا باللون الأخضر والأزرق والأحمر) (B) يتم بحثها أقسام من الحمض النووي الريبي بشكل مستقل باستخدام مجموعات مختلفة من بادئات الفلورسنت (الأسهم السوداء) (C) مجموعة من يتم تجميع ردود الفعل وتحميلها في الآبار A1، B1، C1، الخ، على التوالي، وتوفير تغطية كاملة لل~ 3 KB RNA1. منتجات التفاعل من الرنا 2، 3، 4، وما إلى ذلك قد تكون مستعدة بالمثل لتجزيء في أشواط متتالية الكهربي. انقر هنا لعرض أكبر شخصية.
Protocol
Representative Results
Discussion
نقدم هنا بروتوكول مفصلة لشكل الإنتاجية العالية، وهي تقنية تسمح تحديد هيكل الثانوي لقرار النوكليوتيدات واحد لالرنا من أي حجم. وعلاوة على ذلك، اقتران بيانات الشكل التجريبية مع خوارزميات التنبؤ هيكل ثانوي يسهل جيل من الحمض النووي الريبي نماذج 2D مع درجة أعلى من دقة مما …
Disclosures
The authors have nothing to disclose.
Acknowledgements
معتمدة S. Lusvarghi، J. Sztuba-Solinska، KJ Purzycka، JW RAUSCH وSFJ لو جريس من قبل برنامج بحوث جماعية للمعهد الوطني للسرطان والمعاهد الوطنية للصحة بالولايات المتحدة الأمريكية.
Materials
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5′ labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied – including any 3’terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies |
References
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 ‘-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 ‘-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2′-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues – A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).

