높은 처리량 SHAPE를 사용하여 RNA 이차 구조 예측
Summary
프라이머 확장 (SHAPE)에 의해 분석 높은 처리량의 선택 2 '수산기 실화는 단일 염기 해상도에서 수천 개의 뉴클레오티드로 수백에서의 RNA 구조를 결정하는 기술을 프로빙 소설 화학, 역전사, 모세관 전기 영동 및 이차 구조 예측 소프트웨어를 사용합니다.
Abstract
생물 학적 과정에 관여 RNA의 기능을 이해하는 것은 RNA 구조의 철저한 지식이 필요합니다. 이를 위해, 불리는 방법은, 또는 모양 "프라이머 확장하여 분석 높은 처리량의 선택 2 '수산기 실화는"단일 염기 해상도를 가진 RNA 이차 구조 예측을 할 수 있습니다. 이 방법은 우선적으로 수용액 RNA의 단일 좌초 또는 유연한 지역 acylate 화학 프로빙 에이전트를 사용합니다. 화학적 변형의 사이트는 수정 된 RNA의 역전사에 의해 감지하고,이 반응의 제품은 자동화 된 모세관 전기 영동 (CE)에 의해 분별입니다 수 있습니다. 역전사 효소는 SHAPE 시약에 의해 수정하는 RNA의 뉴클레오티드에서 일시 정지하기 때문에, 결과의 cDNA 라이브러리는 간접적으로 하나의 접힌 RNA의 맥락에서 좌초하는 리보 뉴클레오타이드를 매핑합니다. ShapeFinder 소프트웨어를 사용하여 자동화 CE에 의해 생성 된 electropherograms가 처리되고 뉴로 변환cleotide 반응성 테이블은 그 자체가 RNAStructure (V5.3) 예측 알고리즘에 사용 된 의사 에너지 제약 조건으로 변환됩니다. 실리코 RNA 이차 구조 예측에서와 프로브 형상을 결합하여 얻은 두 가지 차원 RNA 구조는 혼자 두 방법을 사용하여 얻은 구조보다 훨씬 더 정확한 것으로 판명되었다.
Introduction
접합, 번역, 바이러스 복제 및 암의 조절에 관여 촉매 및 비 코딩 RNA를의 기능을 이해하기 위해, RNA 구조의 상세한 지식은 1,2 필요합니다. 불행하게도, RNA 접이식의 정확한 예측은 강력한 도전을 선물한다. 고전 프로빙 에이전트는 고통과 같은 독성, 불완전 염기 범위 및 / 또는 실험 당 100 ~ 150 뉴클레오티드 제한 처리 등 많은 단점에서. 남의 차 구조 예측 알고리즘은 유사하게 불리한 효율적으로 정력적으로 유사한 구조를 구별하기 위해 무능력으로 인한 부정확성으로 인해 수 있습니다. 특히 큰 RNA를 또한 X-선 결정학과 구조적 유연성과 이러한 기술에 필요한 고순도 샘플을 대량으로 인해 분광법 (NMR) 공명 핵 자기와 같은 3D 구조를 결정하는 방법을 자주 내화물입니다.
H고등학교의 처리량 모양의 단일 염기 해상도로 대형의 RNA 구조를 프로빙에 효과적인 간단한 방법을 제공함으로써 많은 문제를 해결합니다. 또한, 모양에 사용되는 시약 처리 안전, 쉽고, 시약 프로브 대부분의 다른 화학 대조적으로, 네 개의 리보 뉴클레오티드로 반응합니다. 이 시약은 또한 가능한 생체 컨텍스트 (들) 3 자신의 RNA를을 조사하기 위해 만들어 세포막을 통과 할 수 있습니다. 원래 주 실험실 4에서 개발, 모양의 RNA 다양한 ~ 9킬로바이트 HIV-1 RNA 게놈 5의 전체 차 구조 결정되는 가장 주목할만한 예제를 분석하는 데 사용되었습니다. SHAPE를 사용하여 다른 주목할만한 업적은 전염성 로이드 6, 인간의 긴 비 코딩 RNAs를 7, 효모 리보솜 8 및 리보 스위치 9뿐만 아니라 식별 할 수 virion의 관련되는 HIV-1 RNA 3 단백질 결합 부위의 구조의 해명이 (가) 있습니다. WHSHAPE 프로토콜의 일 원본과 높은 처리량 변화는 다른 10-12 게시 된, 본 연구는 형광 올리고 뉴클레오타이드를, 베크 만 보습 바로 앞에 달린 풀 베는 CEQ 8000 유전자 분석기를 사용하여 높은 처리량의 형상에 의해 RNA 이차 구조 결정에 대한 자세한 설명을 제공하고, SHAPEfinder 및 RNAStructure (V5.3) 소프트웨어입니다. 이전에 발표되지 않은 기술적 인 세부 사항 및 문제 해결 조언도 포함되어 있습니다.
모양의 변형
SHAPE와 그 변화의 본질은 선택적으로 변형 사이트에서 부피 부가 물을 생산, 2'-히드 록시 (2'-OH) 리보오스 그룹을 acylate 전자 성 무수물로 수용액 RNA의 노출이다. 베이스 쌍 또는 구조적 생성자하는 동안이 화학 반응은 단일 가닥 핵산이 시약에 의한 전자 성 공격에 도움이되는 conformations에 채택하는 경향이기 때문에, 지역의 RNA 구조 역학을 심문하는 수단으로 역할을ained 뉴클레오티드가 작거나 10 반응성이다. 부가 생성물 형성의 사이트는 수정 된 RNA ( "(+)"프라이머 연장 반응)에 특정 사이트에 하이브리드 형광이나 방사성 동위 원소 프라이머에서 시작 역전사에 의해 감지됩니다. 역전사 효소 (RT)는 실화 리보 뉴클레오타이드를 통과하는 데 실패하면, cDNA를 제품의 풀은 그 길이 수정의 사이트와 일치 생산됩니다. 컨트롤은 "(-)"프라이머 확장 시약에 노출되지 않은 RNA를 활용 반응은 그렇게 수행되는 구조 특이 RNA 가닥 파손 등, 5 월을 RNA에 의한 DNA 합성 (예 : "중지")의 조기 종료. 화학적 변형에 의해 생성 일시 중지 구분할 수. 마지막으로, 같은 프라이머에서 시작 두 dideoxy 시퀀싱 반응은 RNA 기본 순서가 전기를 다음과 같이 반응 뉴클레오티드의 상관 관계를 마커로 사용됩니다.
SHAP의 원래 응용 프로그램에서(-), 두 개의 연속 반응 E, 같은 32 P-끝 레이블 프라이머 (+)를 위해 사용됩니다. 이러한 반응의 제품은 5-8 %의 폴리 아크릴 아미드 슬래브 겔에 인접 우물에로드 폴리 아크릴 아마이드 겔 전기 영동 (PAGE, 그림 1) 변성에 의해 분별하고 있습니다. 기존의 모양에 의해 생성 된 겔 이미지의 정량 분석은 사파, 반자동 배출량 측정 분석 소프트웨어 13를 사용하여 수행 할 수 있습니다.
반면, 높은 처리량 모양 찬란 프라이머 및 자동화 모세관 전기 영동을 사용합니다. 특히, 조사 대상 RNA, 일반적인 순서 만 다른 5 '형광 라벨을 합성하거나 구입해야하는 데 네 가지 DNA 프라이머 세트의 각 지역. 이러한 다르게 표지 된 올리고 뉴클레오티드는 주요 두 가지 모양의 반응과 두 개의 연속 반응, 풀링 그리고 분별 / 자동 모세관 전기 영동 (CE)에 의해 감지되는의 제품을 제공합니다. 실어EAS는 RNA의 100 ~ 150 NT의 반응 프로파일은 원래의 방법을 사용하여 네 가지 반응의 집합에서 얻을 수있는, 높은 처리량 SHAPE는 단일 풀링 된 샘플 3 300-600 NT의 해상도를 할 수 있습니다. 많은 96 샘플 12 연속 CE가 실행 (그림 2)의 과정을 통해 분별을 준비 할 수있는 동안, 동시에 분별 할 수 있습니다 최대 반응의 8 세트까지. 또한, CEQ와 다른 유전자 분석기에서 나오는 데이터를 처리하고 분석하기 위해 개발 SHAPEfinder 소프트웨어는 더 자동화 및 SAFA 13이나 젤 분석 패키지보다 훨씬 적은 사용자 개입이 필요합니다.
고급 높은 처리량 방법은 최근에 이러한 얻기 위해 기술을 시퀀싱 다음 세대와 함께 구조 특정 효소보다는 알킬화 시약을 사용 PARS (RNA 구조의 병렬 분석) 14 수류탄 – 시퀀서 (조각 시퀀싱) 15로 등장 정 보RNA 구조에 대한 N. 이러한 기술의 매력에도 불구하고, 프로브 핵산에 내재 많은 제한은 여전히 16 남아있다. 이러한 문제는 차세대 시퀀싱은 기존의 모양에서 수행하는 것과 유사한 방식으로 화학적 변형과의 RNA 역전사 앞에는 SHAPE 시퀀싱 (SHAPE-시퀀서) 17 프로토콜에서 회피 할 수있다. 이러한 방법은 RNA 구조 결정의 미래를 나타낼 수 있지만, 그것은 차세대 시퀀싱은 매우 고가이며, 많은 실험실에 사용할 수없는 상태 것을 기억하는 것이 중요합니다.
셰이프 데이터 분석
유전자 분석기에서 생산 데이터를 마이그레이션 시간의 인덱스에 그려지는 모세관 검출기를 통과하는 시료의 형광 강도 (들)을 상기, electropherogram의 형태로 제공됩니다. 이 그래프는 네 개의 형광 채널에 해당하는 중복 흔적의 형태를 취s는 서로 다른 형광 물질을 감지하는 데 사용되며, 각 추적은 개별 cDNA를 또는 시퀀싱 제품에 해당하는 봉우리 구성입니다. Electropherogram 데이터는 탭으로 구분 된 텍스트 파일로 유전자 분석기에서 보낸 ShapeFinder 변환 및 분석 소프트웨어를 18로 가져옵니다.
ShapeFinder는 처음 마이그레이션 시간과 최대 볼륨이 정확하게 각각 반응 생성물의 ID 및 수량을 반영하도록 데이터를 수학적 일련의 변환을 수행하는 데 사용됩니다. 피크는 다음 정렬 및 통합, 결과는 기본 RNA 시퀀스와 함께 표로 있습니다. RNA의 관련 세그먼트에 대한 "반응 프로파일은"(+) 염기 각 RNA와 관련된 값과 제어 값을 빼서, 아래 설명 된대로 데이터를 정규화하여 얻을 수 있습니다. 이 프로필은 정규화 된 반응성 발을 변환 (V5.3) 소프트웨어 19,20에게, RNAstructure로 가져RNA 이차 구조의 접이식 알고리즘에 통합 의사 에너지 제약에 단말. 이 방법으로 알고리즘을 프로빙 및 접는 화학 물질을 결합하여 현저하게 혼자 12,21 두 방법에 비해 구조 예측의 정확성을 향상시킵니다. RNAstructure의 출력 (V5.3)은 낮은 에너지의 이미지가 포함되어 RNA 이차 구조의 텍스트 점 괄호 표기법 SHAPE 반응성 프로파일 (들)뿐만 아니라 동일한 구조를 색으로 구분. 후자는 이후 같은 바르나 22 PseudoViewer 23와 같은 RNA 이차 구조의 그래픽 디스플레이 전용 소프트웨어로 내보낼 수 있습니다.
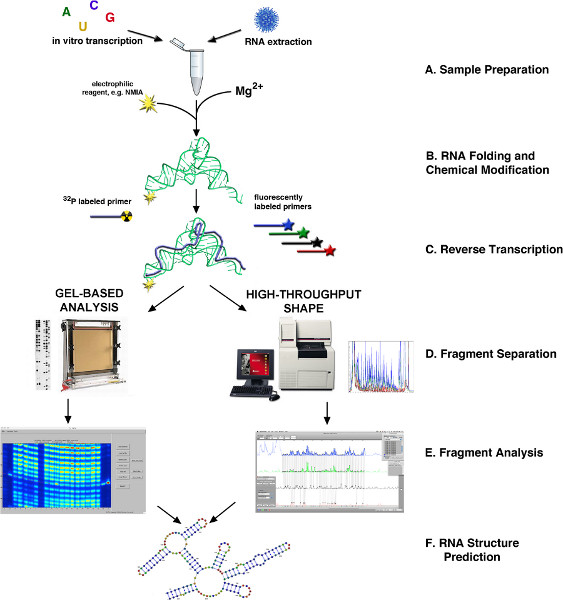
그림 1. SHAPE 4,10를 통해 RNA의 구조 결정의 흐름도. (A) RNA M바깥은 생물 학적 샘플이나 시험 관내 전사하여 얻을 수. (B) 소스에 따라, RNA는 접하거나 처리 SHAPE 시약으로 변경됩니다. (C) 역 전사 형광이나 방사성 표시 프라이머를 사용하여. (D)의 cDNA 제품은 하나 모세관 또는 슬래브 겔 기반의 전기를 통해 분별. (E) 조각 분석. (F) RNA 구조 예측. 큰 그림을 보려면 여기를 클릭하십시오.
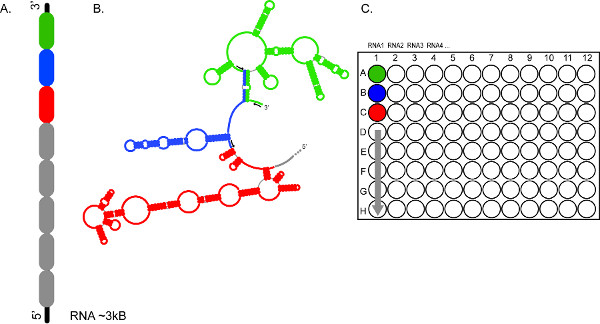
그림 2. CE 기반 SHAPE의 높은 처리량 문자 신속한 여러 RNA를 분석, 및 / 또는 동일한 RNA의 여러 세그먼트를 할 수 있습니다. (A) </st룽>는 RNA가 300-600 NT 섹션 (색, 녹색, 파란색과 빨간색으로 구분) (B) RNA의 섹션은 형광 프라이머의 집합을 (검은 색 화살표)를 사용하여 독립적으로 조사됩니다 (C)의 집합으로 나눌 수 있습니다 방법을 나타냅니다 반응은 ~ 3킬로바이트 RNA1에 대한 완전한 범위를 제공, 각각 우물 A1, B1, C1, 등으로 풀링 및로드됩니다. RNA를 2, 3, 4 등의 반응 제품은 유사하게 연속 전기 영동 실행의 분별을 준비 할 수 있습니다. 큰 그림을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
우리는 여기서 높은 처리량 SHAPE에 대한 자세한 프로토콜, 어떤 크기의 RNA를위한 단일 염기 해상도로 차 구조 결정을 할 수있는 기법을 제시한다. 또한, 이차 구조 예측 알고리즘을 실험 SHAPE 데이터를 결합하는 것은 혼자 두 방법으로 가능한 것보다 높은 정확도와 RNA 차원 모델의 생성을 촉진한다. 찬란 – 라벨 프라이머 및 자동 CE의 조합은 하나의 실험에서 긴 RNA 시퀀스의 해상도를 촉진 기존의 ?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
S. Lusvarghi, J. Sztuba-Solinska, KJ Purzycka, JW Rausch의 및 SFJ 르 Grice는 국립 암 연구소, 건강의 국립 연구소, 미국의 교내 연구 프로그램에 의해 지원됩니다.
Materials
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5′ labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied – including any 3’terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies |
References
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 ‘-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 ‘-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2′-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues – A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).

