РНК предсказание вторичной Структура Использование высоких-пропускная способность SHAPE
Summary
Высокая пропускная способность селективного 2 'гидроксильные ацилирование анализировали с помощью удлинения праймера (ФОРМА) используется новый химический зондирования технологии обратной транскрипции, капиллярный электрофорез и вторичных программное обеспечение предсказания структуры для определения структуры РНК от нескольких сотен до нескольких тысяч нуклеотидов в единичных нуклеотидных разрешение.
Abstract
Понимании функции РНК, участвующих в биологических процессах требует тщательной знания в области конструкций РНК. К этому концу, методология дублированный "гидроксил ацилирование высокой пропускной способностью селективными 2 'анализировали с помощью удлинения праймера», либо SHAPE, позволяет предсказание из РНК вторичной структурой с единичные нуклеотидные разрешение. Этот подход использует химические зондирования агенты, которые предпочтительно ацилирования одноцепочечной или гибких участков РНК в водном растворе. Сайты из химическую модификацию обнаруживаются путем обратной транскрипции из модифицированному РНК, и продукты из этой реакции являются фракционировали с помощью автоматизированный капиллярный электрофорез (СЕ). Так как обратной транскриптазы паузами по крайней такие нуклеотиды, РНК модифицированный по SHAPE реагенты, результирующие кДНК-библиотеку косвенно отображает тех, рибонуклеотиды, которые имеют одно на мель в контекст из сложенной РНК. Используя ShapeFinder программное обеспечение, то электрофореграммы произведенный по автоматизированный CE, обрабатываются и преобразуется в нюcleotide таблицам реактивностью, которые сами являются преобразованы в псевдо-с ограничениями энергопотребления,, используемый в RNAStructure (v5.3) Prediction алгоритму. Двумерной РНК структурах, получаемых соединением SHAPE зондирования в кремнии РНК предсказание вторичной структуры были найдены гораздо более точным, чем структуры, полученные с помощью этих методов в одиночку.
Introduction
Для того чтобы понять функций каталитических и некодирующие РНК участвует в регуляции сплайсинга, трансляции, репликация вирусом и рак, подробные знания в области конструкций РНК требуется 1,2. К сожалению, точного предсказания укладки РНК представляет собой грозным вызов. Классическая зондирования агенты страдают от многих недостатков, таких как токсичность, неполные нуклеотидные покрытия и / или пропускной способности ограничиваясь 100-150 нуклеотидов в эксперименте. Невооруженным вторичные алгоритмы предсказания структуры являются Точно так же невыгодные, вследствие неточностями в результате их неспособность эффективно отличить среди энергетически аналогичных структур. Большие RNAs в частности, являются также часто рефрактерны к методы из 3D-решимость структуру, такие как рентгеновской кристаллографии и ядерном магнитном резонанса (ЯМР) спектроскопия, в связи с их конформационные гибкость и больших количествах из высоко чистых образцов требуются для этих методов.
HIGH-пропускная способность SHAPE решает многих из этих проблем путем предоставления эффективным, простым подход к зондировании структурах больших RNAs при напряжениях с одной нуклеотидными резолюцию. Более того, реагенты, используемые для SHAPE являются безопасными, просты в обращении и, в отличие от большинство других химический зондирование реактивов, вступают в реакцию с все четыре рибонуклеотиды. Эти реагенты также могут проникать клеточных мембран, что делает его возможный того чтобы зондировать RNAs в их в естественных условиях контекст (ы) 3. Первоначально разработанная в Недель лаборатория 4, SHAPE была использована, чтобы анализировать широкий разнообразие из RNAs, наиболее заметными из пример, являющийся определение полной вторичной структуре ~ 9 кб РНК ВИЧ-1 геном 5. Другие заметных достижений использованием SHAPE включают в себя выяснение структур инфекционных вироиды +6, человеческие длинных некодирующие РНК 7, дрожжи рибосомах +8, и riboswitches 9, а также для идентификации сайтов связывания белков в вирионом-ВИЧ, связанной с-1 РНК +3. БелыйИль оригинальные и высокой пропускной способностью вариациями из SHAPE протокол были опубликованы в другом месте 10-12, настоящее работа обеспечивает подробное описание из РНК вторичный решимость структуру путем высокой пропускной способностью SHAPE с использованием флуоресцентных олигонуклеотиды,, то компании Beckman Coulter CEQ 8 000 Genetic Analyzer, и SHAPEfinder и RNAStructure (v5.3) программное обеспечение. Ранее не публиковавшиеся технические детали и рекомендации по устранению неполадок, также включены.
Вариации из SHAPE
Суть SHAPE и его вариациях является воздействие из РНК в водном растворе до электрофильные ангидриды, который селективно ацилировать 2'-гидроксильных (2'-OH) рибозу групп, производя громоздкий аддуктов в то сайтах из модификации. Эта химическая реакция служит средством из опрашивая местных РНК структурную динамику,, как одноцепочечными нуклеотиды являются более склонными к принять конформациях способствующей электрофильной атаке этими реагентами, в то время базой парными или архитектурно Constrained нуклеотиды являются менее или нереакционноспособной +10. Сайты из образование аддукта обнаруживаются путем обратной транскрипции инициировании от флуоресцентно или радиоактивно меченные праймеры, гибридизуют со специфической сайт базирующейся на модифицированных РНК (далее «(+)" удлинения праймера реакция). Когда обратной транскриптазы (RT) не пересекают ацилированных рибонуклеотидов, пула кДНК товарной продукции, длины которых совпадают с участками модификации. Контролем ", (-)" праймер реакции удлинения использующий РНК, которая не была подвергается, чтобы реагент также выполняется так что, что преждевременная прекращение из синтез ДНК (то есть "остановками») за счет РНК структура, неспецифические прядь РНК поломки, и т.д,, может. следует отличать от ПРИОСТАНОВКА произведенный путем химической модификации. И, наконец, два дидезокси-секвенирование реакциями инициировании от теми же самыми праймерами которые используются в качестве маркеры, чтобы коррелировать реактивный нуклеотиды с РНК первичный последовательность, следующую-электрофореза.
В оригинальной применении SHAPE, то же самое 32 Р-конец-меченый праймер используется для (+), (-), и двух реакциях секвенирования. Продукты из этих реакций которые загружаются в смежный скважин в 5-8% гель сляб полиакриламид, и фракционируют помощью электрофореза в денатурирующем электрофорез в полиакриламидном геле (СТРАНИЦЕ; Рисунок 1). Количественный анализ гель изображений производится обычными SHAPE можете быть выполнены с использованием SAFA, полу-автоматизированный футпринтинга программное обеспечение для анализа 13.
В противоположность этому, высокой пропускной способностью SHAPE использует флуоресцентно меченных праймерами и автоматизированных капиллярный электрофорез. В частности, для каждого региона из РНК под следствием, собой набор из четырех ДНК праймеров, имеющих, общих последовательностях значений, но различными 5 'флуоресцентный наклейки должны храниться на быть синтезированы с или приобрели. Эти по-разному-меченых олигонуклеотидов служить к простым две SHAPE реакциям и две секвенирование реакциями, продукты, из, которые объединяли и фракционированный / обнаруженный по автоматизированный капиллярный электрофорез (СЕ). WherEAS реактивность профиля 100-150 нуклеотидов РНК могут быть получены из набора из четырех реакций с использованием оригинального подхода с высокой пропускной SHAPE дает разрешение 300-600 нуклеотидов от одного объединенного образца 3. До 8 наборов реакций может быть фракционированный одновременно, а целых 96 образцов может подготовлены для фракционирования в течение 12 последовательных пробегов CE (рис. 2). Кроме того, SHAPEfinder программное обеспечение, разработанное для обработки и анализа данных выходящих из CEQ и другие генетические анализаторы, более автоматизирована и требуют гораздо меньше вмешательства пользователя, чем SAFA 13 или другой гель-анализа пакетов.
Более продвинутые высокой пропускной методологии в последнее время появились такие, как Парс (параллельный анализ структуры РНК) 14 и Frag-Seq (фрагмент секвенирования) 15, которые используют структуру специфических ферментов, а не алкилирования реагентов в сочетании с следующего поколения секвенирования для получения ИНФОРМАЦИЯн о структуре РНК. Несмотря на привлекательность этих методов, многих ограничений, присущих нуклеазу зондирования по-прежнему остаются 16. Эти проблемы можно избежать в виде последовательности (SHAPE-Seq) 17 протокол, где следующий последовательности поколения предшествует химической модификации и обратной транскрипции РНК в манере, подобной той, что выполняется в обычной форме. Хотя эти методы могут представлять будущее определение структуры РНК, важно помнить, что в следующем последовательности поколения стоит очень дорого, и остается недоступным для многих лабораторий.
Анализ данных фигуры
Данные, полученные в генетическом анализаторе представлен в виде electropherogram, в котором интенсивность флуоресценции образца (ов), протекающей через капилляр детектор представлено в зависимости от индекса миграции времени. Этот участок имеет форму перекрытие следов, соответствующих четырем флуоресценции каналсек использоваться для обнаружения различных флуорофоров, и где каждый след состоит из пиков, соответствующих отдельным кДНК или секвенирования продуктов. Electropherogram данные экспортируются из генетического анализатора как табуляцией текстовый файл и импортировать в преобразовании ShapeFinder и анализ программного обеспечения 18.
ShapeFinder первоначально использоваться для выполнения ряда математических преобразований данных, чтобы гарантировать, что миграция раз и пиковых объемов точно отражают идентичность и количество продуктов реакции соответственно. Пики затем выровнены и объединены, а результаты представлены вместе с первичной последовательности РНК. "Реактивность профилем" для соответствующих сегментов РНК получают вычитанием контрольных значений от (+) значений, связанных с каждым РНК нуклеотидных и нормализации данных, как описано ниже. Этот профиль будет импортирован в RNAstructure (V5.3) программного обеспечения 19,20, который преобразует нормированное Вал реактивностьЕЭС в псевдо-энергии ограничения, которые включены в РНК вторичный алгоритм складной конструкции. Объединение химической зондирования и складывания алгоритмы таким образом значительно повышает точность предсказания структуры по сравнению с любым методом только 12,21. Выход RNAstructure (v5.3) включает в себя изображения наименьшей энергии вторичных структур РНК цветом с профилем SHAPE реактивность (ы), а также те же структуры в текстовой дот-скобок. Последнее может впоследствии быть экспортированы в программное обеспечение, предназначенное для графического отображения вторичной структуры РНК, таких как Варна 22 и PseudoViewer 23.
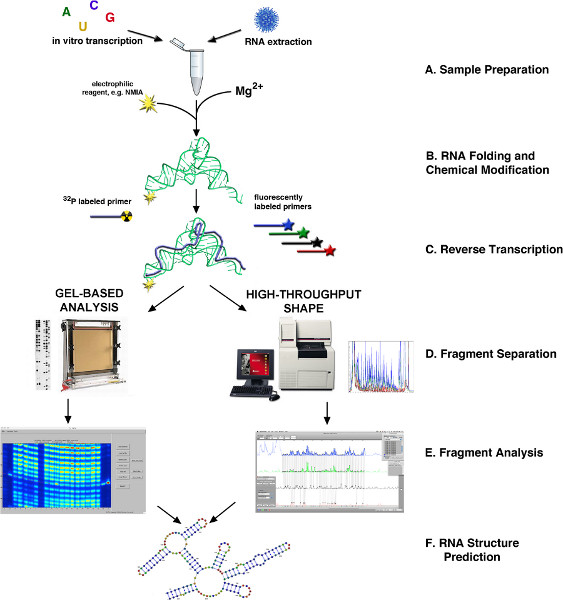
Рисунок 1. Блок-схема РНК определение структуры через SHAPE 4,10. (А) РНК мAY быть получены из биологических образцов или в пробирке транскрипции. (В) в зависимости от источника, РНК сложить или иначе обработаны и модифицированные SHAPE реагента. (C) Обратная транскрипция использованием флуоресцентно или радиоактивно меченных праймеров. (D) кДНК-продукты фракционированного либо через капиллярную или плиты на основе геля электрофореза. (E) Фрагмент анализа. (F) РНК предсказания структуры. Нажмите здесь, чтобы увеличить рисунок.
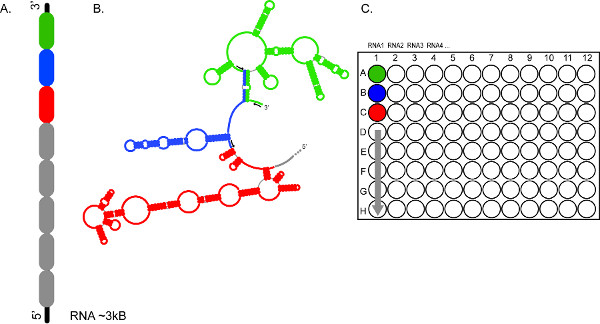
Рисунок 2. Высокой пропускной характер CE на основе форма обеспечивает быстрый анализ нескольких РНК и / или несколько сегментов одного и того же РНК. (A) </stРонг> представляет, как РНК может быть разделена на 300-600 разделов NT (с цветовым кодом в зеленый, синий и красный) (B) Разделы РНК опрашиваются независимо с использованием различных наборов флуоресцентных праймеров (черные стрелки) (C) Наборы реакций, объединяли и загружали в лунки A1, B1, C1, и т.д., соответственно, что обеспечивает полный охват ~ 3 т.п.н. RNA1. Продуктов реакции из РНК 2, 3, 4 и т.д., могут получить аналогичным образом, для фракционирования в последовательных электрофоретических работает. Нажмите здесь, чтобы увеличить рисунок.
Protocol
Representative Results
Discussion
Мы приведем здесь подробный протокол для высокопроизводительного форма, метод, который позволяет вторичное определение структуры однонуклеотидным разрешение для РНК любого размера. Кроме того, муфта экспериментальных данных SHAPE со средним алгоритмов предсказания структуры облегча?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
С. Lusvarghi, J. Sztuba-Solinska, KJ Purzycka, JW Рауш и SFJ Le Грайс поддерживаются Внутренние программы исследований Национального института рака, Национального института здоровья, США.
Materials
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5′ labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied – including any 3’terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies |
References
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 ‘-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 ‘-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2′-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues – A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).

