Ottimizzazione della crescita dei cristalli di endotiapepsina per esperimenti di cristallografia seriale
Summary
Lo scopo di questo articolo è quello di dare allo spettatore una solida comprensione di come trasformare il loro protocollo di diffusione del vapore di piccoli volumi, per la crescita di grandi cristalli proteici singoli, in un metodo di microcristallizzazione batch di grandi volumi per la cristallografia seriale.
Abstract
Qui viene presentato un protocollo per facilitare la creazione di grandi volumi (> 100 μL) di fanghi microcristallini adatti per esperimenti di cristallografia seriale sia a sincrotroni che a XFEL. Il metodo si basa sulla comprensione del diagramma di fase dei cristalli proteici e su come tale conoscenza può essere utilizzata. Il metodo è diviso in tre fasi: (1) ottimizzazione della morfologia dei cristalli, (2) transizione al batch e (3) ridimensionamento. La fase 1 prevede la ricerca di singoli cristalli ben diffusi, si spera ma non necessariamente, che si presentano in una morfologia simile a un cubo. Nella fase 2, la condizione della fase 1 è ottimizzata dal tempo di crescita dei cristalli. Questa strategia può trasformare i cristalli cresciuti dalla diffusione del vapore in batch. Una volta che la crescita dei cristalli può avvenire entro circa 24 ore, un morfogramma della miscela proteica e precipitante può essere tracciato e utilizzato come base per una strategia di ridimensionamento (Fase 3). Quando i cristalli possono essere coltivati in batch, è possibile tentare il ridimensionamento e ottimizzare la dimensione e la concentrazione del cristallo all’aumentare del volume. L’endotiapepsina è stata utilizzata come proteina dimostrativa per questo protocollo. Alcune delle decisioni presentate sono specifiche per l’endotiapepsina. Tuttavia, si spera che il modo in cui sono stati applicati ispiri un modo di pensare a questa procedura che altri possano adattare ai propri progetti.
Introduction
La cristallografia macromolecolare a temperatura ambiente (RT) è di nuovo popolare all’interno della comunità della biologia strutturale. Lo sviluppo di sorgenti luminose XFEL (X-ray Free Electron Laser) ha stimolato lo sviluppo di approcci di consegna del campione RT 1,2,3,4, e questi metodi sono stati ora applicati ai sincrotroni 5,6,7,8. Non solo i metodi RT aprono la possibilità di strageties sperimentali pompa-sonda 9,10,11,12, ma ci sono anche prove crescenti che promuovono stati conformazionali alternativi all’interno delle proteine 13,14,15,16,17.
Tuttavia, la ragione principale per cui i criometodi hanno guadagnato trazione rispetto agli approcci RT alla fine del 1990 è stato il rallentamento del danno da radiazioni da temperature dei cristalli sotto zero18. I criometodi19 hanno iniziato a consentire la raccolta di un set di dati completo da un singolo cristallo proteico. I moderni metodi RT presso XFEL e sincrotroni hanno risolto il problema del danno da radiazione monocristallina mediante lo sviluppodi strategie di rilascio rapido (> 100 Hz) 1,2,3,4. Questi metodi consentono la raccolta di un set di dati completo da migliaia di cristalli esposti individualmente. Questi approcci di rilascio RT richiedono quindi la produzione di grandi quantità di soluzioni contenenti microcristalli omogenei (> 100 μL di cristalli < 50 μm). Tuttavia, poiché i criometodi tendono a richiedere solo cristalli singoli, i metodi per creare tali fanghi microcristallini non sono attualmente onnipresenti nei laboratori di cristallografia proteica.
Ci sono esempi in letteratura di come eseguire parti della procedura di ottimizzazione della microcristallizzazione per campioni di cristallografia seriale. Qui, si dovrebbe fare una distinzione tra proteine di membrana e proteine solubili. I protocolli per ottimizzare la crescita di cristalli proteici micro-membrana cresciuti in monooleina (o qualche altro lipide), per la fase cubica lipidica (LCP), sono stati ben descritti20,21,22. Tuttavia, i metodi per la microcristallizzazione delle proteine solubili, comprese le proteine di membrana coltivate in condizioni non LCP, sono generalmente carenti. Studi precedenti si sono concentrati su parti specifiche del processo, come lo screening dei microcristalli 23,24, il miglioramento della nucleazione24 e il ridimensionamento utilizzando la diffusione a interfaccia libera 25, ma non un metodo completo.
Tuttavia, è stato recentemente descritto un metodo26 che tenta di offrire un protocollo completo. Come molti aspetti della cristallografia delle proteine, non è una novità. Molte delle idee proposte erano già state descritte da Rayment (2002)27. Il metodo mira a mostrare ai cristallografi come eseguire la conversione da un singolo cristallo coltivato utilizzando la diffusione del vapore, a una metodologia batch per far crescere migliaia di microcristalli. Il metodo si concentra sulla diffusione del vapore come punto di partenza comune, poiché il 95% di tutte le deposizioni della Protein Data Bank (PDB) proviene da cristalli cresciuti in piastre di diffusione del vapore26. La diffusione del vapore non è, tuttavia, il metodo ideale per la microcristallizzazione26, quindi viene descritta una metodologia per convertire la diffusione del vapore in cristallizzazione batch. Una volta che i cristalli possono essere coltivati in batch, i percorsi di ridimensionamento a volumi più grandi diventano più praticabili. Dati i capricci della cristallizzazione delle proteine, gli autori sottolineano che questo metodo non è sicuro. Tuttavia, il protocollo dovrebbe, almeno, fornire una panoramica dello “spazio di cristallizzazione” di una proteina.
Questo metodo si basa sul diagramma di fase di cristallizzazione delle proteine e su come la comprensione di tale diagramma possa fungere da guida durante l’ottimizzazione della microcristallizzazione. Un diagramma di fase proteica è comunemente rappresentato come un grafico x/y con concentrazioni di precipitante e proteine rispettivamente sugli assi x e y (Figura 1A). Dal punto di acqua pura (angolo in basso a sinistra – Figura 1A), la concentrazione di proteine e precipitante aumenta fino a raggiungere la linea di solubilità. La linea di solubilità segna il punto di supersaturazione (linea viola – Figura 1A). Quando una proteina è supersatura, la soluzione diventa termodinamicamente instabile e inizierà a separarsi in due fasi: “ricca di proteine” e una soluzione satura stabile. Questa separazione può avvenire ovunque oltre la linea di solubilità e la sua cinetica dipende dalle proprietà della proteina e dai componenti della soluzione.
Quando le concentrazioni di proteine e precipitanti sono troppo grandi, la proteina si decompone in modo instabile dalla soluzione e provoca precipitato amorfo (regione rosa – Figura 1A). Tuttavia, la separazione di fase ordinata può avvenire nella regione di nucleazione [vedi Garcia-Ruiz (2003) 28 per una descrizione dettagliata] e i nucleant cristallini hanno la propensione a formarsi (regione verde – Figura 1A). La nucleazione e la crescita rimuovono le proteine dalla soluzione e spostano la goccia nella regione metastabile dove la crescita può continuare fino al raggiungimento della linea di solubilità [vedi McPherson e Kuznetsov (2014) 29 per una discussione dettagliata]. Il diagramma è, per la stragrande maggioranza delle condizioni di cristallizzazione, una grossolana semplificazione30. Indipendentemente da ciò, tuttavia, il diagramma è ancora di grande utilità per i microcristallografi in quanto la mappatura del diagramma consente di determinare la linea di solubilità e la cinetica della nucleazione.
In termini di creazione di microcristalli, i due fattori durante la cristallizzazione che devono essere ottimizzati sono il numero di cristalli (Xn) e la loro dimensione media più lunga (Xs). X n sarà proporzionale al numero di eventi di nucleazione (n ) (Eq. 1).
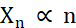 Eq. 1
Eq. 1
X s è proporzionale alla concentrazione di proteine libere al di sopra della linea di solubilità (Ps) divisa per Xn (Eq. 2).
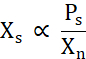 Eq. 2
Eq. 2
In una situazione perfetta, ogni evento di nucleazione produrrebbe un possibile cristallo e ognuno di questi cristalli avrebbe uguale accesso alla proteina disponibile in soluzione. La Figura 2 è una rappresentazione grafica di uno scenario ideale della relazione tra Xn e Xs. In pratica, il controllo principale che un cristallografo ha su Xn e Xs è influenzando la quantità di nucleazione o con l’aggiunta di cristalli di semi. Il microcristallografo deve giudicare come aumentare Xn in modo tale da poter creare una concentrazione di cristallo adatta e una dimensione del cristallo.
La maggior parte delle tecniche di cristallizzazione richiede un “periodo transitorio” (Figura 1B). Ad esempio, in un esperimento di diffusione del vapore, dopo aver miscelato le proteine e le soluzioni precipitanti, le concentrazioni di ciascuna cambieranno man mano che la goccia si equilibra con la soluzione del pozzo. Si spera che questi cambiamenti trasferiscano gradualmente la goccia nella zona di nucleazione dove aumenterà la propensione alla cristallizzazione. Quando i cristalli iniziano a nucleane e crescere, la quantità di proteine in soluzione inizierà a diminuire, diminuendo la probabilità di un’ulteriore nucleazione. La quantità finale di nucleazione sarà specifica per proteine e condizioni, e dipenderà anche dalla profondità di penetrazione nella zona di nucleazione. Data la limitata penetrazione della zona di nucleazione dei metodi che richiedono una fase transitoria, il livello di nucleazione sarà in definitiva limitato alla velocità di nucleazione al limite della regione di nucleazione metastabile.
A causa dell’importanza di essere in grado di migliorare il livello di nucleazione per un microcristallografo, è importante passare a una metodologia di cristallizzazione batch. Il lotto può trarre maggior vantaggio dall’intera regione di nucleazione (Figura 1C). Nei metodi batch, l’idea è quella di mescolare la proteina e il precipitante insieme in modo tale da creare una soluzione supersatura senza la necessità di cambiamenti nelle concentrazioni dei componenti. La nucleazione dovrebbe essere possibile immediatamente dopo la miscelazione. I metodi batch consentono quindi di raggiungere teoricamente l’intera zona di nucleazione. Qualsiasi aumento della cinetica di nucleazione oltre il limite metastabile-nucleazione può quindi essere utilizzato.
Se il livello basale della nucleazione dei cristalli non è sufficiente per generare un grande Xn, è possibile utilizzare metodi di microsemina. Nella micro-semina, i cristalli pre-cresciuti vengono rotti per creare un impasto di frammenti cristallini che possono fungere da impalcatura per la crescita di cristalli freschi31,32. La microsemina è stata ampiamente utilizzata nella preparazione seriale di campioni cristallografici come un modo per aumentare Xn senza la necessità di aumentare la nucleazione dei cristalli (Figura 1C).
La transizione dalla diffusione del vapore al lotto può essere visualizzata su un diagramma di fase come lo spostamento del punto di partenza sperimentale dalle regioni non supersature o metastabili alla zona di nucleazione. Questo può essere fatto aumentando le concentrazioni di proteine e/o precipitanti e/o il rapporto tra i due all’interno della goccia (Figura 1D), e osservando quali condizioni producono cristalli che appaiono rapidamente (< 24 ore)26. L’equilibrio completo della caduta di diffusione del vapore può richiedere giorni o settimane33. Pertanto, cercando condizioni che mostrano cristalli che appaiono rapidamente, è possibile trovare condizioni batch senza dover passare a formati di screening di cristallizzazione alternativi come micro-batch34,35,36,37.
Una volta trovata la zona di nucleazione, è stata trovata una condizione batch e si può creare un morfogramma – qui, un diagramma di fase approssimativo. Il morfogramma è di grande utilità quando si considera se utilizzare un protocollo seeded-batch o straight batch. Tracciando l’Xn in funzione della concentrazione proteica e del precipitante, è possibile effettuare una valutazione della cinetica di nucleazione26. Se X n rimane basso in tutta la regione di nucleazione, può essere necessario un lotto seminato per rendere Xn abbastanza grande da limitare la crescita dei cristalli. Questa valutazione è il primo passo nel processo di scalabilità a volumi più grandi (> 100 μL).
Questo metodo è stato progettato in modo tale da poter essere condotto nella maggior parte dei laboratori di cristallizzazione utilizzando apparecchiature standard di cristallizzazione a diffusione del vapore. Sono stati condotti anche molti studi che descrivono tecniche per facilitare molte parti di questo processo, se l’attrezzatura è disponibile. Questi includono, ma non sono limitati a, diffusione dinamica della luce (DLS) 25,27, imaging non lineare 20,24,25, diffrazione delle polveri 20,24,27 e microscopia elettronica 26 [vedi Cheng et al. (2020) 40 per una bella recensione].
Lo scopo di questo lavoro è quello di fornire una dimostrazione visiva del metodo per passare dalla cristallizzazione a diffusione di vapore di piccolo volume ( 100 μL). Endothiapepsin da Cryphonectria parasitica è stato usato come sistema di esempio per dimostrare questa traduzione. Il tipo di esperimento e il metodo di consegna del campione per cui sono richiesti i microcristalli influenzeranno l’output ideale di Xs 26. Per esperimenti di miscelazione che richiedono una risoluzione di tempo di millisecondi41 o ugelli virtuali gas-dinamici42, può essere auspicabile un X sfinale di < 5 μm. In questo caso, l’obiettivo era quello di produrre cristalli proteici che diffrattassero a circa 1,5 Å, per un esperimento pompa-sonda attivato da fotoni e utilizzando un approccio di consegna a bersaglio fisso.
Per fornire un’illustrazione dei requisiti del campione di un tale esperimento di cristallografia seriale utilizzando endotiapepsina, la Tabella 1 mostra i parametri sperimentali di un ipotetico esperimento. Le informazioni di esempio erano basate sul protocollo descritto di seguito. Date alcune stime prudenti sui tassi di successo e sui requisiti di raccolta dei dati, 50 mg è la stima del consumo totale del campione per l’intero esperimento.
La Figura 3 mostra un diagramma di flusso del processo di ottimizzazione completo dalla cristallizzazione iniziale della diffusione del vapore di piccoli volumi al lotto su larga scala. Per la maggior parte dei progetti di cristallografia seriale, questo protocollo inizierà nella fase 2: “transizione al batch”, poiché la proteina bersaglio sarà già stata cristallizzata. Tuttavia, il passaggio 1 è stato incluso per completezza e per ricordare ai lettori la sua importanza. Trovare una condizione che dia origine a un cristallo diffusore, singolo e grande è il miglior punto di partenza per l’ottimizzazione dei microcristalli. Nella fase 2, questa condizione può quindi essere ottimizzata dalla diffusione del vapore al lotto e può essere tracciato un morfogramma delle regioni di nucleazione e metastabilità. Una volta eseguita questa operazione, è possibile eseguire il ridimensionamento della condizione del batch a volumi più grandi nel passaggio 3. Alla fine del diagramma di flusso, un cristallografo avrà creato un protocollo batch ripetibile, di microcristallizzazione di grandi volumi (> 100 μL) per l’endotiapepsina. Questo metodo può quindi essere applicato alla loro particolare proteina di interesse.
Protocol
Representative Results
Discussion
Il metodo presentato mostra come ottimizzare la cristallizzazione dell’endotiapepsina da cristalli di grandi dimensioni (≥ dimensione più lunga di 100 μm), cresciuti in schermi a 96 pozzetti a matrice sparsa, a microcristalli, cresciuti in provette da centrifuga (volume 300 μL) tramite lotto. L’idea alla base del protocollo è che le misure adottate per ottimizzare l’endotiapepsina potrebbero essere utilizzate anche per altre proteine. In definitiva, rispondere al problema della creazione di grandi volumi (>100 μL) di microcristalli (10-20 μm) per esperimenti di cristallografia seriale a XFEL e sincrotroni.
Il protocollo divide il compito della microcristallizzazione di grandi volumi in tre fasi: (1) ottimizzazione della morfologia dei cristalli, (2) transizione al batch e (3) ridimensionamento. Nella fase 1, la gamma di forme cristalline che una proteina può creare dovrebbe essere esplorata nelle piastre di diffusione del vapore. L’obiettivo dovrebbero essere le condizioni che danno origine a singoli cristalli simili a scatole che si diffrattano alla risoluzione richiesta. Nella fase 2, le condizioni selezionate possono quindi essere trasformate dalla diffusione del vapore in batch. Qui, il criterio di ottimizzazione è il tempo di crescita dei cristalli e trovare le condizioni che danno origine a cristalli proteici entro 24 ore. Un morfogramma può anche essere tracciato dando allo sperimentatore un’idea della posizione della linea di solubilità e dei confini della zona di nucleazione. Questo morfogramma è di grande utilità nel passaggio 3, scalabilità. Il morfogramma darà un’indicazione del fatto che la nucleazione da sola possa aumentare Xn e ridurre Xs. Man mano che il volume dell’esperimento aumenta, Xn e Xs possono essere continuamente valutati come criteri chiave per il successo del ridimensionamento.
Nel caso dell’endotiapepsina, la fase 1 ha portato alla luce quella che potenzialmente era una morfologia cristallina precedentemente sconosciuta per l’endotiapepsina. Questa morfologia aveva lo stesso gruppo spaziale di quelli precedentemente riportati ma, cosa importante per la cristallografia seriale, una forma più scatolare. Anche i singoli cristalli sembravano crescere da singoli punti di nucleazione, a differenza dei ventilatori creati da altre condizioni (Figura 4). Per la condizione selezionata, la fase 2 era già parzialmente soddisfatta poiché la crescita dei cristalli si è verificata in < 24 ore. Il morfogramma indicava che sia un protocollo lineare che un protocollo seeded-batch potrebbe avere successo durante il ridimensionamento nel passaggio 3. Il ridimensionamento iniziale in batch diritto, ha creato una condizione che ha prodotto cristalli con un intervallo Xn e Xs di 3,6 ± 1,2 x 106 cristalli·mL-1 e 42 ± 4,1 μm, rispettivamente. Questi cristalli, sebbene accettabili per alcuni esperimenti di cristallografia seriale, sono stati ritenuti troppo grandi. Quindi sono state eseguite ulteriori ottimizzazioni. Il protocollo finale ha prodotto cristalli con una gamma di concentrazioni e dimensioni di 3,1 x 106 cristalli·mL-1 e 15 ± 3,9 μm, rispettivamente. Questo era più che ideale per gli esperimenti pianificati.
Il metodo si concentra sulla trasformazione di cristalli proteici “solubili” cresciuti in piastre di diffusione del vapore in lotti. La ragione di questa attenzione è che la stragrande maggioranza dei cristalli proteici solubili viene coltivata attraverso la diffusione del vapore26. Tuttavia, i concetti presentati potrebbero essere applicati anche a cristalli proteici solubili coltivati utilizzando altri metodi, come il micro-batch. I concetti possono anche essere applicabili a cristalli proteici di membrana coltivati in LCP; poiché anche questo è un processo di cristallizzazione batch.
Un aspetto chiave del protocollo è il processo di trasformazione delle condizioni dei cristalli cresciuti in piastre di diffusione del vapore in modo tale che possano essere coltivati in batch. Per questa trasformazione, il metodo utilizza il criterio proposto da Beale et al. (2019)26. I cristalli cresciuti attraverso un processo batch, anche in piastre di diffusione del vapore, si formeranno rapidamente (< 24 ore). Questo criterio è un'approssimazione basata sulla velocità di equilibrio della caduta di diffusione del vapore, ed è più vero per le condizioni precipitanti basate su PEG. Tuttavia, le condizioni di cristallizzazione conterranno un'ampia varietà di composti che influenzeranno il tempo di equilibrio. L'equilibrio delle condizioni di cristallizzazione a base di sale, ad esempio cloruro di ammonio altamente concentrato, può avvenire in 1-2 giorni. Pertanto, il criterio delle 24 ore potrebbe non essere vero per le condizioni a base di sale. Le condizioni a base di sale possono anche avere diagrammi di fase più complessi26,30 che potrebbero non essere conformi all’archetipo presentato in questo protocollo. Una riduzione del criterio temporale per le condizioni a base salina a 12 o 6 ore può essere necessaria se il ridimensionamento in volumi più grandi si rivela impossibile.
Un’altra limitazione di questo metodo è la sua apparente complessità. Il protocollo che è stato seguito per ottimizzare la microcristallizzazione dell’endotiapepsina ha effettivamente cambiato relativamente poco la condizione originale dallo schermo a matrice sparsa. Il primo colpo osservato nello schermo PACT è stato 0,1 HEPES pH 7,0, 0,2 M MgCl2 e 20% (w / v) PEG 6.000. Il tampone di cristallizzazione scalato finale era 0,1 Tris-HCl pH 7,0, 0,15 M MgCl2 e 40% (p/v) PEG 6.000. È anche molto probabile che il cambiamento del tampone da HEPES a Tris-HCl e la concentrazione di MgCl2 abbiano contribuito poco al successo del processo. Lasciando l’aumento della concentrazione di PEG 6.000 l’unica ottimizzazione, e quella che avrebbe potuto essere raggiunta molto semplicemente.
Questa valutazione, tuttavia, è anche troppo semplicistica. Non solo sconta i problemi incontrati durante il ridimensionamento (cioè l’uso di semi e tempra), ma anche il fatto che solo perché questa proteina si è rivelata semplice, non vi è alcuna garanzia che anche la prossima si rivelerà tale. I passaggi consigliati nel protocollo sono stati ideati perché ottimizzare il ridimensionamento dei volumi di cristallizzazione delle proteine può essere molto costoso dal punto di vista proteico. Nel corso dei sette studi di ridimensionamento dell’endotiapepsina che sono mostrati, sono stati consumati 100 mg di proteine. È vero che alcuni di questi passi sono stati eseguiti per mostrare le loro conseguenze alla luce di questo protocollo. Anche così, 100 mg di una proteina, più potenzialmente altri 50 mg per le proteine consumate durante un esperimento (Tabella 1), possono essere un investimento significativo in termini di tempo o denaro.
Fortunatamente, non è chiaro se questa massa di campioni richiesti sia onnipresente in tutte le proteine. L’endotiapepsina era altamente solubile e quindi richiedeva una grande concentrazione proteica per raggiungere la supersaturazione. In altri (attualmente in fase di ottimizzazione), la supersaturazione può essere raggiunta a 10 o anche 5 mg / ml. Tali variabili sono specifiche della proteina e devono essere abbracciate quando appaiono.
Altre limitazioni del metodo includono la sua dipendenza da apparecchiature complesse come robot di gestione dei liquidi per la creazione di schermi e lastre e imager per visualizzare automaticamente le lastre quando richiesto. Sono state offerte routine alternative per limitare la necessità di alcune di queste apparecchiature, ma il protocollo richiederà più tempo da seguire senza di esse. Il protocollo suggerisce anche di testare la diffrazione di cristalli ottimizzati. Per i cristallografi senza accesso regolare a un sincrotrone, questi test potrebbero rivelarsi impegnativi. I controlli in ogni fase potrebbero non essere necessari, ma questi test sono fortemente raccomandati una volta identificato un hit e pre e post-ridimensionamento. I cristalli non diffrattori in un XFEL non sono, purtroppo, un evento raro. Detto questo, è meglio sbagliare sul lato della cautela per quanto riguarda le ipotesi sulla diffrazione dei cristalli.
In definitiva, questo protocollo e i risultati presentati qui offriranno una guida, idee e un esempio a coloro che lottano con la produzione di campioni per esperimenti di cristallografia seriale. Si spera che, man mano che la cristallografia seriale viene ulteriormente sviluppata, le richieste di campioni della tecnica saranno ridotte in modo tale da ridurre la necessità di protocolli come questo. Tuttavia, anche in questo caso, le strategie qui presentate saranno comunque utili a coloro che desiderano esplorare lo spazio di cristallizzazione della loro proteina.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Questo progetto ha ricevuto finanziamenti dal programma di ricerca e innovazione Horizon 2020 dell’Unione europea nell’ambito dell’accordo di sovvenzione Marie Skłodowska-Curie n. 701647. Mille grazie per l’assistenza e il supporto degli scienziati della beamline della Swiss Light Source beamline X10SA-PXII.
Materials
| Swissci 96-well 2-Drop plates | Molecular Dimensions | MD11-002 | 96-well 2-drop crystallisation plate |
| Swissci 96-well 3-Drop plates | Molecular Dimensions | MD11-003 | 96-well 3-drop crystallisation plate |
| mosquito LCP liquid handling robot | sptlabtech | mosquito LCP | Crystallisation robot |
| ClearVue Sheets | Molecular Dimensions | MD6-015 | 96-well crystallization plate seals |
| Safe-Tube 1.5 mL | Eppendorf | 30120086 | 1.5 mL centrifuge tubes |
| Scaple | Swan and Morton | No. 3 scalple and No. 3 handle | Scalple for cutting open plate seals |
| MS 3 Vortex | IKA | 3319000 | Vortex for mixing solution and making seed stocks |
| 24-well XRL Plate | Molecular Dimensions | MD3-11 | 24-well hanging-drop plates |
| Tube revolver/rotator | Thermo Fischer Scientific | 88881001 | Tube revolver for mixing solution during scaling |
| Eppendorf Research plus pipettes | Eppendorf | Range of manual pipettes, 0.5-10, 1-20, 10-100, 100-1000 µL | |
| Eppendorf pipette tips | Eppendorf | Range of tip sizes for manual pipettes | |
| Suparen 600 | Prochem AG | Suparen 600 | Endothiapepsin solution |
| Sodium Acetate | Sigma-Aldrich | 241245-1KG | Sodium Acetate |
| Tris | Merck | 8382T014 | Tris |
| Magnesium Chloride | Sigma-Aldrich | M2670-1kg | Magnesium Chloride |
| PEG 6,000 | Sigma-Aldrich | 81255-1kg | PEG 6,000 |
| Ethelyene glycol | Sigma-Aldrich | 324558-1L | Ethelyene glycol for cyro-protecting the crystals |
| PACT Premier HT screen | Molecular Dimensions | MD1-36 | PACT Premier 96-well crystal screen |
| DOW CORNING high vacuum grease | Molecular Dimensions | MD6-02 | Grease for sealing 24-well plates |
| Hirschmann 22 x 22 mm glaser cover slides | Hirschmann | 8000104 | Cover slides for sealing 24-well sitting drop plates |
| Crystal pins | PSI | Manufactured inhouse | Thin-film supports for micro-crystals. |
| 1-1.3 mm SiLibeads Type S | Faust | 6239547 | Glass beads for making mico-seed stocks |
| Macbook Pro | Apple | Macbook Pro | Computer for performing data analysis |
| CCP4 software suite | CCP4 | Diffraction pattern data processing software | |
| Excel | Microsoft | Microsoft Office | Plotting tool for phase diagram |
| Hausser Scientific Bright-Line counting chamber | Thermo Fischer Scientific | 02-671-51B | Tool to calculate crystal concentration |
| PACT Premier | Molecular Dimensions | MD1-29-ECO | Sparse-matrix crystallization screen |
| Rock Imager | Formulatrix | Rock Imager | Temperature controlled crystal plate storage and imager |
| Rock MakerWeb | Formulatrix | Rock MakerWeb | Crystal plate creation and image storage stoftware |
| Formulator | Formulatrix | Formulator | 96-well crystal screen creation liquid handling robot |
| Leica MZ16 Microscope | Leica | Leica MZ16 | Light microscope |
| LAS V4.6 | Leica | LAS V4.6 | Software for Leica microscopes |
| Spectra/Por 3.5 kDa dialysis tubing | Spectrumlabs | Spectra/Por 3 Dialysis Membrane | 3.5 kDa dialysis membrane |
| Dialysis tubing closures | Spectrumlabs | Spectra/Por 3 Duniversal Closures | Clips to seal the dialysis tubing ends |
| Amicon 10 kDa centrifugal concentrator | Merck-Millipore | Amicon Ultra-15 10 kDa centrifugal concentrator | 10 kDa centrifugal filter |
| 5810 R swing bucket centrifuge | Eppendorf | 5810 R Centrifuge | Swing bucket centrifuge |
References
- DePonte, D. P., et al. Gas dynamic virtual nozzle for generation of microscopic droplet streams. Journal of Physics D: Applied Physics. 41 (19), 195505 (2008).
- Hunter, M. S., et al. Fixed-target protein serial microcrystallography with an x-ray free electron laser. Scientific Reports. 4 (1), 6026 (2014).
- Weierstall, U., et al. Lipidic cubic phase injector facilitates membrane protein serial femtosecond crystallography. Nature Communications. 5 (1), 1-6 (2014).
- Roessler, C. G. G., et al. Acoustic Injectors for Drop-On-Demand Serial Femtosecond Crystallography. Structure. 24 (4), 631-640 (2016).
- Sherrell, D. A., et al. A modular and compact portable mini-endstation for high-precision, high-speed fixed target serial crystallography at FEL and synchrotron sources. Journal of Synchrotron Radiation. 22 (6), 1372-1378 (2015).
- Roedig, P., et al. A micro-patterned silicon chip as sample holder for macromolecular crystallography experiments with minimal background scattering. Scientific Reports. 5 (1), 1-11 (2015).
- Botha, S., et al. Room-temperature serial crystallography at synchrotron X-ray sources using slowly flowing free-standing high-viscosity microstreams. Acta Crystallographica Section D Biological Crystallography. 71 (2), 387-397 (2015).
- Weinert, T., et al. Serial millisecond crystallography for routine room-temperature structure determination at synchrotrons. Nature Communications. 8 (1), 542 (2017).
- Tenboer, J., et al. Time-resolved serial crystallography captures high-resolution intermediates of photoactive yellow protein. Science. 346 (6214), 1242-1246 (2014).
- Nango, E., et al. A three-dimensionalmovie of structural changes in bacteriorhodopsin. Science. 354 (6319), 1552-1557 (2016).
- Suga, M., et al. Light-induced structural changes and the site of O=O bond formation in PSII caught by XFEL. Nature. 543 (7643), 131-135 (2017).
- Mehrabi, P., et al. Liquid application method for time-resolved analyses by serial synchrotron crystallography. Nature Methods. 16 (10), 979-982 (2019).
- Halle, B. Biomolecular cryocrystallography: structural changes during flash-cooling. Proceedings of the National Academy of Sciences of the United States of America. 101 (14), 4793-4798 (2004).
- Fraser, J. S., et al. Hidden alternative structures of proline isomerase essential for catalysis. Nature. 462 (7273), 669-673 (2009).
- Fenwick, R. B., van den Bedem, H., Fraser, J. S., Wright, P. E. Integrated description of protein dynamics from room-temperature X-ray crystallography and NMR. Proceedings of the National Academy of Sciences of the United States of America. 111 (4), 445-454 (2014).
- Keedy, D. A., et al. Mapping the conformational landscape of a dynamic enzyme by multitemperature and XFEL crystallography. Elife. 4, (2015).
- Thomaston, J. L., et al. XFEL structures of the influenza M2 proton channel: Room temperature water networks and insights into proton conduction. Proceedings of the National Academy of Sciences of the United States of America. 114 (51), 13357-13362 (2017).
- Haas, D. J., Rossmann, M. G. Crystallographic studies on lactate dehydrogenase at -75 °C. Acta Crystallographica Section B Structural Crystallography and Crystal Chemistry. 26 (7), 998-1004 (1970).
- Hope, H. Cryocrystallography of biological macromolecules: a generally applicable method. Acta Crystallographica Section B Structural Science. 44 (1), 22-26 (1988).
- Wu, W., et al. Batch crystallization of rhodopsin for structural dynamics using an X-ray free-electron laser. Acta Crystallographica Section:F Structural Biology Communications. 71 (7), 856-860 (2015).
- Ishchenko, A., Cherezov, V., Liu, W. Preparation and delivery of protein microcrystals in lipidic cubic phase for serial femtosecond crystallography. Journal of Visualized Experiments. (115), e54463 (2016).
- Andersson, R., et al. Well-based crystallization of lipidic cubic phase microcrystals for serial X-ray crystallography experiments. Acta Crystallographica Section D: Structural Biology. 75 (10), 937-946 (2019).
- Luft, J. R., et al. The detection and subsequent volume optimization of biological nanocrystals. Structural Dynamics. 2 (4), 041710 (2015).
- Lee, D. B., et al. Supersaturation-controlled microcrystallization and visualization analysis for serial femtosecond crystallography. Scientific Reports. 8 (1), 1-10 (2018).
- Kupitz, C., et al. Microcrystallization techniques for serial femtosecond crystallography using photosystem II from Thermosynechococcus elongatus as a model system. Philosophical transactions of the Royal Society of London. Series B, Biological Sciences. 369 (1647), 20130316 (2014).
- Beale, J. H., et al. Successful sample preparation for serial crystallography experiments. Journal of Applied Crystallography. 52, 1385-1396 (2019).
- Rayment, I. Small-scale batch crystallization of proteins revisited: An underutilized way to grow large protein crystals. Structure. 10 (2), 147-151 (2002).
- García-Ruiz, J. M. Nucleation of protein crystals. Journal of Structural Biology. 142 (1), 22-31 (2003).
- McPherson, A., Kuznetsov, Y. G. Mechanisms, kinetics, impurities and defects: Consequences in macromolecular crystallization. Acta Crystallographica Section F:Structural Biology Communications. 70 (4), 384-403 (2014).
- Rupp, B. Origin and use of crystallization phase diagrams. Acta crystallographica. Section F, Structural biology communications. 71, 247-260 (2015).
- Luft, J. R., DeTitta, G. T. A method to produce microseed stock for use in the crystallization of biological macromolecules. Acta Crystallographica Section D Biological Crystallography. 55 (5), 988-993 (1999).
- Ireton, G. C., Stoddard, B. L. Microseed matrix screening to improve crystals of yeast cytosine deaminase. Acta crystallographica. Section D, Biological crystallography. 60 (3), 601-605 (2004).
- Forsythe, E. L., Maxwell, D. L., Pusey, M. Vapor diffusion, nucleation rates and the reservoir to crystallization volume ratio. Acta Crystallographica Section D Biological Crystallography. 58 (10), 1601-1605 (2002).
- Chayen, N. E., Shaw Stewart, P. D., Maeder, D. L., Blow, D. M. IUCr An automated system for micro-batch protein crystallization and screening. Journal of Applied Crystallography. 23 (4), 297-302 (1990).
- Chayen, N. E., Shaw Stewart, P. D., Blow, D. M. Microbatch crystallization under oil – a new technique allowing many small-volume crystallization trials. Journal of Crystal Growth. 122 (1-4), 176-180 (1992).
- Chayen, N. E. Comparative studies of protein crystallization by vapour-diffusion and microbatch techniques. Acta Crystallographica Section D: Biological Crystallography. 54 (1), 8-15 (1998).
- D’Arcy, A., Mac Sweeney, A., Stihle, M., Haber, A. The advantages of using a modified microbatch method for rapid screening of protein crystallization conditions. Acta Crystallographica – Section D Biological Crystallography. 59 (2), 396-399 (2003).
- Darmanin, C., et al. Protein crystal screening and characterization for serial femtosecond nanocrystallography. Scientific Reports. 6, 25345 (2016).
- Gati, C., et al. Atomic structure of granulin determined from native nanocrystalline granulovirus using an X-ray free-electron laser. Proceedings of the National Academy of Sciences of the United States of America. 114 (9), 2247-2252 (2017).
- Cheng, R. K. Y. Towards an optimal sample delivery method for serial crystallography at XFEL. Crystals. 10 (3), 215 (2020).
- Schmidt, M. Mix and Inject: Reaction Initiation by Diffusion for Time-Resolved Macromolecular Crystallography. Advances in Condensed Matter Physics. 2013, 1-10 (2013).
- Oberthuer, D., et al. Double-flow focused liquid injector for efficient serial femtosecond crystallography. Scientific Reports. 7 (1), 44628 (2017).
- McPherson, A., Gavira, J. A. Introduction to protein crystallization. Acta crystallographica. Section F, Structural biology communications. 70, 2-20 (2014).
- Yaoi, M., et al. Effect of stirring method on protein crystallization. Japanese Journal of Applied Physics, Part 2: Letters. 43 (10), 1318 (2004).
- Castro, F., Ferreira, A., Teixeira, J. J., Rocha, F. Influence of Mixing Intensity on Lysozyme Crystallization in a Meso Oscillatory Flow Reactor. Crystal Growth & Design. 18 (10), 5940-5946 (2018).
- Moews, P. C., Bunn, C. W. An X-ray crystallographic study of the rennin-like enzyme of Endothia parasitica. Journal of Molecular Biology. 54 (2), 395-397 (1970).
- Newman, J., et al. Towards rationalization of crystallization screening for small- to medium-sized academic laboratories: the PACT/JCSG+ strategy. Acta crystallographica. Section D, Biological. 61, 1426-1431 (2005).
- Ebrahim, A., et al. Resolving polymorphs and radiation-driven effects in microcrystals using fixed-target serial synchrotron crystallography. Acta Crystallographica Section D Structural Biology. 75, 151-159 (2019).
- Davy, B., et al. Reducing sample consumption for serial crystallography using acoustic drop ejection. Journal of Synchrotron Radiation. 26 (5), (2019).

