Hoogwaardige voorbereiding van hersen- en beenmergkernen voor multioomtesten met één kern
Summary
Het succes van single-cell/single-nuclei transcriptomics en multi-omics hangt grotendeels af van de kwaliteit van cellen/kernen. Daarom moet het isoleren van cellen/kernen uit weefsel en hun zuivering sterk gestandaardiseerd zijn. Dit protocol beschrijft de bereiding van kernen uit de hersenen en het beenmerg voor stroomafwaartse single-nuclei multioomanalyse.
Abstract
Single-cell analyse is de voorkeursbenadering geworden voor het ontrafelen van de complexiteit van biologische processen die het beoordelen van de variabiliteit van individuele cellulaire reacties op behandeling of infectie met single-cell resolutie vereisen.
In de afgelopen 10 jaar zijn er veel technieken voor eencellige moleculaire profilering ontwikkeld en zijn er verschillende specifieke technologieën op de markt gebracht. De 10X Genomics druppelgebaseerde single-cell profiling is een wijdverbreide technologie die kant-en-klare reagentia biedt voor transcriptomische en multi-omic single-cell profilering. De technologie omvat workflows voor single-cell en single-nuclei RNA-sequencing (respectievelijk scRNA-Seq en snRNA-Seq), scATAC-Seq, single-cell immuunprofilering (BCR/TCR-sequencing) en multioom. De laatste combineert transcriptionele (scRNA-Seq) en epigenetische informatie (scATAC-Seq) afkomstig van dezelfde cel.
De kwaliteit (levensvatbaarheid, integriteit, zuiverheid) van eencellige of eenkernige suspensies die uit weefsels zijn geïsoleerd en met een van deze benaderingen worden geanalyseerd, is van cruciaal belang voor het genereren van gegevens van hoge kwaliteit. Daarom moeten de protocollen voor de bereiding van monsters worden aangepast aan de specifieke kenmerken van elk biologisch weefsel en moeten ervoor zorgen dat hoogwaardige cel- en kernsuspensies worden gegenereerd.
Dit artikel beschrijft twee protocollen voor het voorbereiden van hersen- en beenmergmonsters voor de stroomafwaartse multioom 10X Genomics-pijplijn. De protocollen worden stapsgewijs uitgevoerd en hebben betrekking op weefseldissociatie, celsortering, kernisolatie en kwaliteitscontrole van bereide kernsuspensie die wordt gebruikt als uitgangsmateriaal voor celpartitionering en barcodering, bibliotheekvoorbereiding en sequencing. Deze gestandaardiseerde protocollen produceren kernbibliotheken van hoge kwaliteit en robuuste en betrouwbare gegevens.
Introduction
Single-cell technieken zijn al jaren de gouden standaard voor de analyse van biologische processen. Ze waren aanvankelijk beperkt tot fenotypering van eencellige cellen door middel van microscopie, flowcytometrie en soortgelijke tests. Een doorbraak in single-cell analyse kwam met de ontwikkeling van benaderingen voor single-cell moleculaire profilering, in het bijzonder single-cell RNA sequencing (scRNA-Seq) die het mogelijk maakte om het volledige transcriptoom van individuele cellen te karakteriseren. ScRNA-Seq is zeer krachtig en genereert informatie over de transcriptionele status van een cel in een specifieke toestand en op een specifiek tijdstip. Het geeft echter geen zicht op de genregulatie die transcriptie aandrijft, of op de moleculaire modificaties die in de loop van de tijd optreden. Om deze beperking te overwinnen, zijn er veel inspanningen geleverd in de ontwikkeling van single-cell multi-omics-assays die de analyse van meerdere factoren en processen uit dezelfde cel mogelijk maken 1,2,3,4. De eerste succesvolle meting van twee modaliteiten binnen afzonderlijke cellen kwam door het koppelen van multiplex oppervlakte-eiwitexpressiepatronen met het volledige transcriptoom van individuele cellen in de CITE-Seq-benadering5. Recentere evoluties combineren genexpressie met de toegankelijkheid van chromatine (Assay for Transposase-Accessible Chromatin using sequencing, ATAC-Seq), waardoor tegelijkertijd transcriptomische en epigenomische modaliteiten in dezelfde cellen worden vastgelegd (bijv. sci-CAR)6. De eerste commerciële oplossingen die het mogelijk maakten om transcriptomics te associëren met celfenotype of met epigenetische veranderingen van dezelfde cel, kwamen van 10X Genomics.
Experimenten voor eencellige moleculaire profilering omvatten de volgende stappen: (1) weefseldissociatie of bereiding van eencellige suspensies; (2) celzuivering en/of isolatie van kernen; (3) partitionering en streepjescodering; (4) bibliotheekbouw en kwaliteitscontrole; (5) sequencing van de volgende generatie; (6) gegevensanalyse. Hoewel de stappen (3)-(6) aanzienlijk kunnen variëren, afhankelijk van de gebruikte technologie, zijn de eerste stappen over het algemeen gemeenschappelijk voor alle stappen. De kwaliteit van de bereide cel-/kernsuspensie zal de algehele uitkomst van het experiment bepalen. Afhankelijk van het type weefsel kan het verkrijgen van hoogwaardige eencellige/kernsuspensies een uitdaging zijn. De bijzonderheden van sommige weefsels, zoals het hart, de spieren, de hersenen, de longen, de darmen en andere, vereisen methoden voor weefselverstoring en isolatie van kernen die zijn aangepast aan elk type monster om de productie van hoogwaardige kernen voor moleculaire analyse te garanderen 7,8,9,10 . De weefseldisruptiemethoden en dissociatieprotocollen kunnen mechanisch, enzymatisch (bijv. een mix van collagenasen en DNase) of een combinatie van beide zijn, en kunnen handmatig of door instrumenten worden uitgevoerd (bijv. Qiagen DSC-400, gentleMACS).
Single-cell technieken zijn een instrument bij uitstek geworden voor biomedisch onderzoek. In de neurobiologie vereisen de celdiversiteit in de hersenen en de complexiteit van hun functies een analyse met hoge resolutie en hoge doorvoer voor visualisatie van zeldzame celpopulaties en voor het beoordelen van hun heterogeniteit 11,12,13,14. Het koppelen van cellulaire identiteit en genregulatiemechanismen van individuele cellen geeft inzicht in de ontwikkeling en fysiologie van de hersenen. Een ander voorbeeld zijn studies naar immuunrespons in de context van infectieuze, auto-immuun- of kankerziekten, die sterk afhankelijk zijn van eencellige analyses. De heterogeniteit van subsets van immuuncellen en de complexiteit van hun activiteit en interacties met andere celtypen vereisen een oplossing van één cel bij het ontcijferen van de mechanismen die ten grondslag liggen aan de immuunrespons. De immuuncellen zijn afkomstig uit het beenmerg, waar hematopoëtische voorlopercellen zijn samengesteld uit geleidelijk differentiërende cellen die celoppervlaktemarkers verwerven en verliezen tijdens een stapsgewijs proces voordat ze het beenmerg verlaten naar huis in de periferie. Analyse van één cel maakt een minutieuze karakterisering van cellulaire ontwikkelingsstadia mogelijk. Het kan worden bereikt door middel van fenotypering van één cel, conventioneel uitgevoerd door flowcytometrie met meerdere parameters. Er is echter aangetoond dat eencellige transcriptomische handtekeningen een nauwkeurigere identificatie van subtypes van voorlopercellen onthullen, aangezien deze cellen zijn verdeeld in clusters die in elkaar vallen en daarom verkeerd kunnen worden geïdentificeerd bij gebruik van een grove celoppervlaktemarkerbenadering15. Een toenemend aantal studies onthult de epigenetische modificaties die hematopoëtische stam- en voorlopercellen (HSPC’s) kunnen verwerven door blootstelling aan verschillende middelen, wat leidt tot een aanzienlijke impact op de responsiviteit van het immuunsysteem op lange termijn 16,17,18,19. De nieuwe multi-omics-technologieën maken het mogelijk om deze processen te bestuderen met een resolutie van één cel.
Er zijn veel protocollen voor cel- en kernisolatie beschreven voor hersenen 11,20,21,22 en beenmergmonsters23,24. Om vertekening als gevolg van experimentele variabiliteit te minimaliseren, is het noodzakelijk om geoptimaliseerde single-nuclei voorbereidingsprotocollen voor gezamenlijke single-cell transcriptomische en epigenomische sequencing te valideren, waardoor de reproduceerbaarheid van single-cell multiomische assays wordt gegarandeerd.
Hier worden twee robuuste protocollen beschreven voor de voorbereiding van kernen uit (1) vers ingevroren hersenweefsel en (2) verse beenmerg-HSPC’s voor stroomafwaartse single-cell Multioom-analyse (Figuur 1).
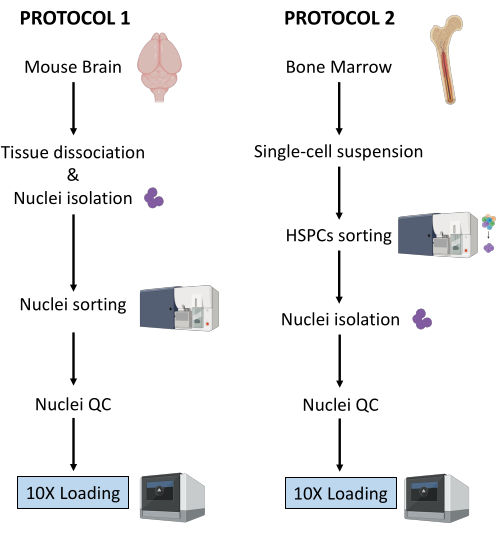
Figuur 1: Schematische weergave van protocollen voor het isoleren van kernen uit vers ingevroren hersen- en beenmergweefsel. Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
De bereiding van hoogwaardige cel- of kernsuspensie is van cruciaal belang voor het succes van single-cell of single-nuclei RNA-Seq en single-cell multi-omic analyses 29,30,31. Hier hebben we protocollen beschreven voor monstervoorbereiding en kernisolatie voor multioomtesten van twee soorten weefsel: hersenen en beenmerg.
Het hersenprotocol dat in dit artikel wordt beschreven, maakt het mogelijk om kernen van hoge kwaliteit te herstellen uit vers ingevroren hersenweefsel. Het omvat de volgende stappen: verstoring van bevroren weefsel, isolatie van kernen, zuivering van kernen en kwaliteitscontrole van het bereide materiaal. Het hersenweefsel is samengesteld uit veel verschillende celtypen en de procedure van weefseldissociatie en kernisolatie moet de verhoudingen van celpopulaties zoals aanwezig in het oorspronkelijke weefsel behouden. Hier werden de samenstelling van de lysisbuffer en de incubatietijd geoptimaliseerd om een volledige en zachte lysis mogelijk te maken van alle celpopulaties waaruit het weefsel bestaat.
Het HSPC-protocol voor beenmerg is enigszins anders, omdat er aan het begin van het experiment een extra stap nodig is om de celpopulatie van belang te isoleren van een heterogene cellulaire suspensie. Na het verzamelen van vers weefsel worden rode bloedcellen gelyseerd en wordt het monster verrijkt voor de celsubset van belang. De beoogde cellen worden gelyseerd, de kernen worden geïsoleerd en de kwaliteit van het bereide materiaal wordt gecontroleerd.
10X Genomics biedt verschillende protocollen die zijn gevalideerd voor het isoleren van kernen in tal van verschillende weefsels32,33. Het bedrijf brengt ook een nuclei-isolatiekit op de markt met een eenvoudige pijplijn voor het isoleren van kernen uit gevalideerde weefsels34. Deze protocollen moeten echter extra worden geoptimaliseerd om de bijzonderheden van bepaalde monsters aan te passen. Een voorbeeld zijn de monsters die moeten worden gewerkt met een lage celinput. Voor deze monsters zijn de meest uitdagende stappen centrifugaties die voldoende streng moeten zijn om het monster te reinigen en zacht genoeg om verlies van cellen/kernen te voorkomen. Met het hier beschreven protocol hebben we het 10X Genomics Shown Protocol – Nuclei Isolation for Single Cell Multiome ATAC + GEX Sequencing (CG000365 – Rev C)27 aangepast om een fijne balans te vinden tussen deze twee vereisten. Zoals aangetoond in het voorbeeld van de bereiding van kernen uit gesorteerde HSPC’s, hebben we de terugwinning van kernen verbeterd zonder dat dit gevolgen heeft voor de kwaliteit van het monster.
Een extra uitdaging is de stap van lysis van gezuiverde cellen voor het isoleren van kernen. Zwaardere lysisomstandigheden en langere incubatietijden kunnen kernen beschadigen en daardoor de kwaliteit van sequentiegegevens beïnvloeden. Figuur 5 toont representatieve beeldvorming van kernen uit beenmergmonsters op verschillende incubatietijden met lysisbuffer en illustreert hoe verschillend de toestand van de kernen kan zijn, afhankelijk van de cellyse. In het voorbeeld van de HSPC’s hebben we 3 min lysis geïdentificeerd als de aandoening die resulteert in het hoogste aandeel gezond ogende, intacte kernen en het laagste aandeel beschadigde kernen. De lysis-incubatietijden moeten voor elk nieuw type monster worden geoptimaliseerd.
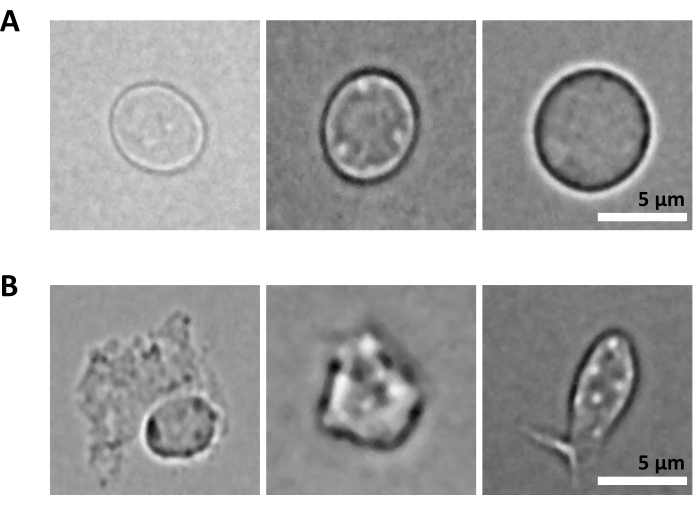
Figuur 5: Kwaliteitscontrole van de kernen door middel van microscopie. Getoond worden representatieve helderveldbeelden van geïsoleerde kernen uit het beenmerg van muizen met (A) intacte en (B) beschadigde kernen. Schaal staaf 5 μm. De foto’s zijn gemaakt met een omgekeerde microscoop met een 40x ELWD NA 0.60 objectief en 1.5x digitale zoom. Klik hier om een grotere versie van deze figuur te bekijken.
Beide protocollen die in dit werk worden beschreven, zijn gebaseerd op het zuiveren van gerichte cellen of kernen door FACS-instrumenten met een hoge doorvoer. Deze stap is van cruciaal belang voor protocollen voor de bereiding van eencellige cellen/kernen, waarbij zeldzame subgroepen van cellen moeten worden geïsoleerd uit heterogene suspensies. In deze, zoals in het hier getoonde voorbeeld voor het sorteren van HSPC’s, kan een hoogdimensionaal flowcytometriepaneel nodig zijn om “gating” op de celpopulatie van belang mogelijk te maken. De sortering is extreem snel en nauwkeurig, wat leidt tot een zuiverheid van meer dan 95% van de gesorteerde celsubsets. Deze benadering stelt de cellulaire suspensie bloot aan een druk tot 70 psi en kan daarom beperkend zijn voor het sorteren van fragiele cellen (bijv. dendritische cellen, neutrofielen), omdat het de breuk van hun celmembraan kan veroorzaken. In deze gevallen moeten alternatieve oplossingen worden gekozen voor cellulaire zuivering, waaronder magnetische sortering, toepassing van instrumenten van de nieuwe generatie (bijv. CellenOne, Cellenion; MACSQuant Tyto, Miltenyi)35,36, of op druppels gebaseerde systemen (bijv. ODIN, Sensific)37. Niettemin is de trage sorteersnelheid van deze technologieën, met celsortering die uren in plaats van minuten duurt, een sterke beperkende factor voor de toepassing van deze benaderingen bij de voorbereiding van levensvatbare cellen voor Multiome en andere eencellige toepassingen op basis van analyse van grote celaantallen.
Voor de zuivering van kernen die uit het weefsel zijn geïsoleerd, is FACS de voorkeursmethode vanwege de doorvoer en de zuiverheid van het geïsoleerde materiaal. Kernen zijn niet gevoelig voor druk en gefilterde weefselisolaten kunnen gemakkelijk worden gezuiverd via de celsorteerder. Als het laboratorium niet is uitgerust met een FACS-instrument, bestaan er andere alternatieven, iets minder efficiënt maar voldoende goed. Voorbeelden hiervan zijn ultracentrifugatie of het gebruik van kleine apparatuur zoals MARS (Applied Cell) die deeltjes scheidt op basis van hun verschil in grootte, met behulp van akoestische golven; CURIOX laminaire ring die gebruik maakt van hydrofobe eigenschappen van cel-/kernsuspensies; of LEVITAS bio die vertrouwt op fysische eigenschappen van cellen (levitatie) om ze van het puin te scheiden.
Hier beschrijven we protocollen om een hoog aantal kernen en de beste zuiverheid voor het stroomafwaartse Multiome-protocol te verkrijgen. FACS-sortering en herhaalde centrifugatiestappen resulteren in een aanzienlijk verlies van het oorspronkelijke materiaal. Om deze reden is in het protocol voor de voorbereiding van kernen uit de hersenen dat we hier beschrijven voldoende overvloedig uitgangsmateriaal nodig om te resulteren in de verzameling van ten minste 500.000 kernen na de FACS-sortering. Alternatieve protocollen moeten worden toegepast als aan dit criterium niet kan worden voldaan. Bij het werken met zeldzame celpopulaties of kleine weefselcoupes kan de beschikbare hoeveelheid uitgangsmateriaal een beperkende factor zijn. Om dit probleem aan te pakken, is het mogelijk om het herstel van kernen te verbeteren door (a) het lysisvolume te verminderen, (b) het wasvolume te verminderen, (c) een enkele wasbeurt met verlengde centrifugatietijd te gebruiken om te proberen het herstel te verbeteren, zoals aangegeven in 10X Genomics-protocollen voor isolatie van kernen met een lage celinput. Voor multiomische analyse van materiaal met een laag gehalte is het de moeite waard om plaatgebaseerde toepassingen te overwegen, zoals scNMT, SNARE-seq en Paired-seq38 , waarvoor veel minder invoermonsters nodig zijn.
Samengevat hebben we twee robuuste protocollen beschreven voor de voorbereiding van kernen uit de hersenen en de HSPC’s van het beenmerg voor stroomafwaartse Multioomanalyse. Deze protocollen zijn toepasbaar in elk wetenschappelijk project dat hoogwaardige suspensies van één kern uit deze twee soorten weefsel vereist, ongeacht de wetenschappelijke vraag die wordt gesteld. Onze groep heeft het protocol voor het isoleren van hersenkernen toegepast in studies van hersenontwikkeling na inactivatie van verschillende gerichte genen en in studies van immuunrespons in de context van neurologische aandoeningen. We gebruiken het protocol voor het isoleren van beenmergkernen voor het ontcijferen van de deelname van verschillende hematopoëtische subpopulaties aan de opbouw van het immuunsysteem.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ana Jeemin Choi werd ondersteund door een stipendium van de Pasteur – Paris University (PPU) International Ph.D. Programma.
Materials
| 18 G x 1 ½ (1.2 mm x 38 mm) Agani needles | Terumo | AN*1838S1 | |
| 15 mL tubes | Falcon | 352097 | |
| 5 mL round bottom FACS tube with cell strainer cap 35 µm | falcon | 352235 | |
| 50 mL tubes | Falcon | 352070 | |
| 7-AAD | BD pharmagen | 559925 | |
| ACK Lysing Buffer | Gibco | A10492-01 | |
| APC anti-mouse CD117 (c-Kit) | BioLegend | 105812 | Clone: 2B8 |
| APC/Cyanine 7 anti-mouse CD16/32 (FcγR) | BioLegend | 101328 | Clone: 93 |
| BD FACSAria III | BD Biosciences | non-applicable | |
| BD FACSDiva Software v8.0.1 | BD Biosciences | non-applicable | |
| Bovine Serum Albumin stock solution 10% | Miltenyi Biotec | 130-091-376 | |
| Cell staining buffer | Biolegend | 420201 | |
| CFI Suprplan Fluor ELWD 40XC ON 0.6 | Nikon | non-applicable | |
| CMOS camera Prime 95B 25 mm | Photometrix | non-applicable | |
| Countess II FL Automated Cell Counter | Invitrogen | AMQAF1000 | |
| Countess cell counting chamber slide | Invitrogen | C10283 | |
| Coverglass 24 mm x 24 mm 0.13-0.17 mm | Brand | BR470819 | |
| Digitonine 5% | Invitrogen | BN2006 | |
| Disposable Scalpels | Swann-Morton | 0508 | |
| DMEM (1x) + GlutaMAX-I | Gibco | 31966-021 | |
| DPBS (10x) | Gibco | 14200-067 | |
| DTT | Sigma aldrich | 646563 | |
| Epifluorescence inverted microscope Nikon Ti2 -E | Nikon | non-applicable | |
| Eppendorf Safe-Lock Tubes 0.5 mL | Eppendorf | 30123603 | |
| Ethanol 70% | VWR | 83801.290 | |
| FITC anti-mouse CD34 | Invitrogen | 11-0341-85 | Clone: RAM34 |
| Forceps for dissection | FST | 11152-10 | |
| Heat-inactivated Fetal Bovine Serum (FBS) | Gibco | 11533387 | |
| Dounce Homogeniser 2 mL | Bellco glass | 1984-10002 | Pestle “A” Large Clearance: .0030-.0050″ and Pestle “B” Small Clearance: .0005-.0025″ |
| LIVE/DEAD fixable aqua dead cell stain kit | Invitrogen | L34957 | |
| Magnesium chloride solution 1 M | Sigma aldrich | M1028 | |
| Microcentrifuge | Eppendorf | 5424R | |
| Mounting medium Fluoromount-G | invitrogen | 00-4958-02 | |
| Nonidet P40 substitute | Sigma aldrich | 74385 | |
| Nuclease free water | ThermoFischer | AM9932 | |
| Nuclei buffer 20x | 10X Genomics | 2000153/2000207 | |
| Nuclei isolation kit EZ prep | Sigma Aldrich | NUC-101 | |
| OneComp eBeads compensation beads | Invitrogen | 01-1111-41 | |
| Pacific Blue anti-mouse lineage cocktail (including anti-mouse CD3, Ly-6G/Ly-6C, CD11b, CD45R/B220, TER-119) |
BioLegend | 133310 | Clones (in the same order as the antibodies listed): 17A2, RB6-8C5, M1/70, RA3-6B2, Ter-119 |
| PCR Tube Strips 0.2 mL | Eppendorf | 951010022 | |
| PE anti-mouse Ly-6A/E (Sca-1) | BioLegend | 122507 | Clone: E13-161.7 |
| Petri dish 100 mm x 20 mm OPTILUX | Falcon | 353003 | |
| Ply-L-lysine 0.01% sterile-filtered suitable for cell culture | Sigma | P4707 | |
| Printed microscope slides 8 well 6 mm numbered | Epredia | ER-301B-CE24 | |
| Protein LoBind Tubes 1.5 mL | Eppendorf | 30108116 | |
| Recombinant Rnase inhibitor 5000 U | Takara | 2313A | |
| Scissors for dissection | FST | 14090-09 | |
| Sodium chloride solution 5 M | Sigma aldrich | 59222C | |
| Syringe filters, PES, 0.2 µm | Fisher Scientific | 15206869 | |
| Transparent nail polish | any | non-applicable | |
| Trizma Hydrochloride solution pH 7.4 | Sigma aldrich | T2194 | |
| Trypan Blue 0.4% | gibco | 15250061 | |
| Tween 20 | Biorad | 1662404 | |
| UltraPure Distilated Water Dnase/Rnase Free | Invitrogen | 10977-035 |
References
- Clark, S. J., et al. scNMT-seq enables joint profiling of chromatin accessibility DNA methylation and transcription in single cells. Nature Communications. 9 (1), 781 (2018).
- Lee, J., Hyeon, D. Y., Hwang, D. Single-cell multiomics: technologies and data analysis methods. Experimental & Molecular Medicine. 52 (9), 1428-1442 (2020).
- Cerrizuela, S., et al. High-throughput scNMT protocol for multiomics profiling of single cells from mouse brain and pancreatic organoids. STAR Protocols. 3 (3), 101555 (2022).
- Dimitriu, M. A., Lazar-Contes, I., Roszkowski, M., Mansuy, I. M. Single-cell multiomics techniques: From conception to applications. Frontiers in Cell and Developmental Biology. 10, 854317 (2022).
- Stoeckius, M., et al. Simultaneous epitope and transcriptome measurement in single cells. Nature Methods. 14 (9), 865-868 (2017).
- Cao, J., et al. . Joint profiling of chromatin accessibility and gene expression in thousands of single cells. 361 (6409), 1380-1385 (2018).
- Narayanan, A., et al. Nuclei Isolation from Fresh Frozen Brain Tumors for Single-Nucleus RNA-seq and ATAC-seq.Journal of Visualized Experiments. JoVE. 162, 61542 (2020).
- Kim, M., et al. Single-nucleus transcriptomics reveals functional compartmentalization in syncytial skeletal muscle cells. Nature Communications. 11 (1), 6375 (2020).
- Santos, M. D., et al. Extraction and sequencing of single nuclei from murine skeletal muscles. STAR Protocols. 2 (3), 100694 (2021).
- Safabakhsh, S., et al. Isolating nuclei from frozen human heart tissue for single-nucleus RNA sequencing. Current Protocols. 2 (7), (2022).
- Lau, S. -. F., Cao, H., Fu, A. K. Y., Ip, N. Y. Single-nucleus transcriptome analysis reveals dysregulation of angiogenic endothelial cells and neuroprotective glia in Alzheimer’s disease. Proceedings of the National Academy of Sciences of the United States of America. 117 (41), 25800-25809 (2020).
- Armand, E. J., Li, J., Xie, F., Luo, C., Mukamel, E. A. Single-cell sequencing of brain cell transcriptomes and epigenomes. Neuron. 109 (1), 11-26 (2021).
- Morabito, S., et al. Single-nucleus chromatin accessibility and transcriptomic characterization of Alzheimer’s disease. Nature Genetics. 53 (8), 1143-1155 (2021).
- Chen, S., et al. Spatially resolved transcriptomics reveals genes associated with the vulnerability of middle temporal gyrus in Alzheimer’s disease. Acta Neuropathologica Communications. 10 (1), (2022).
- Paul, F., et al. Transcriptional heterogeneity and lineage commitment in myeloid progenitors. Cell. 163 (7), 1663-1677 (2015).
- Kaufmann, E., et al. BCG educates hematopoietic stem cells to generate protective innate immunity against tuberculosis. Cell. 172 (1-2), 176-190 (2018).
- Christ, A., et al. diet triggers NLRP3-dependent innate immune reprogramming. Cell. 172 (1-2), 162-175 (2018).
- Moorlag, S. J. C. F. M., et al. β-Glucan Induces protective trained immunity against mycobacterium tuberculosis infection: A key role for IL-1. Cell Reports. 31 (7), 107634 (2020).
- de Laval, B., et al. C/EBPβ-dependent epigenetic memory induces trained immunity in hematopoietic stem cells. Cell Stem Cell. 26 (5), 657-674 (2020).
- Renthal, W., et al. Characterization of human mosaic Rett syndrome brain tissue by single-nucleus RNA sequencing. Nature Neuroscience. 21 (12), 1670-1679 (2018).
- Yang, A. C., et al. A human brain vascular atlas reveals diverse mediators of Alzheimer’s risk. Nature. 603 (7903), 885-892 (2022).
- Lee, D. R., Zhang, Y., Rhodes, C. T., Petros, T. J. Generation of single-cell and single-nuclei suspensions from embryonic and adult mouse brains. STAR Protocols. 4 (1), 101944 (2022).
- Corces, M. R., et al. Lineage-specific and single-cell chromatin accessibility charts human hematopoiesis and leukemia evolution. Nature Genetics. 48 (10), 1193-1203 (2016).
- Ranzoni, A. M., et al. Integrative single-cell RNA-seq and ATAC-seq analysis of human developmental hematopoiesis. Cell Stem Cell. 28 (3), 472-487 (2021).
- . 10X Genomics Chromium Next GEM Single Cell Multiome ATAC + Gene Expression User Guide. Document Number CG000338 Rev F. At. , (2022).
- J, , et al. Microdissection of mouse brain into functionally and anatomically different regions. Journal of Visualized Experiments: JoVE. 168, 61941 (2021).
- . . 10X Genomics 10X Genomics Demonstrated Protocol – Nuclei Isolation for Single Cell Multiome ATAC + GEX sequencing (CG000365 – Rev C). At. , (2022).
- Haag, S., Murthy, A. Murine monocyte and macrophage culture. Bio-Protocol. 11 (6), (2021).
- Haque, A., Engel, J., Teichmann, S. A., Lönnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Medicine. 9 (1), (2017).
- Jiang, P. Quality control of single-cell RNA-seq. Methods in Molecular Biology. , 1-9 (1935).
- Regan, C., Preall, J. Practical considerations for single-cell genomics. Current Protocols. 2 (8), (2022).
- . . 10X Genomics 10X Genomics Demonstrated Protocol – Nuclei Isolation for Single Cell ATAC Sequencing (CG000169 – Rev E). At. , (2022).
- . 10X Genomics 10X Genomics Demonstrated Protocol – Nuclei Isolation from Complex Tissues for Single Cell Multiome. ATAC + Gene Expression Sequencing. (CG000375 – Rev C). At. , (2022).
- . . 10X Genomics 10X Genomics – Chromium Nuclei Isolation Kit (CG000505 – Rev A). AT. , (2022).
- Shomroni, O., et al. A novel single-cell RNA-sequencing approach and its applicability connecting genotype to phenotype in ageing disease. Scientific Reports. 12 (1), 4091 (2022).
- Ocañas, S. R., Pham, K. D., Blankenship, H. E., Machalinski, A. H., Chucair-Elliott, A. J., Freeman, W. M. Minimizing the ex vivo confounds of cell-isolation techniques on transcriptomic and translatomic profiles of purified microglia. 9 (2), 0348-0321 (2022).
- Gérard, A., et al. High-throughput single-cell activity-based screening and sequencing of antibodies using droplet microfluidics. Nature Biotechnology. 38 (6), 715-721 (2020).
- Vandereyken, K., Sifrim, A., Thienpont, B., Voet, T. Methods and applications for single-cell and spatial multi-omics. Nature Reviews. Genetics. 24, 494-515 (2023).

