Preparazione di nuclei cerebrali e del midollo osseo di alta qualità per saggi multiomi a nucleo singolo
Summary
Il successo della trascrittomica e della multiomica a singola cellula/singolo nucleo dipende in gran parte dalla qualità delle cellule/nuclei. Pertanto, l’isolamento di cellule/nuclei dai tessuti e la loro purificazione devono essere altamente standardizzati. Questo protocollo descrive la preparazione di nuclei dal cervello e dal midollo osseo per il test multioma a singolo nucleo a valle.
Abstract
L’analisi a singola cellula è diventata l’approccio di scelta per svelare la complessità dei processi biologici che richiedono la valutazione della variabilità delle risposte cellulari individuali al trattamento o all’infezione con risoluzione a singola cellula.
Negli ultimi 10 anni sono state sviluppate molte tecniche per la profilazione molecolare di singole cellule e sono state commercializzate diverse tecnologie dedicate. La profilazione a singola cellula basata su goccioline 10X Genomics è una tecnologia diffusa che offre reagenti pronti all’uso per la profilazione trascrittomica e multiomica di singole cellule. La tecnologia include flussi di lavoro per il sequenziamento dell’RNA a singola cellula e a singolo nucleo (scRNA-Seq e snRNA-Seq, rispettivamente), scATAC-Seq, profilazione immunitaria a singola cellula (sequenziamento BCR/TCR) e multioma. Quest’ultimo combina informazioni trascrizionali (scRNA-Seq) ed epigenetiche (scATAC-Seq) provenienti dalla stessa cellula.
La qualità (vitalità, integrità, purezza) delle sospensioni unicellulari o mononucleose isolate dai tessuti e analizzate con uno qualsiasi di questi approcci è fondamentale per generare dati di alta qualità. Pertanto, i protocolli di preparazione dei campioni devono essere adattati alle particolarità di ciascun tessuto biologico e garantire la generazione di sospensioni cellulari e nucleiche di alta qualità.
Questo articolo descrive due protocolli per la preparazione di campioni di cervello e midollo osseo per la pipeline 10X Genomics del multioma a valle. I protocolli vengono eseguiti gradualmente e riguardano la dissociazione tissutale, lo smistamento cellulare, l’isolamento dei nuclei e il controllo di qualità della sospensione di nuclei preparata che viene utilizzata come materiale di partenza per il partizionamento cellulare e la codifica a barre, la preparazione della libreria e il sequenziamento. Questi protocolli standardizzati producono librerie di nuclei di alta qualità e dati robusti e affidabili.
Introduction
Per molti anni, le tecniche a singola cellula sono state il gold standard per l’analisi dei processi biologici. Inizialmente erano limitati alla fenotipizzazione di singole cellule attraverso microscopia, citometria a flusso e saggi simili. Una svolta nell’analisi di singole cellule è arrivata con lo sviluppo di approcci per la profilazione molecolare di singole cellule, in particolare il sequenziamento dell’RNA a singola cellula (scRNA-Seq) che ha permesso di caratterizzare l’intero trascrittoma delle singole cellule. Molto potente, scRNA-Seq genera informazioni sullo stato trascrizionale di una cellula in una condizione e in un punto temporale specifici. Tuttavia, non fornisce visibilità sulla regolazione genica che guida la trascrizione, o sulle modificazioni molecolari che si verificano nel tempo. Per superare questa limitazione, molti sforzi sono stati investiti nello sviluppo di saggi multi-omici a singola cellula che consentono l’analisi di più fattori e processi dalla stessa cellula 1,2,3,4. La prima misurazione di successo di due modalità all’interno di singole cellule è avvenuta collegando i modelli di espressione delle proteine di superficie multiplex con il trascrittoma completo delle singole cellule nell’approccio CITE-Seq5. Evoluzioni più recenti combinano l’espressione genica con l’accessibilità della cromatina (Assay for Transposase-Accessible Chromatin using sequencing, ATAC-Seq), catturando così simultaneamente le modalità trascrittomiche ed epigenomiche nelle stesse cellule (ad esempio, sci-CAR)6. Le prime soluzioni commerciali che permettevano di associare la trascrittomica al fenotipo cellulare o a modificazioni epigenetiche della stessa cellula provenivano dalla 10X Genomics.
Gli esperimenti per la profilazione molecolare a singola cellula contengono le seguenti fasi: (1) dissociazione tissutale o preparazione di sospensioni unicellulari; (2) purificazione cellulare e/o isolamento dei nuclei; (3) partizionamento e codifica a barre; (4) costruzione di biblioteche e controllo di qualità; (5) sequenziamento di nuova generazione; (6) analisi dei dati. Mentre le fasi (3)-(6) possono variare in modo significativo a seconda della tecnologia impiegata, le fasi iniziali sono generalmente comuni a tutte. La qualità della sospensione di cellule/nuclei preparata determinerà l’esito complessivo dell’esperimento. A seconda del tipo di tessuto, può essere difficile ottenere sospensioni unicellulari/nucleiche di alta qualità. Le particolarità di alcuni tessuti, come il cuore, il muscolo, il cervello, il polmone, l’intestino e altri, richiedono metodi di disgregazione tissutale e di isolamento dei nuclei adattati a ciascun tipo di campione al fine di garantire la produzione di nuclei di alta qualità per l’analisi molecolare 7,8,9,10 . I metodi di disgregazione tissutale e i protocolli di dissociazione possono essere meccanici, enzimatici (ad esempio, un mix di collagenasi e DNasi) o una combinazione dei due, e possono essere eseguiti manualmente o con strumenti (ad esempio, Qiagen DSC-400, gentleMACS).
Le tecniche unicellulari sono diventate uno strumento privilegiato per la ricerca biomedica. In neurobiologia, la diversità cellulare nel cervello e la complessità delle loro funzioni richiedono analisi ad alta risoluzione e ad alto rendimento per la visualizzazione di popolazioni cellulari rare e per la valutazione della loro eterogeneità 11,12,13,14. Il collegamento tra l’identità cellulare e i meccanismi di regolazione genica delle singole cellule fornisce informazioni sullo sviluppo e sulla fisiologia del cervello. Un altro esempio sono gli studi sulla risposta immunitaria nel contesto di malattie infettive, autoimmuni o tumorali, che si basano fortemente su analisi di singole cellule. L’eterogeneità dei sottoinsiemi di cellule immunitarie e la complessità della loro attività e delle interazioni con altri tipi di cellule richiedono una risoluzione a singola cellula per decifrare i meccanismi alla base della risposta immunitaria. Le cellule immunitarie hanno origine dal midollo osseo, dove i progenitori ematopoietici sono composti da cellule che si differenziano gradualmente e che acquisiscono e perdono marcatori di superficie cellulare durante un processo graduale prima di uscire dal midollo osseo per tornare a casa in periferia. L’analisi di singole cellule consente una caratterizzazione minuziosa delle fasi di sviluppo cellulare. Può essere ottenuto attraverso la fenotipizzazione di una singola cellula, convenzionalmente eseguita mediante citometria a flusso multiparametrica. Tuttavia, è stato dimostrato che le firme trascrittomiche a singola cellula rivelano un’identificazione più precisa dei sottotipi di cellule progenitrici, poiché queste cellule sono distribuite in cluster che cadono l’una nell’altra e possono quindi essere erroneamente identificate quando si utilizza un approccio con marcatori di superficie cellulare grossolani15. Un numero crescente di studi scopre le modificazioni epigenetiche che le cellule staminali e progenitrici ematopoietiche (HSPC) possono acquisire dall’esposizione a vari agenti, portando a un impatto significativo sulla risposta a lungo termine del sistema immunitario 16,17,18,19. Le nuove tecnologie multi-omiche consentono di studiare questi processi con risoluzione a singola cellula.
Molti protocolli per l’isolamento di cellule e nuclei sono stati descritti per i campioni di cervello 11,20,21,22 e midollo osseo 23,24. Per ridurre al minimo le distorsioni dovute alla variabilità sperimentale, è necessario convalidare protocolli di preparazione ottimizzati per il sequenziamento trascrittomico ed epigenomico congiunto a singola cellula, garantendo così la riproducibilità dei saggi multiomici a singola cellula.
Qui, vengono descritti due protocolli robusti per la preparazione dei nuclei da (1) tessuto cerebrale fresco-congelato e (2) HSPC di midollo osseo fresco per l’analisi del multioma a singola cellula a valle (Figura 1).
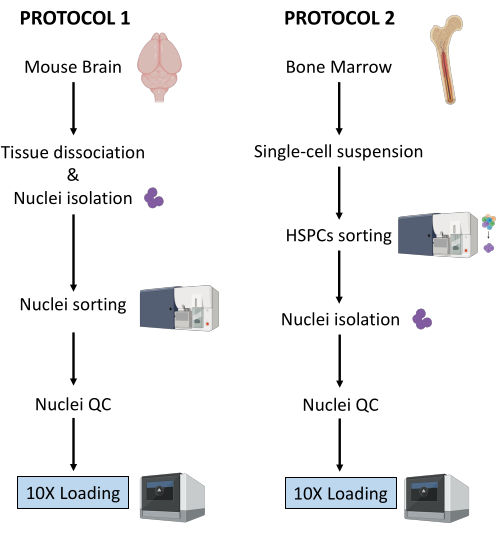
Figura 1: Rappresentazione schematica dei protocolli per l’isolamento dei nuclei da tessuti cerebrali e midollari freschi congelati. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
La preparazione di sospensioni cellulari o nucleiche di alta qualità è di fondamentale importanza per il successo delle analisi RNA-Seq a singola cellula o a singolo nucleo e multi-omiche a singola cellula 29,30,31. Qui, abbiamo descritto i protocolli per la preparazione del campione e l’isolamento dei nuclei per saggi multiomi da due tipi di tessuto: cervello e midollo osseo.
Il protocollo cerebrale descritto in questo articolo consente il recupero di nuclei di alta qualità da tessuto cerebrale appena congelato. Comprende le seguenti fasi: rottura del tessuto congelato, isolamento dei nuclei, purificazione dei nuclei e controllo di qualità del materiale preparato. Il tessuto cerebrale è composto da molti tipi di cellule diverse e la procedura di dissociazione dei tessuti e l’isolamento dei nuclei dovrebbe preservare le proporzioni delle popolazioni cellulari presenti nel tessuto iniziale. In questo caso, la composizione del tampone di lisi e il tempo di incubazione sono stati ottimizzati per consentire una lisi completa e delicata di tutte le popolazioni cellulari che compongono il tessuto.
Il protocollo HSPCs del midollo osseo è un po’ diverso poiché richiede un passaggio aggiuntivo all’inizio dell’esperimento per isolare la popolazione cellulare di interesse da una sospensione cellulare eterogenea. Dopo la raccolta del tessuto fresco, i globuli rossi vengono lisati e il campione viene arricchito per il sottogruppo di cellule di interesse. Le cellule bersaglio vengono lisate, i nuclei vengono isolati e la qualità del materiale preparato viene controllata.
10X Genomics fornisce diversi protocolli validati per l’isolamento dei nuclei in numerosi tessuti diversi32,33. L’azienda commercializza anche un kit di isolamento dei nuclei con una pipeline semplice per isolare i nuclei dai tessuti convalidati34. Tuttavia, questi protocolli necessitano di un’ulteriore ottimizzazione per personalizzare le particolarità di determinati campioni. Un esempio sono gli esempi che richiedono di lavorare con un input di celle basso. Per questi campioni, le fasi più impegnative sono le centrifugazioni che devono essere sufficientemente rigorose per pulire il campione e abbastanza delicate da evitare la perdita di cellule/nuclei. Con il protocollo qui descritto, abbiamo adattato il 10X Genomics Demonstration Protocol – Nuclei Isolation for Single Cell Multiome ATAC + GEX Sequencing (CG000365 – Rev C)27 per trovare un buon equilibrio tra questi due requisiti. Come dimostrato nell’esempio della preparazione di nuclei da HSPC selezionati, abbiamo migliorato il recupero dei nuclei senza alcun impatto sulla qualità del campione.
Un’ulteriore sfida è la fase di lisi delle cellule purificate per l’isolamento dei nuclei. Condizioni di lisi più dure e tempi di incubazione più lunghi possono danneggiare i nuclei e quindi influire sulla qualità dei dati di sequenziamento. La Figura 5 mostra l’imaging rappresentativo dei nuclei da campioni di midollo osseo con diversi tempi di incubazione con tampone di lisi e illustra quanto possa essere diverso lo stato dei nuclei a seconda della lisi cellulare. Nell’esempio delle HSPC, abbiamo identificato la lisi di 3 minuti come la condizione che si traduce nella più alta percentuale di nuclei dall’aspetto sano e intatto e nella più bassa percentuale di nuclei danneggiati. I tempi di incubazione della lisi devono essere ottimizzati per ogni nuovo tipo di campione.
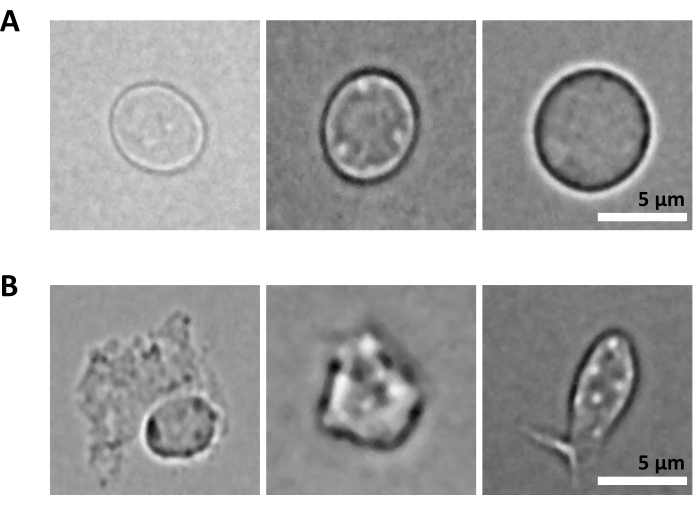
Figura 5: Controllo di qualità dei nuclei al microscopio. Sono mostrate immagini rappresentative in campo chiaro di nuclei isolati dal midollo osseo di topo con (A) nuclei intatti e (B) danneggiati. Barra graduata 5 μm. Le immagini sono state scattate con un microscopio invertito utilizzando un obiettivo ELWD NA 0,60 40x e uno zoom digitale 1,5x. Fare clic qui per visualizzare una versione più grande di questa figura.
Entrambi i protocolli descritti in questo lavoro si basano sulla purificazione di cellule o nuclei mirati mediante strumenti FACS ad alto rendimento. Questo passaggio è di fondamentale importanza per i protocolli di preparazione di singole cellule/nuclei in cui sottoinsiemi rari di cellule devono essere isolati da sospensioni eterogenee. In questi casi, come nell’esempio mostrato qui per lo smistamento delle HSPC, può essere necessario un pannello di citometria a flusso ad alta dimensione per abilitare il “gating” sulla popolazione cellulare di interesse. Lo smistamento è estremamente veloce e accurato, portando a una purezza superiore al 95% dei sottoinsiemi di cellule selezionate. Questo approccio espone la sospensione cellulare a una pressione fino a 70 psi e può quindi essere limitante per lo smistamento di cellule fragili (ad esempio, cellule dendritiche, neutrofili) poiché può causare la rottura della loro membrana cellulare. In questi casi, dovrebbero essere selezionate soluzioni alternative per la purificazione cellulare, incluso il sorting magnetico, l’applicazione di strumenti di nuova generazione (ad esempio, CellenOne, Cellenion; MACSQuant Tyto, Miltenyi)35,36 o sistemi basati su goccioline (ad esempio, ODIN, Sensific)37. Ciononostante, la bassa velocità di smistamento di queste tecnologie, con lo smistamento cellulare che dura ore anziché minuti, è un forte fattore limitante per l’applicazione di questi approcci nella preparazione di cellule vitali per Multiome e altre applicazioni a cella singola basate sull’analisi di un grande numero di cellule.
Per la purificazione dei nuclei isolati dal tessuto, FACS è il metodo di scelta per la sua produttività e la purezza del materiale isolato. I nuclei non sono sensibili alla pressione e gli isolati di tessuto filtrato possono essere facilmente purificati attraverso il selezionatore di cellule. Se il laboratorio non è dotato di uno strumento FACS, esistono altre alternative, un po’ meno efficienti ma sufficientemente buone. Ne sono un esempio l’ultracentrifugazione o l’utilizzo di piccole apparecchiature come MARS (Applied Cell) che separa le particelle in base alla loro differenza di dimensione, utilizzando onde acustiche; Rondella laminare CURIOX che sfrutta le proprietà idrofobiche delle sospensioni cellule/nuclei; o LEVITAS bio che si basa sulle proprietà fisiche delle cellule (levitazione) per separarle dai detriti.
Qui descriviamo i protocolli per ottenere un numero elevato di nuclei e la migliore purezza per il protocollo Multiome a valle. La cernita FACS e le ripetute fasi di centrifugazione comportano una sostanziale perdita del materiale iniziale. Per questo motivo, nel protocollo per la preparazione dei nuclei dal cervello che descriviamo qui è necessario materiale di partenza sufficientemente abbondante per portare alla raccolta di almeno 500.000 nuclei dopo lo smistamento FACS. Se questo criterio non può essere soddisfatto, devono essere applicati protocolli alternativi. Quando si lavora con popolazioni cellulari rare o piccole sezioni di tessuto, la quantità disponibile di materiale iniziale può essere un fattore limitante. Per affrontare questo problema, è possibile migliorare il recupero dei nuclei (a) riducendo il volume di lisi, (b) riducendo il volume di lavaggio, (c) utilizzando un singolo lavaggio con un tempo di centrifugazione prolungato per cercare di migliorare il recupero come indicato nei protocolli di genomica 10X per l’isolamento dei nuclei a basso input cellulare. Per l’analisi multiomica di materiale a basso contenuto, vale la pena prendere in considerazione applicazioni basate su piastre come scNMT, SNARE-seq e Paired-seq38 che richiedono un numero molto inferiore di campioni in ingresso.
In sintesi, abbiamo descritto due protocolli robusti per la preparazione di nuclei dal cervello e dal midollo osseo HSPC per l’analisi del multioma a valle. Questi protocolli sono applicabili in qualsiasi progetto scientifico che richieda sospensioni di singoli nuclei di alta qualità da questi due tipi di tessuto, indipendentemente dal quesito scientifico posto. Il nostro gruppo ha applicato il protocollo di isolamento dei nuclei cerebrali in studi sullo sviluppo cerebrale in seguito all’inattivazione di vari geni bersaglio e in studi sulla risposta immunitaria nel contesto di malattie neurologiche. Stiamo utilizzando il protocollo di isolamento dei nuclei del midollo osseo per decifrare la partecipazione di varie sottopopolazioni ematopoietiche nella costituzione del sistema immunitario.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ana Jeemin Choi è stata sostenuta da una borsa di studio del programma di dottorato internazionale dell’Università Pasteur di Parigi (PPU).
Materials
| 18 G x 1 ½ (1.2 mm x 38 mm) Agani needles | Terumo | AN*1838S1 | |
| 15 mL tubes | Falcon | 352097 | |
| 5 mL round bottom FACS tube with cell strainer cap 35 µm | falcon | 352235 | |
| 50 mL tubes | Falcon | 352070 | |
| 7-AAD | BD pharmagen | 559925 | |
| ACK Lysing Buffer | Gibco | A10492-01 | |
| APC anti-mouse CD117 (c-Kit) | BioLegend | 105812 | Clone: 2B8 |
| APC/Cyanine 7 anti-mouse CD16/32 (FcγR) | BioLegend | 101328 | Clone: 93 |
| BD FACSAria III | BD Biosciences | non-applicable | |
| BD FACSDiva Software v8.0.1 | BD Biosciences | non-applicable | |
| Bovine Serum Albumin stock solution 10% | Miltenyi Biotec | 130-091-376 | |
| Cell staining buffer | Biolegend | 420201 | |
| CFI Suprplan Fluor ELWD 40XC ON 0.6 | Nikon | non-applicable | |
| CMOS camera Prime 95B 25 mm | Photometrix | non-applicable | |
| Countess II FL Automated Cell Counter | Invitrogen | AMQAF1000 | |
| Countess cell counting chamber slide | Invitrogen | C10283 | |
| Coverglass 24 mm x 24 mm 0.13-0.17 mm | Brand | BR470819 | |
| Digitonine 5% | Invitrogen | BN2006 | |
| Disposable Scalpels | Swann-Morton | 0508 | |
| DMEM (1x) + GlutaMAX-I | Gibco | 31966-021 | |
| DPBS (10x) | Gibco | 14200-067 | |
| DTT | Sigma aldrich | 646563 | |
| Epifluorescence inverted microscope Nikon Ti2 -E | Nikon | non-applicable | |
| Eppendorf Safe-Lock Tubes 0.5 mL | Eppendorf | 30123603 | |
| Ethanol 70% | VWR | 83801.290 | |
| FITC anti-mouse CD34 | Invitrogen | 11-0341-85 | Clone: RAM34 |
| Forceps for dissection | FST | 11152-10 | |
| Heat-inactivated Fetal Bovine Serum (FBS) | Gibco | 11533387 | |
| Dounce Homogeniser 2 mL | Bellco glass | 1984-10002 | Pestle “A” Large Clearance: .0030-.0050″ and Pestle “B” Small Clearance: .0005-.0025″ |
| LIVE/DEAD fixable aqua dead cell stain kit | Invitrogen | L34957 | |
| Magnesium chloride solution 1 M | Sigma aldrich | M1028 | |
| Microcentrifuge | Eppendorf | 5424R | |
| Mounting medium Fluoromount-G | invitrogen | 00-4958-02 | |
| Nonidet P40 substitute | Sigma aldrich | 74385 | |
| Nuclease free water | ThermoFischer | AM9932 | |
| Nuclei buffer 20x | 10X Genomics | 2000153/2000207 | |
| Nuclei isolation kit EZ prep | Sigma Aldrich | NUC-101 | |
| OneComp eBeads compensation beads | Invitrogen | 01-1111-41 | |
| Pacific Blue anti-mouse lineage cocktail (including anti-mouse CD3, Ly-6G/Ly-6C, CD11b, CD45R/B220, TER-119) |
BioLegend | 133310 | Clones (in the same order as the antibodies listed): 17A2, RB6-8C5, M1/70, RA3-6B2, Ter-119 |
| PCR Tube Strips 0.2 mL | Eppendorf | 951010022 | |
| PE anti-mouse Ly-6A/E (Sca-1) | BioLegend | 122507 | Clone: E13-161.7 |
| Petri dish 100 mm x 20 mm OPTILUX | Falcon | 353003 | |
| Ply-L-lysine 0.01% sterile-filtered suitable for cell culture | Sigma | P4707 | |
| Printed microscope slides 8 well 6 mm numbered | Epredia | ER-301B-CE24 | |
| Protein LoBind Tubes 1.5 mL | Eppendorf | 30108116 | |
| Recombinant Rnase inhibitor 5000 U | Takara | 2313A | |
| Scissors for dissection | FST | 14090-09 | |
| Sodium chloride solution 5 M | Sigma aldrich | 59222C | |
| Syringe filters, PES, 0.2 µm | Fisher Scientific | 15206869 | |
| Transparent nail polish | any | non-applicable | |
| Trizma Hydrochloride solution pH 7.4 | Sigma aldrich | T2194 | |
| Trypan Blue 0.4% | gibco | 15250061 | |
| Tween 20 | Biorad | 1662404 | |
| UltraPure Distilated Water Dnase/Rnase Free | Invitrogen | 10977-035 |
References
- Clark, S. J., et al. scNMT-seq enables joint profiling of chromatin accessibility DNA methylation and transcription in single cells. Nature Communications. 9 (1), 781 (2018).
- Lee, J., Hyeon, D. Y., Hwang, D. Single-cell multiomics: technologies and data analysis methods. Experimental & Molecular Medicine. 52 (9), 1428-1442 (2020).
- Cerrizuela, S., et al. High-throughput scNMT protocol for multiomics profiling of single cells from mouse brain and pancreatic organoids. STAR Protocols. 3 (3), 101555 (2022).
- Dimitriu, M. A., Lazar-Contes, I., Roszkowski, M., Mansuy, I. M. Single-cell multiomics techniques: From conception to applications. Frontiers in Cell and Developmental Biology. 10, 854317 (2022).
- Stoeckius, M., et al. Simultaneous epitope and transcriptome measurement in single cells. Nature Methods. 14 (9), 865-868 (2017).
- Cao, J., et al. . Joint profiling of chromatin accessibility and gene expression in thousands of single cells. 361 (6409), 1380-1385 (2018).
- Narayanan, A., et al. Nuclei Isolation from Fresh Frozen Brain Tumors for Single-Nucleus RNA-seq and ATAC-seq.Journal of Visualized Experiments. JoVE. 162, 61542 (2020).
- Kim, M., et al. Single-nucleus transcriptomics reveals functional compartmentalization in syncytial skeletal muscle cells. Nature Communications. 11 (1), 6375 (2020).
- Santos, M. D., et al. Extraction and sequencing of single nuclei from murine skeletal muscles. STAR Protocols. 2 (3), 100694 (2021).
- Safabakhsh, S., et al. Isolating nuclei from frozen human heart tissue for single-nucleus RNA sequencing. Current Protocols. 2 (7), (2022).
- Lau, S. -. F., Cao, H., Fu, A. K. Y., Ip, N. Y. Single-nucleus transcriptome analysis reveals dysregulation of angiogenic endothelial cells and neuroprotective glia in Alzheimer’s disease. Proceedings of the National Academy of Sciences of the United States of America. 117 (41), 25800-25809 (2020).
- Armand, E. J., Li, J., Xie, F., Luo, C., Mukamel, E. A. Single-cell sequencing of brain cell transcriptomes and epigenomes. Neuron. 109 (1), 11-26 (2021).
- Morabito, S., et al. Single-nucleus chromatin accessibility and transcriptomic characterization of Alzheimer’s disease. Nature Genetics. 53 (8), 1143-1155 (2021).
- Chen, S., et al. Spatially resolved transcriptomics reveals genes associated with the vulnerability of middle temporal gyrus in Alzheimer’s disease. Acta Neuropathologica Communications. 10 (1), (2022).
- Paul, F., et al. Transcriptional heterogeneity and lineage commitment in myeloid progenitors. Cell. 163 (7), 1663-1677 (2015).
- Kaufmann, E., et al. BCG educates hematopoietic stem cells to generate protective innate immunity against tuberculosis. Cell. 172 (1-2), 176-190 (2018).
- Christ, A., et al. diet triggers NLRP3-dependent innate immune reprogramming. Cell. 172 (1-2), 162-175 (2018).
- Moorlag, S. J. C. F. M., et al. β-Glucan Induces protective trained immunity against mycobacterium tuberculosis infection: A key role for IL-1. Cell Reports. 31 (7), 107634 (2020).
- de Laval, B., et al. C/EBPβ-dependent epigenetic memory induces trained immunity in hematopoietic stem cells. Cell Stem Cell. 26 (5), 657-674 (2020).
- Renthal, W., et al. Characterization of human mosaic Rett syndrome brain tissue by single-nucleus RNA sequencing. Nature Neuroscience. 21 (12), 1670-1679 (2018).
- Yang, A. C., et al. A human brain vascular atlas reveals diverse mediators of Alzheimer’s risk. Nature. 603 (7903), 885-892 (2022).
- Lee, D. R., Zhang, Y., Rhodes, C. T., Petros, T. J. Generation of single-cell and single-nuclei suspensions from embryonic and adult mouse brains. STAR Protocols. 4 (1), 101944 (2022).
- Corces, M. R., et al. Lineage-specific and single-cell chromatin accessibility charts human hematopoiesis and leukemia evolution. Nature Genetics. 48 (10), 1193-1203 (2016).
- Ranzoni, A. M., et al. Integrative single-cell RNA-seq and ATAC-seq analysis of human developmental hematopoiesis. Cell Stem Cell. 28 (3), 472-487 (2021).
- . 10X Genomics Chromium Next GEM Single Cell Multiome ATAC + Gene Expression User Guide. Document Number CG000338 Rev F. At. , (2022).
- J, , et al. Microdissection of mouse brain into functionally and anatomically different regions. Journal of Visualized Experiments: JoVE. 168, 61941 (2021).
- . . 10X Genomics 10X Genomics Demonstrated Protocol – Nuclei Isolation for Single Cell Multiome ATAC + GEX sequencing (CG000365 – Rev C). At. , (2022).
- Haag, S., Murthy, A. Murine monocyte and macrophage culture. Bio-Protocol. 11 (6), (2021).
- Haque, A., Engel, J., Teichmann, S. A., Lönnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Medicine. 9 (1), (2017).
- Jiang, P. Quality control of single-cell RNA-seq. Methods in Molecular Biology. , 1-9 (1935).
- Regan, C., Preall, J. Practical considerations for single-cell genomics. Current Protocols. 2 (8), (2022).
- . . 10X Genomics 10X Genomics Demonstrated Protocol – Nuclei Isolation for Single Cell ATAC Sequencing (CG000169 – Rev E). At. , (2022).
- . 10X Genomics 10X Genomics Demonstrated Protocol – Nuclei Isolation from Complex Tissues for Single Cell Multiome. ATAC + Gene Expression Sequencing. (CG000375 – Rev C). At. , (2022).
- . . 10X Genomics 10X Genomics – Chromium Nuclei Isolation Kit (CG000505 – Rev A). AT. , (2022).
- Shomroni, O., et al. A novel single-cell RNA-sequencing approach and its applicability connecting genotype to phenotype in ageing disease. Scientific Reports. 12 (1), 4091 (2022).
- Ocañas, S. R., Pham, K. D., Blankenship, H. E., Machalinski, A. H., Chucair-Elliott, A. J., Freeman, W. M. Minimizing the ex vivo confounds of cell-isolation techniques on transcriptomic and translatomic profiles of purified microglia. 9 (2), 0348-0321 (2022).
- Gérard, A., et al. High-throughput single-cell activity-based screening and sequencing of antibodies using droplet microfluidics. Nature Biotechnology. 38 (6), 715-721 (2020).
- Vandereyken, K., Sifrim, A., Thienpont, B., Voet, T. Methods and applications for single-cell and spatial multi-omics. Nature Reviews. Genetics. 24, 494-515 (2023).

