Funktionsbewertung von BRCA1-Varianten mit CRISPR-mediated Base Editors
Summary
Menschen mit BRCA1-Mutationen haben ein höheres Risiko, an Krebs zu erkranken, was eine genaue Bewertung der Funktion von BRCA1-Varianten rechtfertigt. Hierin haben wir ein Protokoll zur funktionellen Bewertung von BRCA1-Varianten mit CRISPR-vermittelten Cytosin-Basiseditoren beschrieben, die eine gezielte Konvertierung von C:G in T:A in lebenden Zellen ermöglichen.
Abstract
Jüngste Studien haben die Risiken im Zusammenhang mit BRCA1-Genmutationen mit verschiedenen funktionellen Bewertungsmethoden wie fluoreszierenden Reporter-Assays, embryonalen Stammzell-Viability-Assays und therapeutischen medikamentenbasierten Sensitivitätstests untersucht. Obwohl sie viele BRCA1-Varianten geklärt haben, sind diese Assays, die die Verwendung exogen ausgedrückter BRCA1-Varianten beinhalten, mit Überexpressionsproblemen verbunden und können nicht auf posttranskriptionelle Regulierung angewendet werden. Um diese Einschränkungen zu beheben, berichteten wir zuvor über eine Methode zur funktionellen Analyse von BRCA1-Varianten über den CRISPR-vermittelten Cytosin-Basiseditor, die eine gezielte Nukleotidsubstitution in lebenden Zellen induzieren. Mit dieser Methode identifizierten wir Varianten, deren Funktionen mehrdeutig bleiben, einschließlich c.-97C>T, c.154C>T, c.3847C>T, c.5056C>T und c.4986+5G>A, und bestätigten, dass CRISPR-vermittelte Basiseditoren nützliche Werkzeuge sind, um die in BRCA1unsicheren Varianten der Bedeutung neu zu klassifizieren. Hier beschreiben wir ein Protokoll zur funktionstüchchenanalyse von BRCA1-Varianten mit DEM CRISPR-basierten Cytosin-Basiseditor. Dieses Protokoll enthält Richtlinien für die Auswahl von Zielstandorten, die Funktionsanalyse und Bewertung von BRCA1-Varianten.
Introduction
Das Brustkrebstyp-1-Anfälligkeitsgen (BRCA1) ist ein weithin bekanntes Tumorsuppressorgen. Da das BRCA1-Gen mit der Reparatur von DNA-Schäden zusammenhängt, würden Mutationen in diesem Gen zu einem höheren Risiko für die Krebsentwicklung bei einem Individuum führen1. Brust-, Eierstock-, Prostata- und Bauchspeicheldrüsenkrebs sind mit vererbten Funktionsverlustmutationen (LOF) des BRCA1-Gens 2verbunden. Die funktionelle Bewertung und Identifizierung von BRCA1-Varianten kann bei der Prävention und Diagnose der verschiedenen Krankheiten helfen. Um die Funktion von BRCA1-Varianten zu adressieren, wurden verschiedene Methoden entwickelt und breit zur Untersuchung der Pathogenität von BRCA1-Varianten wie embryonalen Stammzell-Viability-Assays, fluoreszierenden Reporter-Assays und therapeutischen drogenbasierten Sensitivitäts-Assays3,4,5,6verwendet. Obwohl diese Methoden die Funktion vieler BRCA1-Varianten bewertet haben, stellen die Methoden mit exogen exogen exzessiv ausgedrückten BRCA1-Varianten Einschränkungen in Bezug auf Überexpression dar, die die nachgelagerte Regulierung, Gendosierung und Proteinfaltung beeinträchtigen könnten7. Darüber hinaus können diese Assays nicht auf die posttranskriptionelle Regulierung wie mRNA-Spleißen, Transkriptionstabilität und Wirkung der unübersetzten Region8,9genutzt werden.
CRISPR-Cas9 System ermöglicht gezielte Genombearbeitung in lebenden Zellen und Organismen10. Durch eine Single-Guide-RNA kann Cas9 Doppelstrangbrüche (DSBs) in der chromosomalen DNA an bestimmten genomischen Loci induzieren, um zwei DNA-Reparaturpfade zu aktivieren: fehleranfällige nicht-homologe Endverbindung (NHEJ) und fehlerfreie Homologie-gesteuerte Reparatur (HDR)11. HDR ist ein präziser Reparaturmechanismus; DSBs, die durch Cas9-Nuklease für HDR induziert werden, führen jedoch häufig zu unerwünschter Insertion und Deletion (Indel) Mutation. Darüber hinaus benötigt es homologe Spender-DNA-Vorlagen für die Reparatur von DNA-Schäden und hat eine relativ geringe Effizienz. Kürzlich wurden Cas9-Nickase (nCas9) mit Cytidin-Deaminase-Domänen für die Ausrichtung von C:G bis T:A-Substitutionen verschmolzen, ohne dass homologe DNA-Vorlagen und DNA-Doppelstrangbrüche12,13,14,15. Mit dem Cytosin-Basiseditor haben wir eine neue Methode zur funktionstüchchenden Analyse der BRCA1-Varianten16entwickelt.
In dieser Studie verwendeten wir CRISPR-vermittelten Cytosin-Basiseditor, BE314, der effiziente C:G- bis T:A-Punktmutationen induziert, für die Implementierung der funktionellen Bewertung von BRCA1-Varianten und identifizierten erfolgreich die Funktionen mehrerer BRCA1-Varianten (Abbildung 1).
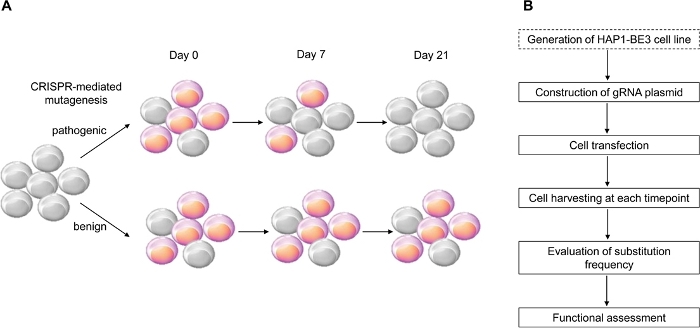
Abbildung 1: Eine Übersicht über den Workflow für die funktionale Bewertung. (A) Schema, das die funktionelle Bewertung von BRCA1zeigt. Da die LOF von BRCA1 die Zelllebensfähigkeit beeinflusst, sterben die Zellen ab, wenn die BRCA1-Mutation pathogen ist, wenn die Durchgangszahl zunimmt. (B) Phasen der funktionellen Bewertung von BRCA1. Gepunktete Box ist optional. Es kann durch Ko-Transfektion von gRNA-Exzess und BE3-exemittiert Erdplasmiden DNA ersetzt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protocol
Representative Results
Discussion
Dieses Protokoll beschreibt eine einfache Methode für funktionale Bewertungen von BRCA1-Varianten mit DEM CRISPR-meditierten Cytosin-Basiseditor. Das Protokoll beschreibt Methoden für die Konstruktion von gRNAs am Zielort und die Konstruktion der Plasmid-DNAs, aus denen sie exprimiert werden. Cytosin-Basis-Editoren induzieren Nukleotid-Konvertierung in einem aktiven Fenster (bei BE3 Nukleotide 4–8 im PAM-distalen Ende der gRNA-Zielsequenzen). Der Forscher sollte sorgfältig Zielsequenzen auswählen, da alle …
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
Diese Arbeit wurde von der National Research Foundation of Korea unterstützt (Grants 2017M3A9B4062419, 2019R1F1A1057637 und 2018R1A5A2020732 an Y.K.).
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
Referenzen
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. Krebsforschung. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

