CRISPR 중재 베이스 편집기사용 BRCA1 변종 기능 평가
Summary
BRCA1 돌연변이를 가진 사람들은 BRCA1 변이체의 기능의 정확한 평가를 보증하는 암 발전의 고위험이 있습니다. 본명, 우리는 살아있는 세포에서 표적 C:G ~T:A 변환을 가능하게 하는 CRISPR 매개 사이토신 베이스 편집기를 사용하여 BRCA1 변이체의 기능적 평가를 위한 프로토콜을 기술했습니다.
Abstract
최근 연구는 형광 기자 의 소사, 배아 줄기 세포 생존 가능성 연구, 치료 약물 기반 감도 검사와 같은 다양한 기능성 평가 방법을 사용하여 BRCA1 유전자 돌연변이와 관련된 위험을 조사했습니다. 그들은 BRCA1 변종을 많이 명확히했지만, 외인성 발현 BRCA1 변종의 사용을 포함하는 이러한 소는 과발현 문제와 관련이 있으며 전사 후 규제에 적용 할 수 없습니다. 이러한 한계를 해결하기 위해, 우리는 이전에 살아있는 세포에서 표적 뉴클레오티드 대체를 유도하는 CRISPR 매개 사이토신 베이스 편집기를 통해 BRCA1 변이체의 기능적 분석을 위한 방법을 보고했습니다. 이 방법을 사용하여 c.-97C>T, c.154C>T, c.3847C>T, c.5056C>T, c.4986+5G>A 등 기능이 모호한 변종을 확인했으며 CRISPR 미디어 베이스 에디터가 BR1의재분류 에 유용한 도구임을 확인했습니다. 여기에서 CRISPR 기반 사이토신 베이스 편집기를 사용하여 BRCA1 변종의 기능 분석을 위한 프로토콜을 설명합니다. 이 프로토콜은 대상 사이트 선택, 기능 분석 및 BRCA1 변형 평가에 대한 지침을 제공합니다.
Introduction
유방암 제1형 감수성유전자(BRCA1)는널리 알려진 종양 억제유전자이다. BRCA1 유전자는 DNA 손상의 복구와 관련이 있기 때문에, 이 유전자에 있는 돌연변이는 개별1에있는 암 발달의 더 중대한 리스크로 이끌어 낼 것입니다. 유방, 난소, 전립선 및 췌장암은 BRCA1 유전자2의승계된 기능 상실 (LOF) 돌연변이에 연결됩니다. BRCA1 변이체의 기능적 평가 및 식별은 다양한 질병을 예방하고 진단하는 데 도움이 될 수 있습니다. BRCA1 변이체의 기능을 해결하기 위해, 배아 줄기 세포 생존성 감수제, 형광 기자 감수제 및 치료약물 기반 감도 감수제3,4,5,6과같은 BRCA1 변종의 병원성을 조사하기 위해 여러 가지 방법이 개발되고 광범위하게 사용되고 있다. 이러한 방법은 BRCA1 변이체의 많은 기능을 평가했지만, 외인성 발현 BRCA1 변이체를 포함하는 방법은 다운스트림 조절, 유전자 투여량 및 단백질 접이식7에영향을 미칠 수 있는 과발현 측면에서 한계를 제기한다. 더욱이, 이러한 소하는 mRNA 접합, 성적증명서 안정성, 번역되지 않은영역8,9의효과와 같은 전사 후 조절에 활용될 수 없다.
CRISPR-Cas9 시스템은 살아있는 세포 및 유기체10에서표적 게놈 편집을 가능하게 합니다. 단일 가이드 RNA를 통해 Cas9는 두 가지 DNA 수리 경로를 활성화하기 위해 특정 게놈 로맥에서 염색체 DNA에서 이중 가닥 브레이크(DSBs)를 유도하여 오류 발생하기 쉬운 비homologous 엔드 결합(NHEJ) 경로 및 오류 없는 homology 지향 수리(HDR)경로(HDR)경로를 활성화할 수 있다. HDR은 정밀한 수리 메커니즘입니다. 그러나, HDR에 대 한 Cas9 핵에 의해 유도 된 DSBs 종종 원치 않는 삽입 및 삭제 결과 (indel) 돌연변이. 추가적으로, DNA 손상을 복구하기 위한 동종 기증자 DNA 템플릿이 필요하고 상대적으로 낮은 효율성을 가지고 있습니다. 최근에는, Cas9 니카아제(nCas9)는 C:G ~T:A 대체를 대상으로 하는 시티딘 데아미나제 도메인과 융합되어 왔으며, 동종 DNA 템플릿및 DNA 이중 가닥 브레이크12,13,14,15에대한 필요 없이. 시토신 베이스 에디터를 사용하여 BRCA1변종(16)의기능적 분석을 위한 새로운 방법을 개발했습니다.
본 연구에서는, CRISPR 매개 사이토신 베이스 에디터, BE314를사용했는데, 이는 효율적인 C:G를 T:A 포인트 돌연변이로 유도하고, BRCA1 변이체의 기능적 평가를 구현하고 여러 BRCA1 변종의 기능을 성공적으로 식별하였다(그림1).
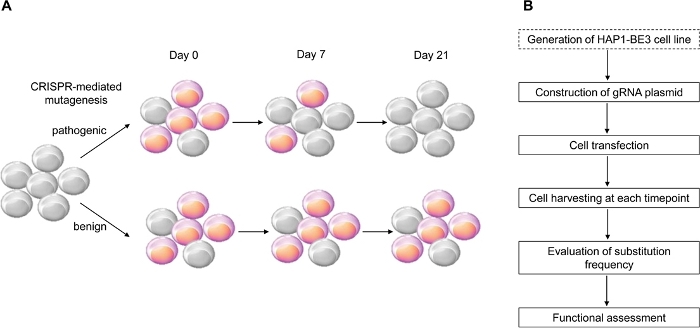
그림 1: 기능 평가를 위한 워크플로에 대한 개요입니다. (A) BRCA1의기능성 평가를 보여주는 회로도. BRCA1의 LOF는 세포 생존에 영향을 미치기 때문에 BRCA1 돌연변이가 병원성일 때, 세포는 통로 수가 증가함에 따라 죽는다. (B) BRCA1의기능성 평가 단계 . 점선 상자는 선택 사항입니다. 그것은 plasmids DNA를 표현하는 gRNA 표현 및 BE3의 공동 형질으로 대체 될 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
이 프로토콜은 CRISPR 명상 사이토신 베이스 편집기를 사용하여 BRCA1 변종의 기능 평가를 위한 간단한 방법을 설명합니다. 이 프로토콜은 표적 궤적에서 gRNA의 설계 및 그들이 표현되는 플라스미드 DNA의 시공에 대한 방법을 설명합니다. Cytosine 염기 편집기는 활성 창에서 뉴클레오티드 변환을 유도합니다(BE3의 경우, 뉴클레오티드 4-8gRNA 표적 서열의 PAM-말단 말단에서). 활성 창에 있는 모든 사?…
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
이 작품은 한국국립연구재단(2017M3A9B4062419, 2019R1F1A1057637, 2018R1A5A2020732)에서 지원했다.
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
Referenzen
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. Krebsforschung. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

