Valutazione funzionale delle varianti BRCA1 utilizzando editor di base mediati da CRISPR
Summary
Le persone con mutazioni BRCA1 hanno un rischio più elevato di sviluppare il cancro, il che giustifica una valutazione accurata della funzione delle varianti BRCA1. Nel presente documento, abbiamo descritto un protocollo per la valutazione funzionale delle varianti BRCA1 utilizzando editor di base citosina mediati da CRISPR che consentono la conversione da C:G a T:A mirata nelle celle viventi.
Abstract
Studi recenti hanno studiato i rischi associati alle mutazioni geniche BRCA1 utilizzando vari metodi di valutazione funzionale come test fluorescenti dei reporter, test di vitalità delle cellule staminali embrionali e test terapeutici di sensibilità a base di farmaci. Sebbene abbiano chiarito molte varianti brca1, questi test che coinvolgono l’uso di varianti BRCA1 espresse esogenamente sono associati a problemi di sovraespressione e non possono essere applicati alla regolamentazione post-trascrizionale. Per risolvere queste limitazioni, abbiamo precedentemente segnalato un metodo per l’analisi funzionale delle varianti BRCA1 tramite l’editor di base di citosina mediato da CRISPR che induce una sostituzione mirata dei nucleotidi nelle cellule viventi. Utilizzando questo metodo, abbiamo identificato varianti le cui funzioni rimangono ambigue, tra cui c.-97C>T, c.154C>T, c.3847C>T, c.5056C>T e c.4986+5G>A, e abbiamo confermato che gli editor di base mediati da CRISPR sono strumenti utili per riclassificare le varianti di significato incerto in BRCA1. Qui, descriviamo un protocollo per l’analisi funzionale delle varianti BRCA1 utilizzando l’editor di base citosina basato su CRISPR. Questo protocollo fornisce linee guida per la selezione dei siti di destinazione, l’analisi funzionale e la valutazione delle varianti BRCA1.
Introduction
Il gene della suscettibilità al cancro al seno di tipo 1(BRCA1)è un gene soppressore tumorale ampiamente noto. Poiché il gene BRCA1 è correlato alla riparazione del danno al DNA, le mutazioni in questo gene porterebbero a un maggiore rischio di sviluppo del cancro in unindividuo 1. I tumori al seno, alle ovaie, alla prostata e al pancreas sono legati a mutazioni ereditarie della perdita di funzione (LOF) del gene BRCA1 2. La valutazione funzionale e l’identificazione delle varianti BRCA1 possono aiutare a prevenire e diagnosticare le varie malattie. Per affrontare la funzione delle varianti BRCA1, sono stati sviluppati diversi metodi e ampiamente utilizzati per indagare la patogenicità delle varianti BRCA1 come test di vitalità delle cellule staminali embrionali, test fluorescenti dei reporter e test terapeutici di sensibilità a base di farmaci3,4,5,6. Sebbene questi metodi abbiano valutato la funzione di molte varianti BRCA1, i metodi che coinvolgono varianti BRCA1 espresse esogenamente pongono limitazioni in termini di sovraespressione che potrebbero influenzare la regolazione a valle, il dosaggio genico e il ripiegamento proteico7. Inoltre, questi saggi non possono essere sfruttati per la regolazione posttrascrizionale come l’affezione dell’mRNA, la stabilità della trascrizione e l’effetto della regionenon tradotta 8,9.
Il sistema CRISPR-Cas9 consente un editing mirato del genoma nelle cellule viventi e negliorganismi 10. Attraverso un RNA a guida singola, Cas9 può indurre rotture a doppio filamento (DSB) nel DNA cromosomico a loci genomici specifici al fine di attivare due percorsi di riparazione del DNA: percorso di terminazione non disomologa (NHEJ) soggetto a errori e percorso di riparazione diretta dall’omologia (HDR) privo dierrori 11. L’HDR è un meccanismo di riparazione preciso; tuttavia, i DSB indotti dalla nucleasi Cas9 per HDR spesso si trae da mutazione indesiderata di inserimento ed eliminazione (indele). Inoltre, ha bisogno di modelli di DNA donatori omologhi per riparare i danni al DNA e ha un’efficienza relativamente bassa. Recentemente, la nickasi Cas9 (nCas9) è stata fusa con domini di deaminasi citidina per indirizzare le sostituzioni da C:G a T:A, senza la necessità di modelli di DNA omologhi e rotture a doppio filamento di DNA12,13,14,15. Utilizzando l’editor di base citosina, abbiamo sviluppato un nuovo metodo per l’analisi funzionale delle varianti BRCA116.
In questo studio, abbiamo utilizzato l’editor di base di citosina mediato da CRISPR, BE314, che induce mutazioni puntili efficienti da C:G a T:A, per implementare la valutazione funzionale delle varianti BRCA1 e ha identificato con successo le funzioni di diverse varianti BRCA1 (Figura 1).
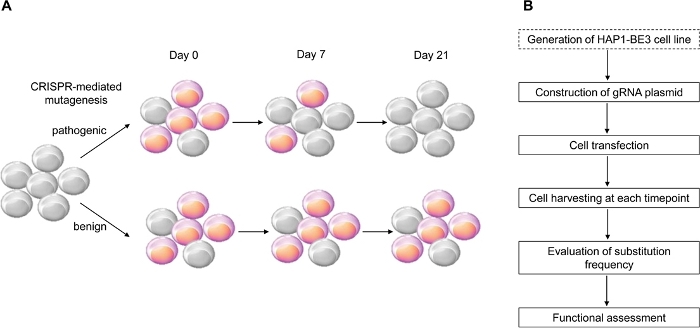
Figura 1: Panoramica del flusso di lavoro per la valutazione funzionale. (A) Schema che mostra la valutazione funzionale di BRCA1. Poiché l’LOF di BRCA1 influisce sulla vitalità cellulare, quando la mutazione BRCA1 è patogena, le cellule muoiono all’aumentare del numero di passaggi. (B) Fasi della valutazione funzionale della BRCA1. La casella punteggiata è facoltativa. Può essere sostituito dalla co-trasfezione del GRNA che esprime e BE3 che esprime DNA di plasmidi. Clicca qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Questo protocollo descrive un metodo semplice per le valutazioni funzionali delle varianti BRCA1 utilizzando l’editor di base citosina meditato da CRISPR. Il protocollo descrive i metodi per la progettazione di gRNA al locus bersaglio e la costruzione dei DNA plasmidici da cui sono espressi. Gli editor di base citosina inducono la conversione nucleotidica in una finestra attiva (nel caso di BE3, nucleotidi 4-8 nell’estremità pam-distale delle sequenze bersaglio del gRNA). Il ricercatore dovrebbe scegliere atten…
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
Questo lavoro è stato sostenuto dalla National Research Foundation of Korea (sovvenzioni 2017M3A9B4062419, 2019R1F1A1057637 e 2018R1A5A2020732 a Y.K.).
Materials
| BamHI | NEB | R3136 | Restriction enzyme |
| Blasticidin | Thermo Fisher Scientific | A1113903 | Drug for selecting transduced cells |
| BsaI | NEB | R0535 | Restriction enzyme |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | Genomic DNA prep. kit |
| Dulbecco’s modified Eagle’s medium | Gibco | 11965092 | Medium for HEK293T/17 cells |
| Fetal bovine serum | Gibco | 16000036 | Supplemetal for cell culture |
| FuGENE HD Transfection Reagent | Promega | E2311 | Transfection reagent |
| Gibson Assembly Master Mix | NEB | E2611L | Gibson assembly kit |
| Iscove’s modified Dulbecco’s medium | Gibco | 12440046 | Medium for HAP1 cells |
| lentiCas9-Blast | Addgene | 52962 | Plasmids DNA for lentiBE3 cloning |
| Lipofectamine 2000 | Thermo Fisher Scientific | 11668027 | Transfection reagent |
| Opti-MEM | Gibco | 31985070 | Transfection materials |
| pCMV-BE3 | Addgene | 73021 | Plasmids DNA for lentiBE3 cloning |
| Penicillin-Streptomycin | Gibco | 15140 | Supplemetal for cell culture |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530SQ | High-fidelity polymerase |
| pMD2.G | Addgene | 12259 | Plasmids DNA for virus prep. |
| pRG2 | Addgene | 104174 | gRNA cloning vector |
| psPAX2 | Addgene | 12260 | Plasmids DNA for virus prep. |
| QIAprep Spin Miniprep kit | Qiagen | 27106 | Plasmid DNA prep. Kit |
| QIAquick Gel extraction Kit | Qiagen | 28704 | Gel extraction kit |
| QIAquick PCR Purification Kit | Qiagen | 28104 | PCR product prep. kit |
| Quick Ligation Kit | NEB | M2200 | Ligase for gRNA cloning |
| T7 Endonuclease I | NEB | M0302 | Materials for T7E1 assay |
| XbaI | NEB | R0145 | Restriction enzyme |
Referenzen
- Roy, R., Chun, J., Powell, S. N. BRCA1 and BRCA2: different roles in a common pathway of genome protection. Nature Reviews Cancer. 12 (1), 68-78 (2011).
- Kuchenbaecker, K. B., et al. Risks of Breast, Ovarian, and Contralateral Breast Cancer for BRCA1 and BRCA2 Mutation Carriers. Journal of the American Medical Association. 317 (23), 2402-2416 (2017).
- Millot, G. A., et al. A guide for functional analysis of BRCA1 variants of uncertain significance. Human Mutation. 33 (11), 1526-1537 (2012).
- Santos, C., et al. Pathogenicity evaluation of BRCA1 and BRCA2 unclassified variants identified in Portuguese breast/ovarian cancer families. Journal of Molecular Diagnostics. 16 (3), 324-334 (2014).
- Starita, L. M., et al. A Multiplex Homology-Directed DNA Repair Assay Reveals the Impact of More Than 1,000 BRCA1 Missense Substitution Variants on Protein Function. American Journal of Human Genetics. 103 (4), 498-508 (2018).
- Anantha, R. W., et al. Functional and mutational landscapes of BRCA1 for homology-directed repair and therapy resistance. Elife. 6, (2017).
- Gibson, T. J., Seiler, M., Veitia, R. A. The transience of transient overexpression. Nature Methods. 10 (8), 715-721 (2013).
- Quann, K., Jing, Y., Rigoutsos, I. Post-transcriptional regulation of BRCA1 through its coding sequence by the miR-15/107 group of miRNAs. Frontiers in Genetics. 6, 242 (2015).
- Saunus, J. M., et al. Posttranscriptional regulation of the breast cancer susceptibility gene BRCA1 by the RNA binding protein HuR. Krebsforschung. 68 (22), 9469-9478 (2008).
- Knott1, G. J., Doudna, J. A. CRISPR-Cas guides the future of genetic engineering. Science. 361, 866-869 (2018).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Hess, G. T., et al. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells. Nature Methods. 13 (12), 1036-1042 (2016).
- Kim, K., et al. Highly efficient RNA-guided base editing in mouse embryos. Nature Biotechnology. 35 (5), 435-437 (2017).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Park, D. S., et al. Targeted Base Editing via RNA-Guided Cytidine Deaminases in Xenopus laevis Embryos. Molecules and Cells. 40 (11), 823-827 (2017).
- Kweon, J., et al. A CRISPR-based base-editing screen for the functional assessment of BRCA1 variants. Oncogene. 39 (1), 30-35 (2020).
- Gibson, D. G. Enzymatic assembly of overlapping DNA fragments. Methods in Enzymology. 498, 349-361 (2011).
- Nageshwaran, S., et al. CRISPR Guide RNA Cloning for Mammalian Systems. Journal of Visualized Experiments. (140), (2018).
- Findlay, G. M., et al. Accurate classification of BRCA1 variants with saturation genome editing. Nature. 562 (7726), 217-222 (2018).
- Kweon, J., Kim, D. E., Jang, A. H., Kim, Y. CRISPR/Cas-based customization of pooled CRISPR libraries. PLoS One. 13 (6), 0199473 (2018).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nature Biotechnology. 31 (3), 251-258 (2013).
- Sayers, E. W., et al. GenBank. Nucleic Acids Research. 47 (1), 94-99 (2019).
- Hwang, G. H., et al. Web-based design and analysis tools for CRISPR base editing. BMC Bioinformatics. 19 (1), 542 (2018).
- Kim, D., Kim, D. E., Lee, G., Cho, S. I., Kim, J. S. Genome-wide target specificity of CRISPR RNA-guided adenine base editors. Nature Biotechnology. 37 (4), 430-435 (2019).
- Clement, K., et al. CRISPResso2 provides accurate and rapid genome editing sequence analysis. Nature Biotechnology. 37 (3), 224-226 (2019).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Walton, R. T., Christie, K. A., Whittaker, M. N., Kleinstiver, B. P. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science. , (2020).
- Kim, D., et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nature Biotechnology. 35 (5), 475-480 (2017).
- Zuo, E., et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science. 364 (6437), 289-292 (2019).
- Jin, S., et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science. 364 (6437), 292-295 (2019).
- Grunewald, J., et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature. 569 (7756), 433-437 (2019).
- Doman, J. L., Raguram, A., Newby, G. A., Liu, D. R. Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors. Nature Biotechnology. 38 (5), 620-628 (2020).
- Gaudelli, N. M., et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature. 551 (7681), 464-471 (2017).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).

