使用GAL4-UAS系统在冈比亚按蚊的功能遗传学中
Summary
二分GAL4-UAS系统是一种多功能工具,用于以受控的时空方式修饰基因表达,从而允许在 冈比亚按蚊中进行功能性遗传分析。使用该系统所描述的程序是半标准化的克隆策略,性别化和蛹荧光蛋白标志物的筛选以及胚胎固定。
Abstract
二分GAL4-UAS系统是功能性遗传分析的多功能且功能强大的工具。该系统的本质是穿越以组织特异性方式表达酵母转录因子GAL4的转基因”驱动”系,转基因”响应者”系携带候选基因/RNA干扰构建体,其表达由结合GAL4的上游激活序列(UAS)控制。在随后的后代中,基因或沉默构建体因此以规定的时空方式表达,从而能够测定所得的表型并推断基因功能。二元系统使实验方法能够灵活地筛选由多种组织特异性模式的转基因表达产生的表型,即使引起严重的适应性成本。我们针对非洲主要疟疾病媒冈 比亚按蚊调整了这一系统。
在本文中,我们提供了GAL4-UAS分析期间使用的一些常用过程。我们描述了已经生成的 An. gambiae GAL4-UAS谱系,以及用于上调和RNAi敲低的新响应器结构的克隆。我们指定了蚊子蛹的分步指南,以建立遗传杂交,其中还包括筛选后代以跟踪标记驾驶员和响应者插入的荧光基因标记的遗传。我们还提出了一个清除 冈比亚锥胚胎 以研究胚胎发育的协议。最后,我们介绍了该方法的潜在适应性,通过CRISPR / Cas9插入靶基因下游的GAL4来产生驱动系。
Introduction
二分GAL4-UAS系统是昆虫模式生物果蝇中基因功能表征的主力1,2,3。为了使用GAL4-UAS系统,在调节序列的控制下表达酵母转录因子GAL4的转基因驱动系与携带目的基因或RNA干扰(RNAi)构建体的响应系交叉,该响应系由GAL4识别的上游激活序列(UAS)控制。该杂交的后代以由控制GAL4表达的启动子决定的时空模式表达感兴趣的转基因(图1)。可以评估驾驶员 – 响应者杂交后代显示的表型,以阐明候选基因的功能。虽然D. melanogaster已被用于检查来自其他生物的基因4,5,6,7,但GAL4-UAS系统现已适应用于具有医学和农业重要性的昆虫,以提供对感兴趣的物种的直接分析8,9,10,11,12,13,14。
在非洲疟疾蚊子冈比亚按蚊中,GAL4-UAS系统首先通过细胞系共转染9进行测试。对多种结构在不同的成对组合中测定了效率,发现当与GAL4驱动器面板一起使用时,14个串联重复的UAS补充了一个小的人造内含子(UAS-14i),显示出最宽的活化电位。为了证明体内功能,然后使用这些构建体通过PiggyBac转化8创建两个单独的转基因冈比亚锥系:一个由中肠特异性启动子驱动的携带GAL4的驱动系,以及一个在UAS序列调节下含有荧光素酶和增强型黄色荧光蛋白(eYFP)基因的响应系。肠道特异性荧光素酶活性和后代荧光表明该系统在按蚊中是有效的。从那时起,已经创建了驱动系,以接近无处不在的模式10表达对载体能力和杀虫剂耐药性很重要的其他组织中表达转基因。还产生了许多UAS系,以测定被认为参与代谢和封存介导的杀虫剂耐药性,角质层碳氢化合物合成以及荧光标记不同细胞和组织类型的基因(表1)。对于响应者系,现在通过ΦC31催化的重组盒交换17,18进行转基因的位点导向整合,以固定UAS调节基因的基因组上下文。通过这种方式,转基因表达在基因组插入位置方面被归一化,从而可以更准确地比较不同候选基因的表型效应。
迄今为止创造的响应细胞系被设计为在升高的水平上表达转基因或通过RNA干扰(RNAi)减少基因表达。 通常,cDNA克隆被融合到UAS序列中以产生合适的表达质粒,但是假设它们对于克隆来说不太大,完整的基因组序列也是可行的。为了产生沉默构建体,我们使用三种不同的方法来获得合适的串联倒置序列,这些序列形成刺激RNAi的发夹dsRNA。这些包括融合PCR,不对称PCR和发夹构建体的商业合成。每种方法的共同点是在倒置序列之间包含一个内含子序列,以提供克隆稳定性。已经开发出可插入目的基因/RNAi构建体的应答质粒15。这些质粒还携带RMCE所需的ΦC31 attB 位点(在Adolfi随附的JoVE论文中详细描述了RCME技术)。本文包含了在选择插入这些质粒之一进行过表达时所需的重要步骤的实验方案。此外,还描述了并说明了用于RNAi发夹构建体创建的两种方案。
在创建新品系时,鉴定稀有转基因个体对于繁殖以建立和维持转基因菌落至关重要。最重要的是,对于GAL4-UAS系统,有必要区分响应者和驾驶员线,以建立交叉并识别携带两种转基因的个体后代。这是通过使用与驱动和应答盒相连的不同显性可选择标记基因来实现的。最常见的是荧光标记基因,使用滤光片(例如,eYFP,eCFP,dsRed)可以清楚地区分。重要的是,标记物以已知且可靠的时空模式表达,因为这可以更容易地识别异常和污染。荧光标记基因表达通常由合成的 3xP3 启动子调节,其在 冈比亚锥发育 的所有阶段引起眼睛和腹侧神经节特异性表达19。由 3xP3 控制的荧光标记物包含在本文描述的所有转化质粒中。本文详细介绍了用于筛选荧光 冈比亚蛹 GAL4-UAS线的常用方法的方案。
GAL4-UAS系统的关键要素之一是必须跨越差分标记的驾驶员和响应者线。为此,每条线的雄性和雌性必须在交配前分开。成虫很容易通过视觉来区分,然而,为了建立遗传杂交,在成虫出现之前将性别分开是明智的,以确保没有发生交配。雄性和雌性 冈比亚蛹 之间的一般大小差异变化太大,无法成为有效和可靠的性别确定方法20。相反,外生殖器的明显形态差异为 冈比亚羚的性行为提供了可靠的基础。在本文中,我们描述了一种可靠的方法,用于对 冈比亚蛹 进行,以建立适当的杂交。
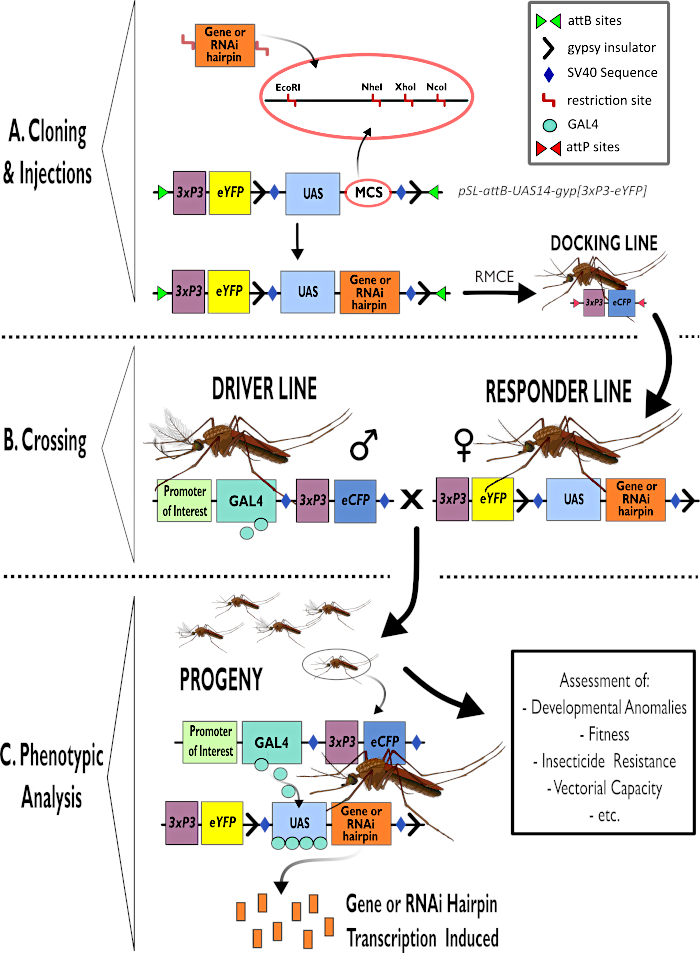
图1 -在冈比亚按蚊中使用二分GAL4-UAS系统的过程示意图。 (A)描述了示例载体(pSL-attB-UAS14-gyp[3xp3-eYFP])的主要成分,详细说明了多个克隆位点中可用于插入目标基因的发夹构建体或编码序列的可用限制位点(EcoRI,NheI,XhoI和NcoI)。还描绘了对接线的结构。(B)交叉步骤图示了来自驱动线的雄性(由感兴趣的启动子携带GAL4驱动并由3xP3启动子驱动的eCFP)和来自响应线的雌性(携带由UAS启动子控制的兴趣基因或发夹构建体和由3xP3启动子控制的eYFP标记)。(C)GAL4驱动B中杂交后代中目的基因表达的图表表示,以及评估的一些典型表型的列表。缩写:多克隆位点(MCS),重组酶介导的盒交换(RMCE),上游激活序列(UAS),增强型黄色荧光蛋白(eYFP),增强型青色荧光蛋白(eCFP)。 请点击此处查看此图的放大版本。
正是交叉的使用提供了GAL4-UAS系统的二分性质,与更线性的方法相比,它具有明显的优势。例如,可以评估的驱动和响应系组合比必须为每个启动子/基因组合生成和维护新的转基因系时可行得多。更重要的是,它允许分析当其表达受到干扰时产生致命或无菌表型的基因,这些基因在线性系统中难以创建/维持。这种致命的表型可以在所有发育阶段表现出来,这取决于基因功能和时空表达,但最常在胚胎发育过程中观察到。可视化蚊子胚胎发育需要清除覆盖卵子的不透明绒毛膜。按照Trpiš(1970)21 和Kaiser等人(2014)22中描述的方法,我们描述了我们用于固定胚胎的方案,同时保持结构完整性,并漂白以清除允许微观可视化和成像的内脏。
Protocol
Representative Results
Discussion
了解蚊子的基因功能对于开发控制按蚊和影响疟疾传播的新方法至关重要。所描述的GAL4-UAS系统是一种多功能且功能强大的系统,用于候选基因的功能分析,迄今为止,我们已使用该系统来检查杀虫剂抗性17和角质层碳氢化合物生产的遗传基础15,23,以及荧光标记不同的蚊子细胞群8.到目前为止,已经通过限制?…
Divulgaciones
The authors have nothing to disclose.
Acknowledgements
我们非常感谢LSTM和IVCC(Adriana Adolfi),BBSRC(新研究员奖(AL),MRC(BCP:MR / P016197 / 1博士生奖学金),Wellcome(Henry Wellcome爵士博士后奖学金LG:215894 / Z / 19 / Z)的资助,这些资金已将Gal4UAS分析纳入提案中。
Materials
| 100 x 15 mm plastic Petri dish | SLS | 2175546 | Pack of 10 |
| 1000 µL Gilson Pipette | Gilson | F144059P | |
| 20/25 mL Universal Tubes | Starlab | E1412-3020 | Pack of 400 |
| 3 mL Pasteur Pipettes | SLS | G612398 | Greiner Pasteur pipette 3 mL sterile individually wrapped |
| 50 mL Falcon Tubes | Fisher Scientific | 11512303 | |
| Absolute Ethanol | Fisher Scientific | BP2818-500 | 500 mL |
| Acetic Acid | SLS | 45726-1L-F | 1 L |
| Cages | SLS | E6099 | 30x30x30 with screen port |
| Fine Paint Brushes | Amazon | UKDPB66 | KOLAMOON 9 Pieces Detail Painting Brush Set Miniture Brushes for Watercolor, Acrylic Painting, Oil Painting (Wine Red) |
| Fish food | Amazon | Tetra Min Fish Food, Complete Food for All Tropical Fish for Health, Colour and Vitality, 10 L | |
| Formaldehyde Solution | Sigma Aldrich | F8775 | |
| Mouth Aspirator | John Hock | 612 | |
| Pond Salt | Amazon | Blagdon Guardian Pond Tonic Salt, for Fish Health, Water Quality, General Tonic, pH Buffer, 9.08 kg, treats 9,092 L | |
| Pupae Pots | Cater4you | SP8OZ | 250 pots with lids |
| Small Plastic Buckets | Amazon | 2.5 L White Plastic Pail Complete with White Lid (Pack of 10) | |
| Sodium Hypochlorite | Fisher Scientific | S25552 |
Referencias
- Brand, A. H., Perimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118 (2), 401-415 (1993).
- Duffy, J. B. GAL4 system in drosophila: A fly geneticist’s swiss army knife. Journal of Genetics and Development. 34 (1-2), 1-15 (2002).
- Dow, J. A. . ELS. , (2012).
- Edi, C. V., et al. CYP6 P450 Enzymes and ACE-1 Duplication Produce Extreme and Multiple Insecticide Resistance in the Malaria Mosquito Anopheles gambiae. PLoS Genetics. 10 (3), 1004236 (2014).
- Daborn, P. J., et al. Using Drosophila melanogaster to validate metabolism-based insecticide resistance from insect pests. Insect Biochemistry and Molecular Biology. 42 (12), 918-924 (2012).
- Riveron, J. M., et al. Genome-wide transcription and functional analyses reveal heterogeneous molecular mechanisms driving pyrethroids resistance in the major malaria vector Anopheles funestus across Africa. Genes Genomes Genetics. 7 (6), 1819-1832 (2017).
- Riveron, J. M., et al. A single mutation in the GSTe2 gene allows tracking of metabolically based insecticide resistance in a major malaria vector. Genome Biology. 15 (2), (2014).
- Lynd, A., Lycett, G. J. Development of the Bi-Partite Gal4-UAS System in the African Malaria Mosquito, Anopheles gambiae. PLoS ONE. 7 (2), 31552 (2012).
- Lynd, A., Lycett, G. J. Optimization of the Gal4-UAS system in an Anopheles gambiae cell line. Insect Molecular Biology. 20 (5), 599-608 (2011).
- Adolfi, A., Pondeville, E., Lynd, A., Bourgouin, C., Lycett, G. J. Multi-tissue GAL4-mediated gene expression in all Anopheles gambiae life stages using an endogenous polyubiquitin promoter. Insect Biochemistry and Molecular Biology. 96, 1-9 (2018).
- Kokoza, V. A., Raikhel, A. A. Targeted gene expression in the transgenic Aedes aegypti using the binary Gal4-UAS system. Insect Biochemistry and Molecular Biology. 41, 637-644 (2011).
- O’Brochta, D. A., Pilitt, K. L., Harrell, R. A., Aluvihare, C., Alford, R. T. Gal4-based Enhancer-Trapping in the Malaria Mosquito Anopheles stephensi. Genes Genomes Genetics. 2, 21305-21315 (2012).
- Zhao, B., et al. Regulation of the Gut-Specific Carboxypeptidase: A Study Using the Binary Gal4/UAS System in the Mosquito Aedes Aegypti. Insect Biochemistry and Molecular Biology. 54, 1-10 (2014).
- Imamura, M., et al. Targeted Gene Expression Using the GAL4/UAS System in the Silkworm Bombyx mori. Genética. 165 (3), 1329-1340 (2003).
- Lynd, A., et al. Development of a functional genetic tool for Anopheles gambiae oenocyte characterisation: application to cuticular hydrocarbon synthesis. bioRxiv. , (2019).
- Pondeville, E., et al. Hemocyte-targeted gene expression in the female malaria mosquito using the hemolectin promoter from Drosophila. Insect Biochemistry and Molecular Biology. 120, 103339 (2020).
- Adolfi, A., et al. Functional genetic validation of key genes conferring insecticide resistance in the major African malaria vector, Anopheles gambiae. Proceedings of the National Academy of Sciences of the United States of America. 116 (51), 25764-25772 (2019).
- Pondeville, E., et al. Efficient integrase-mediated site-specific germline transformation of Anopheles gambiae. Nature Protocols. 9 (7), 1698-1712 (2014).
- Horn, C., Schmid, B. G. M., Pogoda, F. S., Wimmer, E. A. Fluorescent transformation markers for insect transgenesis. Insect Biochemistry and Molecular Biology. 32, 1221-1235 (2002).
- Clements, A. . A. Biology of Mosquitoes, Volume 1: Development, Nutrition and Reproduction. 1, (1992).
- Trpiš, M. A new bleaching and decalcifying method for general use in zoology. Canadian Journal of Zoology. 48, 892-893 (1970).
- Kaiser, M. L., Duncan, F. D., Brooke, B. D. Embryonic Development and Rates of Metabolic Activity in Early and Late Hatching Eggs of the Major Malaria Vector Anopheles gambiae. PLoS ONE. 9 (12), 114381 (2014).
- Grigoraki, L., Grau-Bové, X., Yates, H. C., Lycett, G. J., Ranson, H. Isolation and transcriptomic analysis of Anopheles gambiae oenocytes enables the delineation of hydrocarbon biosynthesis. eLife. 9, 58019 (2020).
- Xiao, Y. -. H., Yin, M. -. H., Hou, L., Pei, Y. Direct amplification of intron-containing hairpin RNA construct from genomic DNA. BioTechniques. 41 (5), 548-552 (2006).
- Livak, K. J. Organization and Mapping of a Sequence on the Drosophila melanogaster X and Y Chromosomes That Is Transcribed during Spermatogenesis. Genética. 107 (4), 611-634 (1984).
- MR4, CDC, NEI & beiResources. . The MR4 Methods in Anopheles Research Laboratory Manual. 5th Edition. , (2015).
- Sik Lee, Y., Carthew, R. W. Making a better RNAi vector for Drosophila: use of intron spacers. Methods. 30 (4), 322-329 (2003).
- Cha-aim, K., Hoshida, H., Fukunaga, T., Akada, R., Peccoud, J. . Gene Synthesis: Methods and Protocols. , 97-110 (2012).
- Cavener, D. R. Comparison of the consensus sequence flanking translational start sites in Drosophila and vertebrates. Nucleic Acids Research. 15 (4), 1353-1361 (1987).
- Wang, Y., Wang, F., Wang, R., Zhao, P., Xia, Q. 2A self-cleaving peptide-based multi-gene expression system in the silkworm Bombyx mori. Scientific Reports. 5, (2015).
- Galizi, R., et al. A synthetic sex ratio distortion system for the control of the human malaria mosquito. Nature Communications. 5, 3977 (2014).
- Kondo, S., et al. Neurochemical organisation of the Drosophila Brain Visualised by Endogenously Tagged Neurotransmitter Receptors. Cell Reports. 30 (1), 284-297 (2020).
- Lee, P. -. T., et al. A gene-specific T2A-GAL4 library for Drosophila. eLife. 7, 35574 (2018).
- Marois, E., et al. High-throughput sorting of mosquito larvae for laboratory studies and for future vector control interventions. Malaria Journal. 11, 302 (2012).
- Crawford, J. E., et al. Efficient production of male Wolbachia-infected Aedes aegypti mosquitoes enables large-scale suppression of wild populations. Nature Biotechnology. 38 (4), 482-492 (2020).
- Goltsev, Y., et al. Developmental and evolutionary basis for drought tolerance of the Anopheles gambiae embryo. Biología del desarrollo. 330 (2), 462-470 (2009).
- Rezende, G. L., et al. Embryonic desiccation resistance in Aedes aegypti: presumptive role of the chitinized Serosal Cuticle. BMC Developmental Biology. 8 (1), 82 (2008).
- Vargas, H. C. M., Farnesi, L. C., Martins, A. J., Valle, D., Rezende, G. L. Serosal cuticle formation and distinct degrees of desiccation resistance in embryos of the mosquito vectors Aedes aegypti, Anopheles aquasalis and Culex quinquefasciatus. Journal of Insect Physiology. 62, 54-60 (2014).
- Chang, C. -. H., et al. The non-canonical Notch signaling is essential for the control of fertility in Aedes aegypti. PLOS Neglected Tropical Diseases. 12 (3), 0006307 (2018).
- Clemons, A., Flannery, E., Kast, K., Severson, D., Duman-Scheel, M. Immunohistochemical Analysis of Protein Expression during Aedes aegypti Development. Spring Harbor Protocols. 10, 1-4 (2010).
- Juhn, J., James, A. A. Hybridization in situ of Salivary Glands, Ovaries and Embryos of Vector Mosquitoes. Journal of Visualized Experiments. , e3709 (2012).

