사례 연구로 담배에 과도 단백질 발현 : 실험 접근의 디자인을 사용하여 복잡한 시스템의 특성
Summary
우리는 식물에서 단일 클론 항체와 리포터 단백질의 과도 발현 유전자 조절 요소, 식물의 성장과 발달 파라미터 및 배양 조건의 영향을 결정하고 모델링하는 데 사용될 수있는 실험 방법의 설계를 설명한다.
Abstract
식물은 낮은 비용, 확장 성 및 안전성을 포함한 생물 약제의 제조를위한 다수의 이점을 제공한다. 일시적 발현은 짧은 개발 및 생산 시간의 추가적인 장점을 제공하지만, 발현 따라서 좋은 제조 연습의 맥락에서 규제 우려로 상승을주는 일괄 처리 사이에 상당히 다를 수 있습니다. 우리는 일괄 간의 발현의 변동에 대한식이 동안 규정 식 구조 요소, 식물의 성장과 발달 파라미터 및 배양 조건 등 주요 요인의 영향을 결정하기 위하여 실험 (DOE) 접근법의 디자인을 사용했다. 우리는 모델 항 HIV 단일 클론 항체 (2G12)과 형광 마커 단백질 (DsRed를)을 표현하는 식물을 시험했다. 우리는 모델의 특정 속성을 선택하기위한 이론적 근거를 설명하고 잠재적 한계를 식별합니다. 일반적인 방법은 간단하게 다른 문제로 전송 될 수 있기 때문에 모델의 원리광범위하게 다시 적용 : 작은 모듈, 최적의 실험 조합의 소프트웨어 가이드 설정 및 단계적 디자인 증가로 초기 문제를 분할하여 지식 기반 파라미터 선택, 복잡성 감소. 따라서, 방법론뿐만 아니라 식물의 단백질 발현을 특성화뿐만 아니라 기계적인 설명이 부족한 다른 복잡한 시스템의 조사에 유용합니다. 파라미터 사이의 상호 접속을 설명하는 예측 식들은 다른 복잡한 기계적 시스템 모델을 확립하는데 사용될 수있다.
Introduction
식물이 성장 저렴하기 때문에 식물에서 바이오 의약품 단백질의 생산이 유리하다, 플랫폼은 더 많은 식물을 재배하여 확장 할 수 있으며, 인간의 병원체는 1,2를 복제 할 수 없습니다. DNA 전달 및 정제 된 제품의 배달 지점 사이의 시간을 세 미만 2 달 이상 3로 감소하기 때문에 아그로 박테 리움 튜메 파시 엔스 잎의 침투에 예를 들어 기반의 일시적 발현 전략은 추가 혜택을 제공한다. 일시적 발현은 기능 상실 돌연변이를 보완하기 위해 또는 단백질 상호 작용 4-6을 조사하기 위해 자신의 능력에 대한 유전자를 테스트하는 기능 분석, 예를 들어 사용됩니다. 그러나, 일시적인 발현은 형질 전환 식물체 7-9의 발현 수준보다 큰 배치 별 편차를 표시하는 경향이있다. 이는 바이오 의약품의 제조 공정은 일시적인 발현 위스콘신에 근거한다는 가능성을 줄여재현성이 중요한 품질 특성과 위험 평가 10 될 수 있으므로 우수 제조 기준 (GMP)의 맥락에서 승인 될 것이다. 이러한 변화는 연구자가 조사하고자하는 모든 상호 작용에 마스크를 적용 할 수 있습니다. 따라서 식물에서 과도 발현 수준에 영향을 고품질 정량적 예측 모델을 구축하는 주요 요인의 동정을 설정.
하나의 요인 -에서 – 타임 (OFAT) 접근 방식은 종종 실험 11의 결과에 대한 특정 매개 변수 (요인)의 영향 (효과) (응답)를 특성화하는 데 사용됩니다. 연구 (실험) 동안 개별 테스트 (실행) (설계 공간)을 테스트하는 요인에 의해 스팬 잠재적 인 영역을 통해 문자열에 진주처럼 정렬됩니다 때문에이 차선이다. 디자인 공간과 실험에서 파생 된 정보 따라서 정도의 범위는낮은,도 1a에 도시 된 바와 같이 12. 도 1b 13에 도시 된 바와 같이 또 다른 요인 (인자의 상호 작용) 사이의 상호 의존성은별로 모델 및 / 또는 거짓 옵티마의 예측 결과 은폐 남아있을 수있다.
전술 한 단점은 둘 이상의 요소가이 실행 (14) 사이에 변화되는 것을 의미하는 실험의 실행이 디자인 공간에 걸쳐 균등하게 분산되어있는 실험 (DOE) 방식의 설계를 사용하여 회피 할 수있다. 요인 (요인 설계) 및 반응에 인자에 미치는 영향의 정량 (반응 표면 방법, RSM 초) 15를 선별 혼합물에 대한 전문적인 디자인이있다. 또한, RSM의 중부 – 복합체 설계로 실현 될 수 있지만, 또한 실행의 선택을위한 다른 기준을 적용 할 수있는 특수한 소프트웨어를 사용하여 효과적으로 성취 될 수있다. 예를 들어, 소위 D-optimalitY 기준은 IV-최적 성 기준은 설계 공간 (15, 16)에 걸쳐 가장 낮은 예측 분산을 달성 실행을 선택하는 반면, 이렇게 생성 된 모델의 계수들에서 오류를 최소화하기 위해 실행을 선택한다. 우리는 여기 설명 RSM은 식물에서 과도 단백질 발현의 정확한 정량을 허용하지만, 간단하게 (5-8 ~) 몇개 관련된 모든 시스템으로 전송 될 수있는 숫자 요인 (예 : 온도, 시간, 농도) 및 소수 (~ 2 – 4) 기계 론적 설명은 모델에 사용할 수 없거나 너무 복잡 categoric 요인 (예를 들어, 프로모터, 색상)하는.
피해자의 접근 방식은 농업 과학에서 유래하지만, 그것이 신뢰할 수있는 데이터를 확보하는 데 필요한 실행의 수를 줄이고 복잡한 프로세스에 대한 설명이 포함 된 모델을 생성하는 데 유용합니다 어떤 상황에 양도 할 수 있기 때문에 다른 지역으로 확산하고있다. 이것은 차례로은 "지침에서 미상의 포함되었다산업은 인간의 사용에 대한 제약 (ICH) (17)의 등록을위한 기술 요구 사항의 조화에 관한 국제 회의에서 발표 Q8 (R2) 의약품 개발은 ". 미상은 현재 과학 연구 및 산업 (18)에 널리 사용된다. 그러나, 치료 중에주의해야 다중 선형 회귀 모델 (기본 모델)에 대한 부적절한 다항식 정도를 선택하기 때문에 계획과 실험의 실행은 제대로 모든 인자의 영향을 모델링하는 추가 실행을위한 필요를 소개 할 수있다. 또한, 손상되거나 누락 된 데이터가 잘못된 모델과 결함을 생성 예측, 심지어 프로토콜 및 토론 섹션 (18)에서 설명한 바와 같이 임의의 모형 구축 시도를 방지 할 수있다. 프로토콜 섹션에서, 우리는 처음 RSM 기반 실험을위한 가장 중요한 계획 단계를 설정 한 후 미상에 기반 디자인을 설명 할 것이다 소프트웨어 DesignExpert v8.1이. 그러나 유사한 디자인은 다른 소프트웨어 includi을 빌드 할 수 있습니다NG JMP, Modde 및 STATISTICA. 실험 절차는 데이터 분석 및 평가를위한 지침에 따른다.
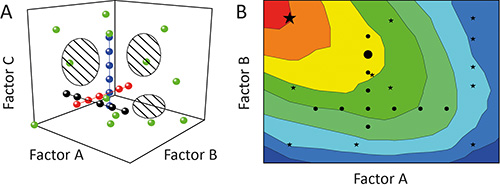
그림 1. OFAT 및 미상.의 비교. 실험 (검은 색, 빨간색과 파란색 동그라미)의 시간 (OFAT) 하나의 인자의 연속 변화는 설계 공간 (빗금 친 지역)의 낮은 범위를 달성 할 수있다. 대조적으로, 실험 (DOE) 전략 (녹색 원)의 디자인을 사용하여 한 번에 하나 이상의 인자의 변형에 따르면 얻어진 모델. 따라서 B의 정밀도를 향상시킨다. 바이어스 설계 공간의 범위는 OFAT 실험 (검은 색 원)도, 최적의 작업 영역 (빨간색)를 식별 및 하위 최적의 솔루션 (큰 검은 원)을 예측하는 데 실패 할 수 있다는 것을 의미하는 반면 피해자의 strategiES (검은 별) 바람직 조건 (큰 검은 별)를 식별 할 가능성이 높습니다.
Protocol
Representative Results
Discussion
자원은 종종 부족하고 비용 때문에 모든 실험은 신중한 계획이 필요합니다. 계획 단계 (예를 들어, 모든 중요한 요소의 상호 작용을 포함하지 않는 기본 모델을 선택)하는 동안 오류가 실질적 결과 모델의 예측 능력을 감소함으로써 전체 실험을 평가 절하 할 수 있기 때문 미상 전략에 특히 사실이다. 그러나 이러한 오류는 쉽게 기본 절차에 따라 피할 수 있습니다.
미상 …
Divulgations
The authors have nothing to disclose.
Acknowledgements
저자는이 연구에서 사용 된 담배 식물을 재배의 pPAM 식물 발현 벡터 및 이브라힘 알 Amedi를 제공하는 토마스 박사 Rademacher는에게 감사의 말씀을 전합니다. 우리는 원고를 편집하는 그의 도움 박사 리처드 M. 트 와이에게 감사의 말씀을 전합니다. 이 작품은 부분적으로 유럽 연구위원회 고급 그랜트 "미래 제약"에 의해 추진, 제안 번호 269110과 프라운호퍼 Zukunftsstiftung (프라운호퍼 미래 재단).
Materials
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10x10cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
References
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ – The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5’UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).

