L'élevage et d'ARN double brin médiée Gene Knockdown dans le Hide Beetle,<em> Dermestes maculatus</em
Summary
Ici, nous présentons des protocoles d'élevage un coléoptère intermédiaire germe, Dermestes maculatus (D. maculatus) dans le laboratoire. Nous partageons également des protocoles pour l'embryon et des parents ARNi et méthodes pour l'analyse des phénotypes embryonnaires pour étudier la fonction des gènes de cette espèce.
Abstract
Advances in genomics have raised the possibility of probing biodiversity at an unprecedented scale. However, sequence alone will not be informative without tools to study gene function. The development and sharing of detailed protocols for the establishment of new model systems in laboratories, and for tools to carry out functional studies, is thus crucial for leveraging the power of genomics. Coleoptera (beetles) are the largest clade of insects and occupy virtually all types of habitats on the planet. In addition to providing ideal models for fundamental research, studies of beetles can have impacts on pest control as they are often pests of households, agriculture, and food industries. Detailed protocols for rearing and maintenance of D. maculatus laboratory colonies and for carrying out dsRNA-mediated interference in D. maculatus are presented. Both embryonic and parental RNAi procedures-including apparatus set up, preparation, injection, and post-injection recovery-are described. Methods are also presented for analyzing embryonic phenotypes, including viability, patterning defects in hatched larvae, and cuticle preparations for unhatched larvae. These assays, together with in situ hybridization and immunostaining for molecular markers, make D. maculatus an accessible model system for basic and applied research. They further provide useful information for establishing procedures in other emerging insect model systems.
Introduction
En 1998, le feu et Mello ont rapporté que l' ARN double brin (ARNdb) peut induire l' inhibition de la fonction des gènes dans Caenorhabditis elegans 1. Cette réponse déclenchée par l' ARN double brin a été appelé ARN interférence (ARNi), et un tel silençage génique à médiation par ARNi a été rapportée être conservée chez les animaux, les plantes et les champignons de 2-7. Chez les plantes et certains animaux, les fonctions ARNi systémique, ce qui signifie que l'effet peut se propager à d' autres cellules / tissus où ARNdb est pas directement introduit (revue dans 8-10). Les scientifiques ont fait usage de cette réponse ARNi cellulaire endogène en concevant ARNdb pour cibler des gènes d'intérêt, ce qui frappe vers le bas la fonction du gène sans manipuler directement le génome (revue dans 11-14).
L'ARNi est un outil puissant pour des études fonctionnelles en raison des avantages suivants. Tout d'abord, même avec des informations sur la séquence du gène minimum, un gène peut être ciblé en utilisant l'ARNi. Ceci est particulièrement important pour les rudies d'organismes non-modèles dépourvus de données génomiques ou transcriptomiques. D'autre part, dans les organismes où la réponse ARNi est robuste systémique, knock-down de gènes à médiation par ARNi peut être réalisée à pratiquement n'importe quel stade de développement. Cette fonctionnalité est très utile pour étudier la fonction des gènes pléiotropiques. Troisièmement, dans certains cas, les effets ARNi se propagent aux gonades et descendants, tels que les phénotypes sont observés chez la progéniture 15,16. Ce phénomène, connu sous le nom ARNi parental (pRNAi), est particulièrement avantageux pour les gènes impact sur le développement embryonnaire, aussi nombreux descendants produits par un seul parent injecté peut être examinée sans manipulation directe des œufs. Pour ces raisons, pRNAi est la méthode de choix. Toutefois, si pRNAi est inefficace, par exemple pour les gènes nécessaires à l'ovogenèse, puis embryonnaires ARNi (eRNAi) doit être utilisé. Quatrièmement, l'ARNi peut être utilisé pour générer l'équivalent d'une série allélique en ce que la quantité d'ARNdb livré peut varier sur une plage pour produire faible pour les défauts forts. Une telle gradation des phénotypes peut être utile pour la compréhension de la fonction des gènes lorsque le gène est impliqué dans un processus complexe et / ou la perte complète de la fonction est létale. Cinquièmement, la livraison d'ARNdb est généralement facile et réalisable, en particulier chez les animaux montrant robustes réponses systémiques ARNi. ARNdb peut être introduit par microinjection 1,5, l' alimentation / l' ingestion 17,18, 19,20 trempage et le virus de la livraison / bactéries médiée 21,22. Sixième, à la différence des méthodes de ciblage / d'édition de gènes, il n'y a pas besoin de dépister les organismes porteurs de la mutation ou de procéder à des croisements génétiques pour générer des homozygotes lors de l'utilisation d'ARNi. Par conséquent, par rapport à de nombreuses autres techniques pour étudier la fonction des gènes, l' ARNi est rapide, peu coûteuse et peut être appliqué pour les écrans de grande envergure 23-25.
La grande utilité de l'ARNi fournit des moyens de réaliser des études fonctionnelles dans un large éventail d'organismes, en élargissant la gamme des espèces disponibles pour l'étude beyond les systèmes modèles traditionnels pour lesquels des outils génétiques ont été développés. Par exemple, des études utilisant des systèmes non-modèle sont tenus de donner un aperçu de l'évolution des gènes et des réseaux de gènes en comparant les fonctions de orthologues d'espèces représentant différents modes de développement ou présentant des caractéristiques morphologiques distinctes 26-29. Ces types d'études fourniront une meilleure compréhension de la diversité biologique, avec des impacts de la recherche à la fois fondamentale et appliquée.
Étant le plus grand groupe d'animaux sur la planète, les insectes fournissent une excellente occasion d'explorer les mécanismes de la diversité sous-jacente. En outre, les insectes sont généralement de petite taille, ont des cycles de vie courts, une fécondité élevée, et sont faciles à élever en laboratoire. Au cours des deux dernières décennies, l' ARNi a été appliquée avec succès dans les insectes couvrant les commandes, y compris les diptères (mouches vraies) 5, lépidoptères (papillons et mites) 30, coléoptères (scarabées) 16,31, hyménoptères (Sawfmensonges, les guêpes, les fourmis et les abeilles) 32, Hemiptera (punaises), isoptères (termites) 34, Blattodea (cafards) 35, orthoptères (grillons, sauterelles, criquets et sauterelles) 36 et Phthiraptera (poux) 37. L' application réussie de l' ARNi a fourni des données fonctionnelles pour les études de structuration dans l' embryogenèse précoce (antéro-postérieur axe 32, dorso-ventral axe 28, segmentation 26,38), la détermination du sexe 39,40, chitine / cuticule biosynthèse 41, ecdysone de signalisation 42, comportement social 43, et plus encore. Méthodes d' ARNi développées pour différentes espèces d'insectes peuvent être d'avantage supplémentaire en ce sens qu'elles sont susceptibles d'être utiles pour la lutte antiparasitaire (passé en revue dans 44-46). les effets d'ARNi seront aussi bien que spécifique de l'espèce spécifique du gène, aussi longtemps que les régions non conservées sont choisies pour le ciblage. Pour les espèces d'insectes bénéfiques comme les abeilles et les vers à soie, en ciblant les gènes vitaux pour la survie devirus ou des parasites pour contrôler l' infection peuvent fournir une nouvelle stratégie visant à protéger ces espèces 47,48.
Dermestes maculatus (D. maculatus), nom commun hide scarabée, est distribué dans le monde entier à l' exception de l' Antarctique. Comme un insecte holométabole, le cycle de vie D. maculatus comprend embryonnaire, larvaire, pupe et stades adultes (Figure 1). Parce qu'il se nourrit de chair, D. maculatus est utilisé dans les musées à squelettage animaux morts et entomologistes médico – légale peut l' utiliser pour estimer le temps de la mort 49,50. D. maculatus se nourrit de produits d'origine animale , y compris les carcasses, la viande séchée, du fromage et des pupes / cocons d'autres insectes et provoque ainsi des dommages aux ménages, les aliments stockés, et la soie, le fromage et les industries de la viande 51,52. L'application de l'ARNi dans ce coléoptère pourrait fournir un moyen efficace et respectueux de l'environnement afin de minimiser son impact économique. Notre laboratoire a utilisé D. maculatus comme une nouvelle model insectes pour étudier la segmentation 53. En plus d'être prête à l' élevage de laboratoire, D. maculatus est d'intérêt pour la recherche fondamentale car elle est un développeur intermédiaire germe, ce qui en fait une espèce utile pour étudier la transition entre court et le développement à long germe.
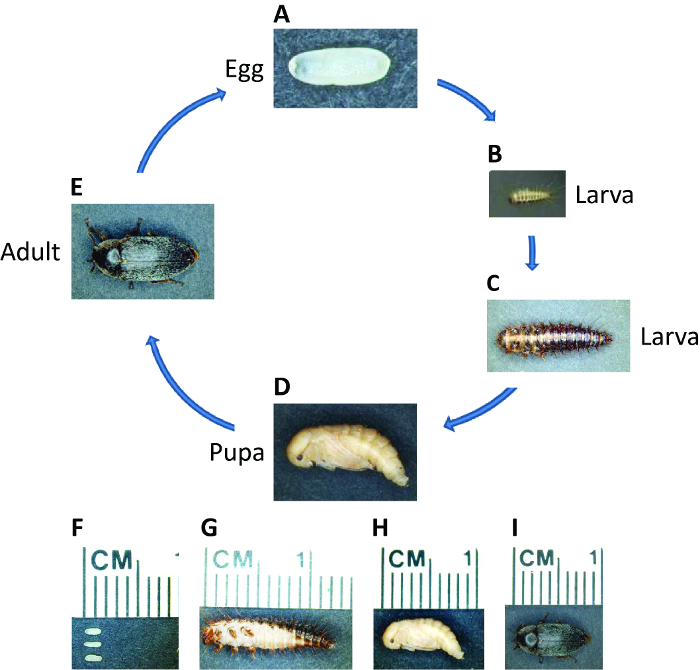
Figure 1: Cycle de vie de D. maculatus. Photographies de D. maculatus à différents stades de la vie, comme indiqué. Le cycle de vie de l'œuf à l'adulte dure trois semaines à 30 ° C mais plus à des températures plus basses. (A, F) des embryons fraîchement pondus sont blancs à jaune clair et ovales, d' environ 1,5 mm de longueur. Embryogenèse prend ~ 55 heures à 30 ° C. (B, C et G) Larves ont des rayures pigmentées sombres et sont recouverts de soies. Les larves passent par plusieurs stades en fonction de l'environnement et leur longueur peut aller jusqu'à plus de 1 cm. (D, H) </strong> Jeune pupes sont jaune clair. La nymphose a ~ 5 – 7 jours à 30 ° C. (E, I) Peu de temps après l' éclosion, la pigmentation foncée apparaît sur le corps de coléoptère adulte. Les adultes peuvent vivre jusqu'à plusieurs mois et une femelle peut pondre des centaines d'embryons au cours de sa vie. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Auparavant, nous avons montré que l' ARNi est efficace pour abattre la fonction du gène dans D. maculatus 53. Voici notre expérience d' élevage colonies D. maculatus dans le laboratoire est partagé avec les protocoles étape par étape pour RNAi set-up à la fois embryonnaire et parental, l' injection, les soins post-injection, et l' analyse phénotypique. Les knockdown et méthodes d' analyse de gènes ARNdb médiées introduits ici non seulement fournir des informations détaillées pour aborder les questions de D. maculatus, mais aussi avoir une signification potentielle for l'application de l'ARNi dans d'autres espèces d'insectes / non-modèle coléoptères.
Protocol
Representative Results
Discussion
Bien qu'un petit nombre de systèmes modèles sophistiqués (souris, les mouches, les vers) ont été développés au cours du 20 e siècle, le 21ème siècle a vu une vague de nouveaux systèmes d'animaux en cours de développement dans les laboratoires à travers le monde. Ces nouveaux systèmes permettent aux scientifiques d'aborder comparatifs, des questions d'évolution qui ne peut être sondé en utilisant uniquement les systèmes modèles «standard». Ce déploiement de nouv…
Divulgations
The authors have nothing to disclose.
Acknowledgements
We thank Drs. Alison Heffer and Yong Lu for setting up the microinjection apparatus and sharing their invaluable knowledge and experience with insect RNAi. This work was supported by the National Institutes of Health (R01GM113230 to L.P.).
Materials
| Dermestes maculatus live beetles | Our lab or Carolina Biological Supply | #144168 | Our lab strain was verified by COI barcoding; strain variation from Carolina cannot be ruled out |
| Wet cat food | Fancy Feast | Chunks of meat with gravy. Can buy at most pet food and grocery stores | |
| Dry dog food | Purina Puppy Chow | Can buy at most pet food and grocery stores | |
| Insect cage (size medium, 30.5x19x20.3 cm) | Exo Terra | PT2260 | For colony maintenance. Can use larger cage if needed |
| Insect cage (size mini, 17.8×10.2×12.7 cm) | Exo Terra | PT2250 | For embryo collection |
| Petri dish | VWR | 89038-968 | |
| Cotton ball | Fisher | 22-456-883 | |
| Megascript T7 transcription kit | Fisher | AM1334 | For 40 reactions |
| Pneumatic pump | WPI | PV830 | |
| Capillary holder | WPI | ||
| Micromanipulator | NARISHIGE | MN-151 | |
| Black filter paper (90 mm) | VWR | 28342-010 | |
| Food coloring (green) | McCormick | ||
| Borosilicate glass capillary | Hilgenberg | 1406119 | |
| Needle puller (micropipette puller) | Sutter Instrument Co. | P-97 | |
| Microscope glass slide | WorldWide Life Sciences Division | 41351157 | |
| Sealing film (Parafilm M) | Fisher | 13-374-12 | |
| Model 801 Syringe (10 µl ) | Hamilton | 7642-01 | |
| Needle (32-gauge) | Hamilton | 7762-05 | |
| Fixation Solution (Pampel's) | BioQuip Products, Inc. | 1184C | Toxic, needs to be handled in fume hood |
| Forcep (DUMONT #5) | Fine Science Tools | 11252-30 | |
| Cover slip (24X50 mm, No. 1.5) | Globe Scientific | 1415-15 | |
| Eppendorf Femtotips Microloader pipette tip | Fisher | E5242956003 | |
| Dissecting microscopy for embryo injection | Leica | M420 | |
| Dissecting microscopy for larval phenotypic visualization | Zeiss | SteREO Discover. V12 | |
| DIC microscopy | Zeiss | AXIO Imager. M1 | |
References
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Wianny, F., Zernicka-Goetz, M. Specific interference with gene function by double-stranded RNA in early mouse development. Nat Cell Biol. 2, 70-75 (2000).
- Svoboda, P., Stein, P., Hayashi, H., Schultz, R. M. Selective reduction of dormant maternal mRNAs in mouse oocytes by RNA interference. Development. 127, 4147-4156 (2000).
- Zimmermann, T. S., et al. RNAi-mediated gene silencing in non-human primates. Nature. 441, 111-114 (2006).
- Kennerdell, J. R., Carthew, R. W. Use of dsRNA-mediated genetic interference to demonstrate that frizzled and frizzled 2 act in the wingless pathway. Cell. 95, 1017-1026 (1998).
- Cogoni, C., et al. Transgene silencing of the al-1 gene in vegetative cells of Neurospora is mediated by a cytoplasmic effector and does not depend on DNA-DNA interactions or DNA methylation. EMBO J. 15, 3153-3163 (1996).
- Napoli, C., Lemieux, C., Jorgensen, R. Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans. Plant Cell. 2, 279-289 (1990).
- van Roessel, P., Brand, A. H. Spreading silence with Sid. Genome Biol. 5, 208 (2004).
- Grishok, A. RNAi mechanisms in Caenorhabditis elegans. FEBS Lett. 579, 5932-5939 (2005).
- Jose, A. M., Hunter, C. P. Transport of sequence-specific RNA interference information between cells. Annu Rev Genet. 41, 305-330 (2007).
- Hannon, G. J. RNA interference. Nature. 418, 244-251 (2002).
- Hammond, S. M., Caudy, A. A., Hannon, G. J. Post-transcriptional gene silencing by double-stranded RNA. Nat Rev Genet. 2, 110-119 (2001).
- Dorsett, Y., Tuschl, T. siRNAs: applications in functional genomics and potential as therapeutics. Nat Rev Drug Discov. 3, 318-329 (2004).
- Agrawal, N., et al. RNA interference: biology, mechanism, and applications. Microbiol Mol Biol Rev. 67, 657-685 (2003).
- Grishok, A., Tabara, H., Mello, C. C. Genetic requirements for inheritance of RNAi in C. elegans. Science. 287, 2494-2497 (2000).
- Bucher, G., Scholten, J., Klingler, M. Parental RNAi in Tribolium (Coleoptera). Curr Biol. 12, 85-86 (2002).
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854 (1998).
- Turner, C. T., et al. RNA interference in the light brown apple moth, Epiphyas postvittana (Walker) induced by double-stranded RNA feeding. Insect Mol Biol. 15, 383-391 (2006).
- Tabara, H., Grishok, A., Mello, C. C. RNAi in C. elegans: soaking in the genome sequence. Science. 282, 430-431 (1998).
- Eaton, B. A., Fetter, R. D., Davis, G. W. Dynactin is necessary for synapse stabilization. Neuron. 34, 729-741 (2002).
- Travanty, E. A., et al. Using RNA interference to develop dengue virus resistance in genetically modified Aedes aegypti. Insect Biochem Mol Biol. 34, 607-613 (2004).
- Whitten, M. M., et al. Symbiont-mediated RNA interference in insects. Proc Biol Sci. 283, (2016).
- Schmitt-Engel, C., et al. The iBeetle large-scale RNAi screen reveals gene functions for insect development and physiology. Nat Commun. 6, 7822 (2015).
- Dönitz, J., et al. iBeetle-Base: a database for RNAi phenotypes in the red flour beetle Tribolium castaneum. Nucleic Acids Res. 43, 720-725 (2015).
- Ulrich, J., et al. Large scale RNAi screen in Tribolium reveals novel target genes for pest control and the proteasome as prime target. BMC Genomics. 16, 674 (2015).
- Choe, C. P., Miller, S. C., Brown, S. J. A pair-rule gene circuit defines segments sequentially in the short-germ insect Tribolium castaneum. Proc. Natl. Acad. Sci. U. S. A. 103, 6560-6564 (2006).
- Angelini, D. R., Kaufman, T. C. Functional analyses in the hemipteran Oncopeltus fasciatus reveal conserved and derived aspects of appendage patterning in insects. Dev Biol. 271, 306-321 (2004).
- Lynch, J. A., Peel, A. D., Drechsler, A., Averof, M., Roth, S. EGF signaling and the origin of axial polarity among the insects. Curr Biol. 20, 1042-1047 (2010).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223, 171-181 (2013).
- Quan, G. X., Kanda, T., Tamura, T. Induction of the white egg 3 mutant phenotype by injection of the double-stranded RNA of the silkworm white gene. Insect Mol Biol. 11, 217-222 (2002).
- Brown, S. J., Mahaffey, J. P., Lorenzen, M. D., Denell, R. E., Mahaffey, J. W. Using RNAi to investigate orthologous homeotic gene function during development of distantly related insects. Evol Dev. 1, 11-15 (1999).
- Lynch, J. A., Brent, A. E., Leaf, D. S., Pultz, M. A., Desplan, C. Localized maternal orthodenticle patterns anterior and posterior in the long germ wasp Nasonia. Nature. 439, 728-732 (2006).
- Liu, P. Z., Kaufman, T. C. hunchback is required for suppression of abdominal identity, and for proper germband growth and segmentation in the intermediate germband insect Oncopeltus fasciatus. Development. 131, 1515-1527 (2004).
- Zhou, X., Wheeler, M. M., Oi, F. M., Scharf, M. E. RNA interference in the termite Reticulitermes flavipes through ingestion of double-stranded RNA. Insect Biochem Mol Biol. 38, 805-815 (2008).
- Ciudad, L., Piulachs, M. D., Bellés, X. Systemic RNAi of the cockroach vitellogenin receptor results in a phenotype similar to that of the Drosophila yolkless mutant. FEBS J. 273, 325-335 (2006).
- Mito, T., et al. Non-canonical functions of hunchback in segment patterning of the intermediate germ cricket Gryllus bimaculatus. Development. 132, 2069-2079 (2005).
- Yoon, K. S., et al. Brief exposures of human body lice to sublethal amounts of ivermectin over-transcribes detoxification genes involved in tolerance. Insect Mol Biol. 20, 687-699 (2011).
- Rosenberg, M. I., Brent, A. E., Payre, F., Desplan, C. Dual mode of embryonic development is highlighted by expression and function of Nasonia pair-rule genes. Elife. 3, 01440 (2014).
- Hasselmann, M., et al. Evidence for the evolutionary nascence of a novel sex determination pathway in honeybees. Nature. 454, 519-522 (2008).
- Shukla, J. N., Palli, S. R. Sex determination in beetles: production of all male progeny by parental RNAi knockdown of transformer. Sci Rep. 2, 602 (2012).
- Arakane, Y., et al. The Tribolium chitin synthase genes TcCHS1 and TcCHS2 are specialized for synthesis of epidermal cuticle and midgut peritrophic matrix. Insect Mol Biol. 14, 453-463 (2005).
- Cruz, J., Mané-Padròs, D., Bellés, X., Martìn, D. Functions of the ecdysone receptor isoform-A in the hemimetabolous insect Blattella germanica revealed by systemic RNAi in vivo. Dev Biol. 297, 158-171 (2006).
- Guidugli, K. R., et al. Vitellogenin regulates hormonal dynamics in the worker caste of a eusocial insect. FEBS Lett. 579, 4961-4965 (2005).
- Zhang, H., Li, H. C., Miao, X. X. Feasibility, limitation and possible solutions of RNAi-based technology for insect pest control. Insect Sci. 20, 15-30 (2013).
- Huvenne, H., Smagghe, G. Mechanisms of dsRNA uptake in insects and potential of RNAi for pest control: a review. J Insect Physiol. 56, 227-235 (2010).
- Price, D. R., Gatehouse, J. A. RNAi-mediated crop protection against insects. Trends Biotechnol. 26, 393-400 (2008).
- Paldi, N., et al. Effective gene silencing in a microsporidian parasite associated with honeybee (Apis mellifera) colony declines. Appl Environ Microbiol. 76, 5960-5964 (2010).
- Kanginakudru, S., et al. Targeting ie-1 gene by RNAi induces baculoviral resistance in lepidopteran cell lines and in transgenic silkworms. Insect Mol Biol. 16, 635-644 (2007).
- Magni, P. A., Voss, S. C., Testi, R., Borrini, M., Dadour, I. R. A Biological and Procedural Review of Forensically Significant Dermestes Species (Coleoptera: Dermestidae). J Med Entomol. 52, 755-769 (2015).
- Zanetti, N. I., Visciarelli, E. C., Centeno, N. D. The Effect of Temperature and Laboratory Rearing Conditions on the Development of Dermestes maculatus (Coleoptera: Dermestidae). J Forensic Sci. , (2015).
- Veer, V., Negi, B. K., Rao, K. M. Dermestid beetles and some other insect pests associated with stored silkworm cocoons in India, including a world list of dermestid species found attacking this commodity. Journal of Stored Products Research. 32, 69-89 (1996).
- Xiang, J., Forrest, I. S., Pick, L. Dermestes maculatus: an intermediate-germ beetle model system for evo-devo. Evodevo. 6, 32 (2015).
- Fontenot, E. A., Arthur, F. H., Hartzer, K. L. Oviposition of Dermestes maculatus DeGeer, the hide beetle, as affected by biological and environmental conditions. Journal of Stored Products Research. 64, 154-159 (2015).
- Heffer, A., Grubbs, N., Mahaffey, J., Pick, L. The evolving role of the orphan nuclear receptor ftz-f1, a pair-rule segmentation gene. Evol Dev. 15, 406-417 (2013).
- Heffer, A., Shultz, J. W., Pick, L. Surprising flexibility in a conserved Hox transcription factor over 550 million years of evolution. Proc. Natl. Acad. Sci. U. S. A. 107, 18040-18045 (2010).
- Wilson, M. J., Dearden, P. K. Pair-rule gene orthologues have unexpected maternal roles in the honeybee (Apis mellifera). PLoS One. 7, 46490 (2012).
- Dawes, R., Dawson, I., Falciani, F., Tear, G., Akam, M. Dax, a locust Hox gene related to fushi-tarazu but showing no pair-rule expression. Development. 120, 1561-1572 (1994).
- Erezyilmaz, D. F., Kelstrup, H. C., Riddiford, L. M. The nuclear receptor E75A has a novel pair-rule-like function in patterning the milkweed bug, Oncopeltus fasciatus. Dev Biol. 334, 300-310 (2009).
- Stuart, J. J., Brown, S. J., Beeman, R. W., Denell, R. E. A deficiency of the homeotic complex of the beetle Tribolium. Nature. 350, 72-74 (1991).
- Aranda, M., Marques-Souza, H., Bayer, T., Tautz, D. The role of the segmentation gene hairy in Tribolium. Dev Genes Evol. 218, 465-477 (2008).
- Mito, T., et al. even-skipped has gap-like, pair-rule-like, and segmental functions in the cricket Gryllus bimaculatus, a basal, intermediate germ insect (Orthoptera). Dev Biol. 303, 202-213 (2007).
- Patel, N. H., Ball, E. E., Goodman, C. S. Changing role of even-skipped during the evolution of insect pattern formation. Nature. 357, 339-342 (1992).
- Nüsslein-Volhard, C., Wieschaus, E. Mutations affecting segment number and polarity in Drosophila. Nature. 287, 795-801 (1980).
- Jürgens, G., Wieschaus, E., Nüsslein-Volhard, C., Kluding, H. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. II. Zygotic loci on the third chromosome. Wilhelm Roux’s archives of developmental biology. 193, 283-295 (1984).
- Wakimoto, B. T., Kaufman, T. C. Analysis of larval segmentation in lethal genotypes associated with the Aantennapedia gene complex in Drosophila melanogaster. Dev. Biol. 81, 51-64 (1981).
- Yu, Y., et al. The nuclear hormone receptor Ftz-F1 is a cofactor for the Drosophila homeodomain protein Ftz. Nature. 385, 552-555 (1997).
- Nüsslein-Volhard, C., Wieschaus, E., Kluding, H. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. I.Zygotic loci on the second chromosome. Wilhelm Roux’s archives of developmental biology. 193, 267-282 (1984).
- Wieschaus, E., Nüsslein-Volhard, C., Jürgens, G. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. III.Zygotic loci on the X-chromosome and fourth chromosome. ‘Wilhelm Roux’s archives of developmental biology. 193, 296-307 (1984).
- Guichet, A., et al. The nuclear receptor homologue Ftz-F1 and the homeodomain protein Ftz are mutually dependent cofactors. Nature. 385, 548-552 (1997).
- Fontenot, E. A., Arthur, F. H., Hartzer, K. L. Effect of diet and refugia on development of Dermestes maculatus DeGeer reared in a laboratory. J Pest Sci. 88, 113-119 (2014).
- Yang, Y., et al. Biodegradation and Mineralization of Polystyrene by Plastic-Eating Mealworms: Part 1. Chemical and Physical Characterization and Isotopic Tests. Environ Sci Technol. 49, 12080-12086 (2015).
- Kitzmann, P., Schwirz, J., Schmitt-Engel, C., Bucher, G. RNAi phenotypes are influenced by the genetic background of the injected strain. BMC Genomics. 14, 5 (2013).
- Chandler, C. H., Chari, S., Tack, D., Dworkin, I. Causes and consequences of genetic background effects illuminated by integrative genomic analysis. Génétique. 196, 1321-1336 (2014).
- Montagutelli, X. Effect of the genetic background on the phenotype of mouse mutations. J Am Soc Nephrol. 11, 101-105 (2000).
- Doetschman, T. Influence of genetic background on genetically engineered mouse phenotypes. Methods Mol Biol. 530, 423-433 (2009).

