Allevamento e RNA a doppio filamento mediata silenziamento genico nel Hide Beetle,<em> Dermestes maculatus</em
Summary
Qui, vi presentiamo i protocolli per l'allevamento di un coleottero intermedio di germi, Dermestes maculatus (D. maculatus) in laboratorio. Condividiamo anche protocolli per embrionale e genitori RNAi e metodi per l'analisi dei fenotipi embrionali per studiare la funzione dei geni in questa specie.
Abstract
Advances in genomics have raised the possibility of probing biodiversity at an unprecedented scale. However, sequence alone will not be informative without tools to study gene function. The development and sharing of detailed protocols for the establishment of new model systems in laboratories, and for tools to carry out functional studies, is thus crucial for leveraging the power of genomics. Coleoptera (beetles) are the largest clade of insects and occupy virtually all types of habitats on the planet. In addition to providing ideal models for fundamental research, studies of beetles can have impacts on pest control as they are often pests of households, agriculture, and food industries. Detailed protocols for rearing and maintenance of D. maculatus laboratory colonies and for carrying out dsRNA-mediated interference in D. maculatus are presented. Both embryonic and parental RNAi procedures-including apparatus set up, preparation, injection, and post-injection recovery-are described. Methods are also presented for analyzing embryonic phenotypes, including viability, patterning defects in hatched larvae, and cuticle preparations for unhatched larvae. These assays, together with in situ hybridization and immunostaining for molecular markers, make D. maculatus an accessible model system for basic and applied research. They further provide useful information for establishing procedures in other emerging insect model systems.
Introduction
Nel 1998, Fuoco e Mello hanno riferito che a doppio filamento di RNA (dsRNA) può indurre inibizione della funzione del gene in Caenorhabditis elegans 1. Questa risposta innescato da dsRNA è stato chiamato interferenza dell'RNA (RNAi), e tale silenziamento genico RNAi-mediata è stato segnalato per essere conservato in animali, piante e funghi 2-7. In piante e alcuni animali, funzioni RNAi sistemica, il che significa che l'effetto può diffondersi ad altre cellule / tessuti dove dsRNA non viene introdotto direttamente (rivisto nel 8-10). Gli scienziati hanno fatto uso di questo endogena risposta RNAi cellulare progettando dsRNAs di indirizzare i geni di interesse, battendo così giù la funzione del gene senza manipolare direttamente il genoma (rivisto nel 11-14).
RNAi è un potente strumento per studi funzionali causa dei seguenti vantaggi. In primo luogo, anche con il minimo informazioni sequenza del gene, un gene può essere mirata utilizzando RNAi. Ciò è particolarmente importante per studies di organismi non-modello privi di dati genomici e trascrittomica. In secondo luogo, negli organismi dove la risposta RNAi è robusta sistemica RNAi mediata knockdown gene può essere effettuata in quasi ogni stadio di sviluppo. Questa funzione è molto utile per studiare la funzione dei geni pleiotropici. In terzo luogo, in alcuni casi, effetti RNAi diffuso ai gonadi e discendenti, in modo tale che i fenotipi si osservano nella prole 15,16. Questo fenomeno, noto come genitori RNAi (pRNAi), è particolarmente vantaggioso per i geni che influenzano lo sviluppo embrionale, come numerosa prole prodotta da un solo genitore iniettato può essere esaminato, senza manipolazione diretta di uova. Per queste ragioni, pRNAi è il metodo di scelta. Tuttavia, se pRNAi è inefficace, ad esempio per i geni necessari per la oogenesi, allora embrionale RNAi (eRNAi) deve essere utilizzato. Quarto, RNAi può essere utilizzato per generare l'equivalente di una serie allelica dal fatto che la quantità di dsRNA consegnato può essere variata su un intervallo di produrre debole per difetti forti. Tale gradazione di fenotipi può essere utile per comprendere la funzione del gene quando il gene è coinvolto in un processo complesso e / o completa perdita di funzione è letale. In quinto luogo, la consegna di dsRNA è generalmente facile e fattibile, soprattutto negli animali che mostrano solide risposte RNAi sistemiche. dsRNA può essere introdotto da microiniezione 1,5, alimentazione / ingestione 17,18, ammollo, 19,20 e il virus / batteri-mediata consegna 21,22. Sesto, a differenza di alcuni metodi di targeting / modifica dei geni, non vi è alcuna necessità di screening per gli organismi che trasportano la mutazione o di effettuare incroci genetici per generare omozigoti quando si usa RNAi. Pertanto, rispetto a molte altre tecniche per studiare la funzione del gene, RNAi è veloce, poco costoso, e può essere applicato per schermi di grandi dimensioni 23-25.
L'ampio programma di utilità di RNAi fornisce i mezzi per effettuare studi funzionali in una vasta gamma di organismi, ampliando la gamma di specie disponibili per lo studio beyond i sistemi modello tradizionali per i quali sono stati sviluppati strumenti genetici. Ad esempio, gli studi che utilizzano sistemi non-modello sono tenuti a dare intuizioni l'evoluzione dei geni e reti geniche confrontando le funzioni di ortologhi da specie che rappresentano diverse modalità di sviluppo o esporre distinte caratteristiche morfologiche 26-29. Questi tipi di studi forniranno una migliore comprensione della diversità biologica, con conseguenze per la ricerca sia applicata e di base.
Essendo il più grande gruppo di animali sul pianeta, gli insetti offrono una grande opportunità per esplorare i meccanismi alla base della diversità. Inoltre, gli insetti sono in genere piccole, hanno cicli di vita brevi, ad alta fecondità, e sono facili da dietro in laboratorio. Negli ultimi due decenni, RNAi è stato applicato con successo negli insetti che abbracciano gli ordini, tra cui Ditteri (vere mosche) 5, lepidotteri (farfalle e falene) 30, Coleotteri (coleotteri) 16,31, Imenotteri (sawfbugie, vespe, formiche e api) 32, Hemiptera (veri insetti), Isotteri (termiti) 34, Blattodea (scarafaggi) 35, ortotteri (grilli, cavallette, locuste e grilli) 36 e Phthiraptera (pidocchi) 37. Applicazione di successo di RNAi ha fornito dati funzionali per gli studi di patterning in embriogenesi precoce (asse anteriore-posteriore 32, dorso-ventrale asse 28, la segmentazione 26,38), determinazione del sesso, 39,40 chitina / cuticola biosintesi 41, ecdysone segnalazione 42, comportamento sociale 43, e altro ancora. Metodi RNAi sviluppati per diverse specie di insetti possono essere di ulteriore vantaggio in quanto sono suscettibili di essere utile per il controllo dei parassiti (rivisto nel 44-46). effetti RNAi saranno nonché specie-specifico gene-specifico, purché regioni non conservate sono scelti per il targeting. Per le specie di insetti utili come le api e bachi da seta, di targeting dei geni vitali per la sopravvivenza divirus o parassiti per controllare l'infezione possono fornire una nuova strategia per proteggere queste specie 47,48.
Dermestes maculatus (D. maculatus), comune nome nascondere coleottero, è distribuito in tutto il mondo tranne che per l'Antartide. Come un insetto olometaboli, il ciclo di vita D. maculatus comprende embrionale, larvale, pupa, e stadi adulti (Figura 1). Perché si nutre di carne, D. maculatus è utilizzato nei musei per skeletonize animali morti e entomologi forensi può essere utilizzato per stimare momento della morte 49,50. D. maculatus si nutre di prodotti di origine animale, tra cui carcasse, carne secca, formaggio e le pupe / bozzoli di altri insetti e provoca così danni alle famiglie, alimenti conservati, e la seta, il formaggio, e industrie della carne 51,52. L'applicazione di RNAi in questo coleottero potrebbe fornire un modo efficiente ed ecologico per ridurre al minimo il suo impatto economico. Il nostro laboratorio ha utilizzato D. maculatus come nuovo model insetto per studiare la segmentazione 53. Oltre ad essere suscettibili di laboratorio allevamento, D. maculatus è di interesse per la ricerca di base in quanto è uno sviluppatore intermedio di germi, che lo rende una specie utili per studiare la transizione tra breve e di sviluppo a lungo germe.
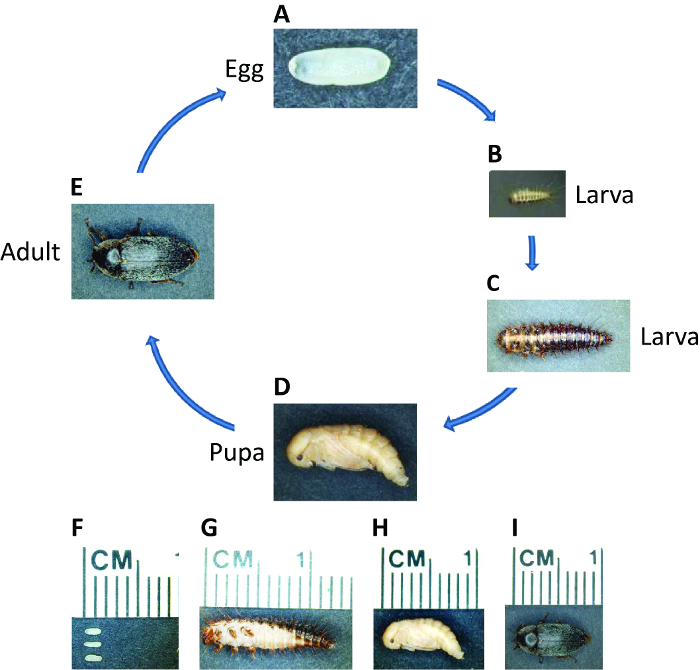
Figura 1: Ciclo di vita di D. maculatus. Fotografie di D. maculatus in diverse fasi della vita, come indicato. Il ciclo di vita da uovo ad adulto dura tre settimane a 30 ° C, ma più a lungo a temperature più basse. (A, F) embrioni appena deposte sono di colore bianco a giallo chiaro e ovali, circa 1,5 mm di lunghezza. Embriogenesi prende ~ 55 ore a 30 ° C. (B, C e G) larve hanno le strisce pigmentate scure e sono ricoperte di setole. Le larve passano attraverso diversi stadi a seconda dell'ambiente e la loro lunghezza può estendersi fino a oltre 1 centimetro. (D, H) </strong> Giovani pupe sono giallo chiaro. Pupation prende ~ 5 – 7 giorni a 30 ° C. (E, I) Poco dopo eclosion, pigmentazione scura appare sopra il corpo adulto coleottero. Gli adulti possono vivere fino a diversi mesi e una femmina può deporre centinaia di embrioni sopra il suo ciclo di vita. Clicca qui per vedere una versione più grande di questa figura.
In precedenza, abbiamo dimostrato che RNAi è efficace nel abbattendo la funzione del gene in D. maculatus 53. Qui la nostra esperienza di allevamento colonie D. maculatus in laboratorio è condiviso con i protocolli passo-passo sia embrionale e genitori RNAi set-up, iniezione, cura post-iniezione, e l'analisi fenotipica. Il silenziamento genico e analisi metodi dsRNA-mediate introdotti qui non solo di fornire informazioni dettagliate per affrontare questioni in D. maculatus, ma hanno anche un significato potenziale foR applicazione di RNAi in altre specie non-modello scarabeo / insetto.
Protocol
Representative Results
Discussion
Mentre un piccolo numero di sistemi modello sofisticati (topi, mosche, vermi) sono stati sviluppati nel corso del 20 ° secolo, il 21 ° secolo ha visto un'ondata di nuovi sistemi di animali in fase di sviluppo nei laboratori di tutto il mondo. Questi nuovi sistemi consentono agli scienziati di affrontare comparativi, domande evolutive che non può essere sondato utilizzando solo gli 'standard' sistemi modello. Questa distribuzione di nuovi modelli richiede il rapido sviluppo di metodi …
Divulgations
The authors have nothing to disclose.
Acknowledgements
We thank Drs. Alison Heffer and Yong Lu for setting up the microinjection apparatus and sharing their invaluable knowledge and experience with insect RNAi. This work was supported by the National Institutes of Health (R01GM113230 to L.P.).
Materials
| Dermestes maculatus live beetles | Our lab or Carolina Biological Supply | #144168 | Our lab strain was verified by COI barcoding; strain variation from Carolina cannot be ruled out |
| Wet cat food | Fancy Feast | Chunks of meat with gravy. Can buy at most pet food and grocery stores | |
| Dry dog food | Purina Puppy Chow | Can buy at most pet food and grocery stores | |
| Insect cage (size medium, 30.5x19x20.3 cm) | Exo Terra | PT2260 | For colony maintenance. Can use larger cage if needed |
| Insect cage (size mini, 17.8×10.2×12.7 cm) | Exo Terra | PT2250 | For embryo collection |
| Petri dish | VWR | 89038-968 | |
| Cotton ball | Fisher | 22-456-883 | |
| Megascript T7 transcription kit | Fisher | AM1334 | For 40 reactions |
| Pneumatic pump | WPI | PV830 | |
| Capillary holder | WPI | ||
| Micromanipulator | NARISHIGE | MN-151 | |
| Black filter paper (90 mm) | VWR | 28342-010 | |
| Food coloring (green) | McCormick | ||
| Borosilicate glass capillary | Hilgenberg | 1406119 | |
| Needle puller (micropipette puller) | Sutter Instrument Co. | P-97 | |
| Microscope glass slide | WorldWide Life Sciences Division | 41351157 | |
| Sealing film (Parafilm M) | Fisher | 13-374-12 | |
| Model 801 Syringe (10 µl ) | Hamilton | 7642-01 | |
| Needle (32-gauge) | Hamilton | 7762-05 | |
| Fixation Solution (Pampel's) | BioQuip Products, Inc. | 1184C | Toxic, needs to be handled in fume hood |
| Forcep (DUMONT #5) | Fine Science Tools | 11252-30 | |
| Cover slip (24X50 mm, No. 1.5) | Globe Scientific | 1415-15 | |
| Eppendorf Femtotips Microloader pipette tip | Fisher | E5242956003 | |
| Dissecting microscopy for embryo injection | Leica | M420 | |
| Dissecting microscopy for larval phenotypic visualization | Zeiss | SteREO Discover. V12 | |
| DIC microscopy | Zeiss | AXIO Imager. M1 | |
References
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Wianny, F., Zernicka-Goetz, M. Specific interference with gene function by double-stranded RNA in early mouse development. Nat Cell Biol. 2, 70-75 (2000).
- Svoboda, P., Stein, P., Hayashi, H., Schultz, R. M. Selective reduction of dormant maternal mRNAs in mouse oocytes by RNA interference. Development. 127, 4147-4156 (2000).
- Zimmermann, T. S., et al. RNAi-mediated gene silencing in non-human primates. Nature. 441, 111-114 (2006).
- Kennerdell, J. R., Carthew, R. W. Use of dsRNA-mediated genetic interference to demonstrate that frizzled and frizzled 2 act in the wingless pathway. Cell. 95, 1017-1026 (1998).
- Cogoni, C., et al. Transgene silencing of the al-1 gene in vegetative cells of Neurospora is mediated by a cytoplasmic effector and does not depend on DNA-DNA interactions or DNA methylation. EMBO J. 15, 3153-3163 (1996).
- Napoli, C., Lemieux, C., Jorgensen, R. Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans. Plant Cell. 2, 279-289 (1990).
- van Roessel, P., Brand, A. H. Spreading silence with Sid. Genome Biol. 5, 208 (2004).
- Grishok, A. RNAi mechanisms in Caenorhabditis elegans. FEBS Lett. 579, 5932-5939 (2005).
- Jose, A. M., Hunter, C. P. Transport of sequence-specific RNA interference information between cells. Annu Rev Genet. 41, 305-330 (2007).
- Hannon, G. J. RNA interference. Nature. 418, 244-251 (2002).
- Hammond, S. M., Caudy, A. A., Hannon, G. J. Post-transcriptional gene silencing by double-stranded RNA. Nat Rev Genet. 2, 110-119 (2001).
- Dorsett, Y., Tuschl, T. siRNAs: applications in functional genomics and potential as therapeutics. Nat Rev Drug Discov. 3, 318-329 (2004).
- Agrawal, N., et al. RNA interference: biology, mechanism, and applications. Microbiol Mol Biol Rev. 67, 657-685 (2003).
- Grishok, A., Tabara, H., Mello, C. C. Genetic requirements for inheritance of RNAi in C. elegans. Science. 287, 2494-2497 (2000).
- Bucher, G., Scholten, J., Klingler, M. Parental RNAi in Tribolium (Coleoptera). Curr Biol. 12, 85-86 (2002).
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854 (1998).
- Turner, C. T., et al. RNA interference in the light brown apple moth, Epiphyas postvittana (Walker) induced by double-stranded RNA feeding. Insect Mol Biol. 15, 383-391 (2006).
- Tabara, H., Grishok, A., Mello, C. C. RNAi in C. elegans: soaking in the genome sequence. Science. 282, 430-431 (1998).
- Eaton, B. A., Fetter, R. D., Davis, G. W. Dynactin is necessary for synapse stabilization. Neuron. 34, 729-741 (2002).
- Travanty, E. A., et al. Using RNA interference to develop dengue virus resistance in genetically modified Aedes aegypti. Insect Biochem Mol Biol. 34, 607-613 (2004).
- Whitten, M. M., et al. Symbiont-mediated RNA interference in insects. Proc Biol Sci. 283, (2016).
- Schmitt-Engel, C., et al. The iBeetle large-scale RNAi screen reveals gene functions for insect development and physiology. Nat Commun. 6, 7822 (2015).
- Dönitz, J., et al. iBeetle-Base: a database for RNAi phenotypes in the red flour beetle Tribolium castaneum. Nucleic Acids Res. 43, 720-725 (2015).
- Ulrich, J., et al. Large scale RNAi screen in Tribolium reveals novel target genes for pest control and the proteasome as prime target. BMC Genomics. 16, 674 (2015).
- Choe, C. P., Miller, S. C., Brown, S. J. A pair-rule gene circuit defines segments sequentially in the short-germ insect Tribolium castaneum. Proc. Natl. Acad. Sci. U. S. A. 103, 6560-6564 (2006).
- Angelini, D. R., Kaufman, T. C. Functional analyses in the hemipteran Oncopeltus fasciatus reveal conserved and derived aspects of appendage patterning in insects. Dev Biol. 271, 306-321 (2004).
- Lynch, J. A., Peel, A. D., Drechsler, A., Averof, M., Roth, S. EGF signaling and the origin of axial polarity among the insects. Curr Biol. 20, 1042-1047 (2010).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223, 171-181 (2013).
- Quan, G. X., Kanda, T., Tamura, T. Induction of the white egg 3 mutant phenotype by injection of the double-stranded RNA of the silkworm white gene. Insect Mol Biol. 11, 217-222 (2002).
- Brown, S. J., Mahaffey, J. P., Lorenzen, M. D., Denell, R. E., Mahaffey, J. W. Using RNAi to investigate orthologous homeotic gene function during development of distantly related insects. Evol Dev. 1, 11-15 (1999).
- Lynch, J. A., Brent, A. E., Leaf, D. S., Pultz, M. A., Desplan, C. Localized maternal orthodenticle patterns anterior and posterior in the long germ wasp Nasonia. Nature. 439, 728-732 (2006).
- Liu, P. Z., Kaufman, T. C. hunchback is required for suppression of abdominal identity, and for proper germband growth and segmentation in the intermediate germband insect Oncopeltus fasciatus. Development. 131, 1515-1527 (2004).
- Zhou, X., Wheeler, M. M., Oi, F. M., Scharf, M. E. RNA interference in the termite Reticulitermes flavipes through ingestion of double-stranded RNA. Insect Biochem Mol Biol. 38, 805-815 (2008).
- Ciudad, L., Piulachs, M. D., Bellés, X. Systemic RNAi of the cockroach vitellogenin receptor results in a phenotype similar to that of the Drosophila yolkless mutant. FEBS J. 273, 325-335 (2006).
- Mito, T., et al. Non-canonical functions of hunchback in segment patterning of the intermediate germ cricket Gryllus bimaculatus. Development. 132, 2069-2079 (2005).
- Yoon, K. S., et al. Brief exposures of human body lice to sublethal amounts of ivermectin over-transcribes detoxification genes involved in tolerance. Insect Mol Biol. 20, 687-699 (2011).
- Rosenberg, M. I., Brent, A. E., Payre, F., Desplan, C. Dual mode of embryonic development is highlighted by expression and function of Nasonia pair-rule genes. Elife. 3, 01440 (2014).
- Hasselmann, M., et al. Evidence for the evolutionary nascence of a novel sex determination pathway in honeybees. Nature. 454, 519-522 (2008).
- Shukla, J. N., Palli, S. R. Sex determination in beetles: production of all male progeny by parental RNAi knockdown of transformer. Sci Rep. 2, 602 (2012).
- Arakane, Y., et al. The Tribolium chitin synthase genes TcCHS1 and TcCHS2 are specialized for synthesis of epidermal cuticle and midgut peritrophic matrix. Insect Mol Biol. 14, 453-463 (2005).
- Cruz, J., Mané-Padròs, D., Bellés, X., Martìn, D. Functions of the ecdysone receptor isoform-A in the hemimetabolous insect Blattella germanica revealed by systemic RNAi in vivo. Dev Biol. 297, 158-171 (2006).
- Guidugli, K. R., et al. Vitellogenin regulates hormonal dynamics in the worker caste of a eusocial insect. FEBS Lett. 579, 4961-4965 (2005).
- Zhang, H., Li, H. C., Miao, X. X. Feasibility, limitation and possible solutions of RNAi-based technology for insect pest control. Insect Sci. 20, 15-30 (2013).
- Huvenne, H., Smagghe, G. Mechanisms of dsRNA uptake in insects and potential of RNAi for pest control: a review. J Insect Physiol. 56, 227-235 (2010).
- Price, D. R., Gatehouse, J. A. RNAi-mediated crop protection against insects. Trends Biotechnol. 26, 393-400 (2008).
- Paldi, N., et al. Effective gene silencing in a microsporidian parasite associated with honeybee (Apis mellifera) colony declines. Appl Environ Microbiol. 76, 5960-5964 (2010).
- Kanginakudru, S., et al. Targeting ie-1 gene by RNAi induces baculoviral resistance in lepidopteran cell lines and in transgenic silkworms. Insect Mol Biol. 16, 635-644 (2007).
- Magni, P. A., Voss, S. C., Testi, R., Borrini, M., Dadour, I. R. A Biological and Procedural Review of Forensically Significant Dermestes Species (Coleoptera: Dermestidae). J Med Entomol. 52, 755-769 (2015).
- Zanetti, N. I., Visciarelli, E. C., Centeno, N. D. The Effect of Temperature and Laboratory Rearing Conditions on the Development of Dermestes maculatus (Coleoptera: Dermestidae). J Forensic Sci. , (2015).
- Veer, V., Negi, B. K., Rao, K. M. Dermestid beetles and some other insect pests associated with stored silkworm cocoons in India, including a world list of dermestid species found attacking this commodity. Journal of Stored Products Research. 32, 69-89 (1996).
- Xiang, J., Forrest, I. S., Pick, L. Dermestes maculatus: an intermediate-germ beetle model system for evo-devo. Evodevo. 6, 32 (2015).
- Fontenot, E. A., Arthur, F. H., Hartzer, K. L. Oviposition of Dermestes maculatus DeGeer, the hide beetle, as affected by biological and environmental conditions. Journal of Stored Products Research. 64, 154-159 (2015).
- Heffer, A., Grubbs, N., Mahaffey, J., Pick, L. The evolving role of the orphan nuclear receptor ftz-f1, a pair-rule segmentation gene. Evol Dev. 15, 406-417 (2013).
- Heffer, A., Shultz, J. W., Pick, L. Surprising flexibility in a conserved Hox transcription factor over 550 million years of evolution. Proc. Natl. Acad. Sci. U. S. A. 107, 18040-18045 (2010).
- Wilson, M. J., Dearden, P. K. Pair-rule gene orthologues have unexpected maternal roles in the honeybee (Apis mellifera). PLoS One. 7, 46490 (2012).
- Dawes, R., Dawson, I., Falciani, F., Tear, G., Akam, M. Dax, a locust Hox gene related to fushi-tarazu but showing no pair-rule expression. Development. 120, 1561-1572 (1994).
- Erezyilmaz, D. F., Kelstrup, H. C., Riddiford, L. M. The nuclear receptor E75A has a novel pair-rule-like function in patterning the milkweed bug, Oncopeltus fasciatus. Dev Biol. 334, 300-310 (2009).
- Stuart, J. J., Brown, S. J., Beeman, R. W., Denell, R. E. A deficiency of the homeotic complex of the beetle Tribolium. Nature. 350, 72-74 (1991).
- Aranda, M., Marques-Souza, H., Bayer, T., Tautz, D. The role of the segmentation gene hairy in Tribolium. Dev Genes Evol. 218, 465-477 (2008).
- Mito, T., et al. even-skipped has gap-like, pair-rule-like, and segmental functions in the cricket Gryllus bimaculatus, a basal, intermediate germ insect (Orthoptera). Dev Biol. 303, 202-213 (2007).
- Patel, N. H., Ball, E. E., Goodman, C. S. Changing role of even-skipped during the evolution of insect pattern formation. Nature. 357, 339-342 (1992).
- Nüsslein-Volhard, C., Wieschaus, E. Mutations affecting segment number and polarity in Drosophila. Nature. 287, 795-801 (1980).
- Jürgens, G., Wieschaus, E., Nüsslein-Volhard, C., Kluding, H. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. II. Zygotic loci on the third chromosome. Wilhelm Roux’s archives of developmental biology. 193, 283-295 (1984).
- Wakimoto, B. T., Kaufman, T. C. Analysis of larval segmentation in lethal genotypes associated with the Aantennapedia gene complex in Drosophila melanogaster. Dev. Biol. 81, 51-64 (1981).
- Yu, Y., et al. The nuclear hormone receptor Ftz-F1 is a cofactor for the Drosophila homeodomain protein Ftz. Nature. 385, 552-555 (1997).
- Nüsslein-Volhard, C., Wieschaus, E., Kluding, H. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. I.Zygotic loci on the second chromosome. Wilhelm Roux’s archives of developmental biology. 193, 267-282 (1984).
- Wieschaus, E., Nüsslein-Volhard, C., Jürgens, G. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. III.Zygotic loci on the X-chromosome and fourth chromosome. ‘Wilhelm Roux’s archives of developmental biology. 193, 296-307 (1984).
- Guichet, A., et al. The nuclear receptor homologue Ftz-F1 and the homeodomain protein Ftz are mutually dependent cofactors. Nature. 385, 548-552 (1997).
- Fontenot, E. A., Arthur, F. H., Hartzer, K. L. Effect of diet and refugia on development of Dermestes maculatus DeGeer reared in a laboratory. J Pest Sci. 88, 113-119 (2014).
- Yang, Y., et al. Biodegradation and Mineralization of Polystyrene by Plastic-Eating Mealworms: Part 1. Chemical and Physical Characterization and Isotopic Tests. Environ Sci Technol. 49, 12080-12086 (2015).
- Kitzmann, P., Schwirz, J., Schmitt-Engel, C., Bucher, G. RNAi phenotypes are influenced by the genetic background of the injected strain. BMC Genomics. 14, 5 (2013).
- Chandler, C. H., Chari, S., Tack, D., Dworkin, I. Causes and consequences of genetic background effects illuminated by integrative genomic analysis. Génétique. 196, 1321-1336 (2014).
- Montagutelli, X. Effect of the genetic background on the phenotype of mouse mutations. J Am Soc Nephrol. 11, 101-105 (2000).
- Doetschman, T. Influence of genetic background on genetically engineered mouse phenotypes. Methods Mol Biol. 530, 423-433 (2009).

