隠すビートルにノックダウン飼育し、二本鎖RNA媒介遺伝子、<em> Dermestes maculatus</em
Summary
ここでは、研究室で(D. maculatus)Dermestes maculatus、中間胚芽カブトムシを飼育するためのプロトコルを提示します。我々はまた、この種における遺伝子機能を研究するために胚の表現型を解析するための胚および親のRNAiのためのプロトコルとメソッドを共有しています。
Abstract
Advances in genomics have raised the possibility of probing biodiversity at an unprecedented scale. However, sequence alone will not be informative without tools to study gene function. The development and sharing of detailed protocols for the establishment of new model systems in laboratories, and for tools to carry out functional studies, is thus crucial for leveraging the power of genomics. Coleoptera (beetles) are the largest clade of insects and occupy virtually all types of habitats on the planet. In addition to providing ideal models for fundamental research, studies of beetles can have impacts on pest control as they are often pests of households, agriculture, and food industries. Detailed protocols for rearing and maintenance of D. maculatus laboratory colonies and for carrying out dsRNA-mediated interference in D. maculatus are presented. Both embryonic and parental RNAi procedures-including apparatus set up, preparation, injection, and post-injection recovery-are described. Methods are also presented for analyzing embryonic phenotypes, including viability, patterning defects in hatched larvae, and cuticle preparations for unhatched larvae. These assays, together with in situ hybridization and immunostaining for molecular markers, make D. maculatus an accessible model system for basic and applied research. They further provide useful information for establishing procedures in other emerging insect model systems.
Introduction
1998年には、火とメロは、二本鎖RNA(dsRNA)が線虫(Caenorhabditis elegans)1における遺伝子機能の抑制を誘導することができることを報告しました。該dsRNAによって誘発この応答は、RNA干渉(RNAi)と命名し、そしてそのようなRNAiを介した遺伝子サイレンシングは、動物、植物、および真菌2-7に保存されることが報告されました。植物や一部の動物において、RNAi機能は、全身、意味の効果が(8月10日に概説)のdsRNAが直接導入されていない他の細胞/組織に広がることができます。科学者たちは、それによって直接(11月14日に見直さ)ゲノムを操作することなく、遺伝子機能をノックダウン、目的の遺伝子を標的とするためのdsRNAを設計することにより、この内因性細胞RNAi応答を利用してきました。
RNAiは、次のような利点のために機能的研究のための強力なツールです。まず、偶数最小限の遺伝子配列情報を用いて、遺伝子は、RNAiを使用して標的化することができます。これは、STのために特に重要ですゲノムやトランスクリプトームデータを欠く非モデル生物のudies。第二に、RNAi応答がロバスト全身である生物で、RNAiを介した遺伝子ノックダウンは、ほぼすべての発達段階で行うことができます。この機能は、多面的な遺伝子の機能を研究するために非常に有用です。第三に、いくつかの場合には、RNAi効果を表現型子孫15,16において観察されるように、生殖腺および子孫に広がります。単一注入親によって生成多数の子孫は、卵を直接操作することなく検査することができるように、親のRNAi(pRNAi)として知られるこの現象は、胚発生に影響を与える遺伝子について特に有利です。これらの理由から、pRNAiを選択する方法です。 pRNAiが無効である場合は、卵形成のために必要な遺伝子のために、たとえば、その後、胚のRNAi(eRNAi)を使用する必要があります。 dsRNAの量は強い欠陥に弱い生成する範囲にわたって変化させることができる送達という点で第四に、RNAiは、対立遺伝子シリーズの等価物を生成するために使用することができます。表現型のようなグラデーションは、遺伝子は致命的である複雑なプロセスおよび/または機能の完全な喪失に関与しているときに遺伝子機能を理解するのに役立つことができます。第五に、dsRNAの配信は、特に堅牢な全身のRNAi応答を示す動物では、一般的に簡単で、実現可能です。 dsRNAは、マイクロインジェクション1,5、摂食/摂取17,18、浸漬、19,20およびウイルス/細菌媒介送達21,22によって導入することができます。第六に、いくつかの遺伝子ターゲティング/編集方法とは異なり、変異を有する生物をスクリーニングするまたはRNAiを用いた場合にホモ接合体を生成するための遺伝的交雑を行う必要はありません。したがって、遺伝子機能を研究するための多くの他の技術と比較して、RNAiは、速く、安価であり、かつ大規模なスクリーニング23-25に適用することができます。
RNAiの広範なユーティリティがbeyon研究のために利用可能な種の範囲を拡大し、生物の広い範囲で機能的研究を遂行するための手段を提供します遺伝的ツールが開発されているために、従来のモデルシステムをdは。例えば、非モデル系を用いた研究は、異なる開発モードを表すか、別個の形態学的特徴26〜29を示す種由来のオルソログの機能を比較することにより、遺伝子および遺伝子ネットワークの進化への洞察を得るために必要とされます。研究のこれらのタイプは、印加されたと基本の両方の研究のための影響で、生物多様性のより良い理解を提供します。
地球上で最大の動物群である、昆虫は多様性のメカニズムを探求する絶好の機会を提供します。また、昆虫は、一般的に小さく、短いライフサイクル、高い繁殖力を持っている、と研究室の後部に簡単です。過去20年間において、RNAiが正常に双翅目(真のハエ)5を含む、注文にまたがる昆虫で適用されてきた、鱗翅目(蝶や蛾)30、鞘翅目(甲虫)16,31、膜翅目(sawf嘘、ハチ、アリやハチ)32、半翅目(真のバグ)、等翅目(シロアリ)34、Blattodea(ゴキブリ)35、直翅目(コオロギ、バッタ、イナゴ、とキリギリス)36と、シラミ目(シラミ)37。 RNAiの成功のアプリケーションは、エクジソンが42シグナル伝達、初期胚発生におけるパターン形成の研究のための機能データ(前後軸32、背腹軸28、セグメンテーション26,38)、性別決意39,40、キチン/キューティクルの生合成41を提供してきました社会的行動43、およびより多くの。彼らは(44-46に概説)害虫防除のために有用である可能性があるという点で異なる昆虫種のために開発されたRNAi法は、付加的な有益であり得ます。 RNAi効果は、非保存領域を標的とするために選択されている限り、遺伝子特異的なだけでなく、種特異的であろう。の生存に不可欠な遺伝子を標的ミツバチやカイコなどの有益な昆虫種については、感染を制御するためのウイルスまたは寄生虫は、これらの種47,48を保護するための新規な戦略を提供することができます。
Dermestes maculatus(D. maculatus)、一般名の非表示カブトムシは、南極大陸を除く世界的に分布しています。完全変態の昆虫としては、D. maculatusのライフサイクルは、胚、幼虫、さなぎ、および成体期( 図1)が含まれています。それは肉を餌にするので、D. maculatusは死んだ動物を骸骨にするために美術館に使用され、法医学昆虫学者は、死亡49,50の時間を推定するためにそれを使用することができます。 D. maculatusは死体、乾燥肉、チーズ、および他の昆虫の蛹/繭などの動物性食品に供給し、このように家計へのダメージ、保存された食品、シルク、チーズ、肉産業51,52の原因となります。この甲虫でRNAiを適用すると、その経済的影響を最小化するための効率的かつ環境に優しい方法を提供することができます。私たちの研究室では、新しいメートルとしてD. maculatusを使用していますセグメンテーション53を研究するodel昆虫。それはそれの有用な種は短期および長期の生殖発達の間の移行を検討すること、中間胚の開発者であるとして、実験室飼育の影響を受けやすいことに加えて、D. maculatusは、基礎研究のために重要です。
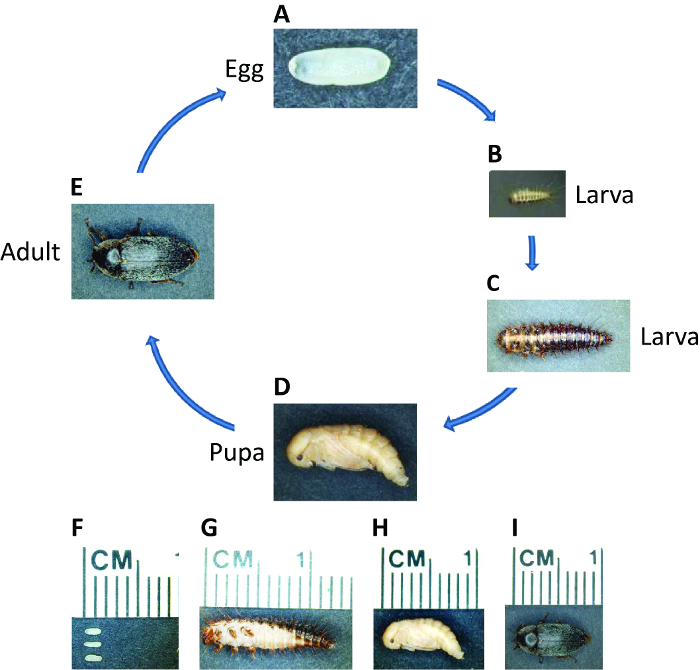
図1:D. maculatusのライフサイクル。示されているように異なるライフステージでのD. maculatusの写真、。卵から大人までのライフサイクルは、より低い温度でより長い30℃で3週間かかりますが。 (A、F)産まれたての胚は、長さ約1.5ミリメートル、淡黄色の楕円形に白です。胚は〜30℃で55時間かかります。 (B、C、およびG)幼虫が暗く着色されたストライプを有しており、毛で覆われています。幼虫は、環境に応じて、いくつかの齢を通過し、その長さは1オーバーセンチにまで拡張することができます。 (D、H) </strオング>ヤング蛹は淡黄色です。 30℃で7日間 – 蛹化は〜5かかります。 (E、I)間もなく羽化後、暗い色素沈着は、大人のカブトムシの体の上に表示されます。大人数ヶ月まで生きることができ、一人の女性が生涯にわたって胚の数百人を置くことができます。 この図の拡大版をご覧になるにはこちらをクリックしてください。
以前、我々は、RNAiは、D. maculatus 53における遺伝子機能のノックダウンに有効であることが示されました。ここでは実験室でD. maculatusコロニーを飼育経験は、胚と親の両方のRNAiセットアップ、注射、注射後のケア、および表現型解析のためのステップバイステップのプロトコルと一緒に共有されています。 D. maculatusで質問に対処するための詳細な情報を提供するだけでなく、foの潜在的な重要性を持っているだけでなく、ここで紹介したdsRNA媒介遺伝子ノックダウンと分析方法rの他の非モデル甲虫/昆虫種でRNAiを適用します。
Protocol
Representative Results
Discussion
洗練されたモデル系(マウス、ハエ、ワーム)の数が少ないが、20 世紀中に開発されたが、21世紀には、世界中の研究室で開発中の新しい動物系の波を見ています。これらの新しいシステムは、科学者が唯一の「標準」モデル系を用いて探索することができません比較、進化の質問に対応することができます。新モデルのこの展開は、迅速な実験室培養方法の開発、遺伝子同?…
Divulgations
The authors have nothing to disclose.
Acknowledgements
We thank Drs. Alison Heffer and Yong Lu for setting up the microinjection apparatus and sharing their invaluable knowledge and experience with insect RNAi. This work was supported by the National Institutes of Health (R01GM113230 to L.P.).
Materials
| Dermestes maculatus live beetles | Our lab or Carolina Biological Supply | #144168 | Our lab strain was verified by COI barcoding; strain variation from Carolina cannot be ruled out |
| Wet cat food | Fancy Feast | Chunks of meat with gravy. Can buy at most pet food and grocery stores | |
| Dry dog food | Purina Puppy Chow | Can buy at most pet food and grocery stores | |
| Insect cage (size medium, 30.5x19x20.3 cm) | Exo Terra | PT2260 | For colony maintenance. Can use larger cage if needed |
| Insect cage (size mini, 17.8×10.2×12.7 cm) | Exo Terra | PT2250 | For embryo collection |
| Petri dish | VWR | 89038-968 | |
| Cotton ball | Fisher | 22-456-883 | |
| Megascript T7 transcription kit | Fisher | AM1334 | For 40 reactions |
| Pneumatic pump | WPI | PV830 | |
| Capillary holder | WPI | ||
| Micromanipulator | NARISHIGE | MN-151 | |
| Black filter paper (90 mm) | VWR | 28342-010 | |
| Food coloring (green) | McCormick | ||
| Borosilicate glass capillary | Hilgenberg | 1406119 | |
| Needle puller (micropipette puller) | Sutter Instrument Co. | P-97 | |
| Microscope glass slide | WorldWide Life Sciences Division | 41351157 | |
| Sealing film (Parafilm M) | Fisher | 13-374-12 | |
| Model 801 Syringe (10 µl ) | Hamilton | 7642-01 | |
| Needle (32-gauge) | Hamilton | 7762-05 | |
| Fixation Solution (Pampel's) | BioQuip Products, Inc. | 1184C | Toxic, needs to be handled in fume hood |
| Forcep (DUMONT #5) | Fine Science Tools | 11252-30 | |
| Cover slip (24X50 mm, No. 1.5) | Globe Scientific | 1415-15 | |
| Eppendorf Femtotips Microloader pipette tip | Fisher | E5242956003 | |
| Dissecting microscopy for embryo injection | Leica | M420 | |
| Dissecting microscopy for larval phenotypic visualization | Zeiss | SteREO Discover. V12 | |
| DIC microscopy | Zeiss | AXIO Imager. M1 | |
References
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Wianny, F., Zernicka-Goetz, M. Specific interference with gene function by double-stranded RNA in early mouse development. Nat Cell Biol. 2, 70-75 (2000).
- Svoboda, P., Stein, P., Hayashi, H., Schultz, R. M. Selective reduction of dormant maternal mRNAs in mouse oocytes by RNA interference. Development. 127, 4147-4156 (2000).
- Zimmermann, T. S., et al. RNAi-mediated gene silencing in non-human primates. Nature. 441, 111-114 (2006).
- Kennerdell, J. R., Carthew, R. W. Use of dsRNA-mediated genetic interference to demonstrate that frizzled and frizzled 2 act in the wingless pathway. Cell. 95, 1017-1026 (1998).
- Cogoni, C., et al. Transgene silencing of the al-1 gene in vegetative cells of Neurospora is mediated by a cytoplasmic effector and does not depend on DNA-DNA interactions or DNA methylation. EMBO J. 15, 3153-3163 (1996).
- Napoli, C., Lemieux, C., Jorgensen, R. Introduction of a Chimeric Chalcone Synthase Gene into Petunia Results in Reversible Co-Suppression of Homologous Genes in trans. Plant Cell. 2, 279-289 (1990).
- van Roessel, P., Brand, A. H. Spreading silence with Sid. Genome Biol. 5, 208 (2004).
- Grishok, A. RNAi mechanisms in Caenorhabditis elegans. FEBS Lett. 579, 5932-5939 (2005).
- Jose, A. M., Hunter, C. P. Transport of sequence-specific RNA interference information between cells. Annu Rev Genet. 41, 305-330 (2007).
- Hannon, G. J. RNA interference. Nature. 418, 244-251 (2002).
- Hammond, S. M., Caudy, A. A., Hannon, G. J. Post-transcriptional gene silencing by double-stranded RNA. Nat Rev Genet. 2, 110-119 (2001).
- Dorsett, Y., Tuschl, T. siRNAs: applications in functional genomics and potential as therapeutics. Nat Rev Drug Discov. 3, 318-329 (2004).
- Agrawal, N., et al. RNA interference: biology, mechanism, and applications. Microbiol Mol Biol Rev. 67, 657-685 (2003).
- Grishok, A., Tabara, H., Mello, C. C. Genetic requirements for inheritance of RNAi in C. elegans. Science. 287, 2494-2497 (2000).
- Bucher, G., Scholten, J., Klingler, M. Parental RNAi in Tribolium (Coleoptera). Curr Biol. 12, 85-86 (2002).
- Timmons, L., Fire, A. Specific interference by ingested dsRNA. Nature. 395, 854 (1998).
- Turner, C. T., et al. RNA interference in the light brown apple moth, Epiphyas postvittana (Walker) induced by double-stranded RNA feeding. Insect Mol Biol. 15, 383-391 (2006).
- Tabara, H., Grishok, A., Mello, C. C. RNAi in C. elegans: soaking in the genome sequence. Science. 282, 430-431 (1998).
- Eaton, B. A., Fetter, R. D., Davis, G. W. Dynactin is necessary for synapse stabilization. Neuron. 34, 729-741 (2002).
- Travanty, E. A., et al. Using RNA interference to develop dengue virus resistance in genetically modified Aedes aegypti. Insect Biochem Mol Biol. 34, 607-613 (2004).
- Whitten, M. M., et al. Symbiont-mediated RNA interference in insects. Proc Biol Sci. 283, (2016).
- Schmitt-Engel, C., et al. The iBeetle large-scale RNAi screen reveals gene functions for insect development and physiology. Nat Commun. 6, 7822 (2015).
- Dönitz, J., et al. iBeetle-Base: a database for RNAi phenotypes in the red flour beetle Tribolium castaneum. Nucleic Acids Res. 43, 720-725 (2015).
- Ulrich, J., et al. Large scale RNAi screen in Tribolium reveals novel target genes for pest control and the proteasome as prime target. BMC Genomics. 16, 674 (2015).
- Choe, C. P., Miller, S. C., Brown, S. J. A pair-rule gene circuit defines segments sequentially in the short-germ insect Tribolium castaneum. Proc. Natl. Acad. Sci. U. S. A. 103, 6560-6564 (2006).
- Angelini, D. R., Kaufman, T. C. Functional analyses in the hemipteran Oncopeltus fasciatus reveal conserved and derived aspects of appendage patterning in insects. Dev Biol. 271, 306-321 (2004).
- Lynch, J. A., Peel, A. D., Drechsler, A., Averof, M., Roth, S. EGF signaling and the origin of axial polarity among the insects. Curr Biol. 20, 1042-1047 (2010).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223, 171-181 (2013).
- Quan, G. X., Kanda, T., Tamura, T. Induction of the white egg 3 mutant phenotype by injection of the double-stranded RNA of the silkworm white gene. Insect Mol Biol. 11, 217-222 (2002).
- Brown, S. J., Mahaffey, J. P., Lorenzen, M. D., Denell, R. E., Mahaffey, J. W. Using RNAi to investigate orthologous homeotic gene function during development of distantly related insects. Evol Dev. 1, 11-15 (1999).
- Lynch, J. A., Brent, A. E., Leaf, D. S., Pultz, M. A., Desplan, C. Localized maternal orthodenticle patterns anterior and posterior in the long germ wasp Nasonia. Nature. 439, 728-732 (2006).
- Liu, P. Z., Kaufman, T. C. hunchback is required for suppression of abdominal identity, and for proper germband growth and segmentation in the intermediate germband insect Oncopeltus fasciatus. Development. 131, 1515-1527 (2004).
- Zhou, X., Wheeler, M. M., Oi, F. M., Scharf, M. E. RNA interference in the termite Reticulitermes flavipes through ingestion of double-stranded RNA. Insect Biochem Mol Biol. 38, 805-815 (2008).
- Ciudad, L., Piulachs, M. D., Bellés, X. Systemic RNAi of the cockroach vitellogenin receptor results in a phenotype similar to that of the Drosophila yolkless mutant. FEBS J. 273, 325-335 (2006).
- Mito, T., et al. Non-canonical functions of hunchback in segment patterning of the intermediate germ cricket Gryllus bimaculatus. Development. 132, 2069-2079 (2005).
- Yoon, K. S., et al. Brief exposures of human body lice to sublethal amounts of ivermectin over-transcribes detoxification genes involved in tolerance. Insect Mol Biol. 20, 687-699 (2011).
- Rosenberg, M. I., Brent, A. E., Payre, F., Desplan, C. Dual mode of embryonic development is highlighted by expression and function of Nasonia pair-rule genes. Elife. 3, 01440 (2014).
- Hasselmann, M., et al. Evidence for the evolutionary nascence of a novel sex determination pathway in honeybees. Nature. 454, 519-522 (2008).
- Shukla, J. N., Palli, S. R. Sex determination in beetles: production of all male progeny by parental RNAi knockdown of transformer. Sci Rep. 2, 602 (2012).
- Arakane, Y., et al. The Tribolium chitin synthase genes TcCHS1 and TcCHS2 are specialized for synthesis of epidermal cuticle and midgut peritrophic matrix. Insect Mol Biol. 14, 453-463 (2005).
- Cruz, J., Mané-Padròs, D., Bellés, X., Martìn, D. Functions of the ecdysone receptor isoform-A in the hemimetabolous insect Blattella germanica revealed by systemic RNAi in vivo. Dev Biol. 297, 158-171 (2006).
- Guidugli, K. R., et al. Vitellogenin regulates hormonal dynamics in the worker caste of a eusocial insect. FEBS Lett. 579, 4961-4965 (2005).
- Zhang, H., Li, H. C., Miao, X. X. Feasibility, limitation and possible solutions of RNAi-based technology for insect pest control. Insect Sci. 20, 15-30 (2013).
- Huvenne, H., Smagghe, G. Mechanisms of dsRNA uptake in insects and potential of RNAi for pest control: a review. J Insect Physiol. 56, 227-235 (2010).
- Price, D. R., Gatehouse, J. A. RNAi-mediated crop protection against insects. Trends Biotechnol. 26, 393-400 (2008).
- Paldi, N., et al. Effective gene silencing in a microsporidian parasite associated with honeybee (Apis mellifera) colony declines. Appl Environ Microbiol. 76, 5960-5964 (2010).
- Kanginakudru, S., et al. Targeting ie-1 gene by RNAi induces baculoviral resistance in lepidopteran cell lines and in transgenic silkworms. Insect Mol Biol. 16, 635-644 (2007).
- Magni, P. A., Voss, S. C., Testi, R., Borrini, M., Dadour, I. R. A Biological and Procedural Review of Forensically Significant Dermestes Species (Coleoptera: Dermestidae). J Med Entomol. 52, 755-769 (2015).
- Zanetti, N. I., Visciarelli, E. C., Centeno, N. D. The Effect of Temperature and Laboratory Rearing Conditions on the Development of Dermestes maculatus (Coleoptera: Dermestidae). J Forensic Sci. , (2015).
- Veer, V., Negi, B. K., Rao, K. M. Dermestid beetles and some other insect pests associated with stored silkworm cocoons in India, including a world list of dermestid species found attacking this commodity. Journal of Stored Products Research. 32, 69-89 (1996).
- Xiang, J., Forrest, I. S., Pick, L. Dermestes maculatus: an intermediate-germ beetle model system for evo-devo. Evodevo. 6, 32 (2015).
- Fontenot, E. A., Arthur, F. H., Hartzer, K. L. Oviposition of Dermestes maculatus DeGeer, the hide beetle, as affected by biological and environmental conditions. Journal of Stored Products Research. 64, 154-159 (2015).
- Heffer, A., Grubbs, N., Mahaffey, J., Pick, L. The evolving role of the orphan nuclear receptor ftz-f1, a pair-rule segmentation gene. Evol Dev. 15, 406-417 (2013).
- Heffer, A., Shultz, J. W., Pick, L. Surprising flexibility in a conserved Hox transcription factor over 550 million years of evolution. Proc. Natl. Acad. Sci. U. S. A. 107, 18040-18045 (2010).
- Wilson, M. J., Dearden, P. K. Pair-rule gene orthologues have unexpected maternal roles in the honeybee (Apis mellifera). PLoS One. 7, 46490 (2012).
- Dawes, R., Dawson, I., Falciani, F., Tear, G., Akam, M. Dax, a locust Hox gene related to fushi-tarazu but showing no pair-rule expression. Development. 120, 1561-1572 (1994).
- Erezyilmaz, D. F., Kelstrup, H. C., Riddiford, L. M. The nuclear receptor E75A has a novel pair-rule-like function in patterning the milkweed bug, Oncopeltus fasciatus. Dev Biol. 334, 300-310 (2009).
- Stuart, J. J., Brown, S. J., Beeman, R. W., Denell, R. E. A deficiency of the homeotic complex of the beetle Tribolium. Nature. 350, 72-74 (1991).
- Aranda, M., Marques-Souza, H., Bayer, T., Tautz, D. The role of the segmentation gene hairy in Tribolium. Dev Genes Evol. 218, 465-477 (2008).
- Mito, T., et al. even-skipped has gap-like, pair-rule-like, and segmental functions in the cricket Gryllus bimaculatus, a basal, intermediate germ insect (Orthoptera). Dev Biol. 303, 202-213 (2007).
- Patel, N. H., Ball, E. E., Goodman, C. S. Changing role of even-skipped during the evolution of insect pattern formation. Nature. 357, 339-342 (1992).
- Nüsslein-Volhard, C., Wieschaus, E. Mutations affecting segment number and polarity in Drosophila. Nature. 287, 795-801 (1980).
- Jürgens, G., Wieschaus, E., Nüsslein-Volhard, C., Kluding, H. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. II. Zygotic loci on the third chromosome. Wilhelm Roux’s archives of developmental biology. 193, 283-295 (1984).
- Wakimoto, B. T., Kaufman, T. C. Analysis of larval segmentation in lethal genotypes associated with the Aantennapedia gene complex in Drosophila melanogaster. Dev. Biol. 81, 51-64 (1981).
- Yu, Y., et al. The nuclear hormone receptor Ftz-F1 is a cofactor for the Drosophila homeodomain protein Ftz. Nature. 385, 552-555 (1997).
- Nüsslein-Volhard, C., Wieschaus, E., Kluding, H. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. I.Zygotic loci on the second chromosome. Wilhelm Roux’s archives of developmental biology. 193, 267-282 (1984).
- Wieschaus, E., Nüsslein-Volhard, C., Jürgens, G. Mutations affecting the pattern of the larval cuticle in Drosophila melanogaster. III.Zygotic loci on the X-chromosome and fourth chromosome. ‘Wilhelm Roux’s archives of developmental biology. 193, 296-307 (1984).
- Guichet, A., et al. The nuclear receptor homologue Ftz-F1 and the homeodomain protein Ftz are mutually dependent cofactors. Nature. 385, 548-552 (1997).
- Fontenot, E. A., Arthur, F. H., Hartzer, K. L. Effect of diet and refugia on development of Dermestes maculatus DeGeer reared in a laboratory. J Pest Sci. 88, 113-119 (2014).
- Yang, Y., et al. Biodegradation and Mineralization of Polystyrene by Plastic-Eating Mealworms: Part 1. Chemical and Physical Characterization and Isotopic Tests. Environ Sci Technol. 49, 12080-12086 (2015).
- Kitzmann, P., Schwirz, J., Schmitt-Engel, C., Bucher, G. RNAi phenotypes are influenced by the genetic background of the injected strain. BMC Genomics. 14, 5 (2013).
- Chandler, C. H., Chari, S., Tack, D., Dworkin, I. Causes and consequences of genetic background effects illuminated by integrative genomic analysis. Génétique. 196, 1321-1336 (2014).
- Montagutelli, X. Effect of the genetic background on the phenotype of mouse mutations. J Am Soc Nephrol. 11, 101-105 (2000).
- Doetschman, T. Influence of genetic background on genetically engineered mouse phenotypes. Methods Mol Biol. 530, 423-433 (2009).

