伪多莫纳斯航空感染模型实时评估工具
Summary
合成囊性纤维化痰介质(SCFM2)可与共聚焦激光扫描显微镜和荧光活性细胞分拣相结合,以高分辨率观察细菌聚集物。本文详细介绍了在抗菌治疗期间评估总人口的方法,作为未来研究的平台。
Abstract
伪多莫纳斯航空(Pa) 是与囊性纤维化(CF)相关的最常见的机会型病原体之一。 一旦Pa 殖民化建立,很大一部分感染细菌在气道痰中形成生物膜。从CF痰中分离出来的 Pa 生物膜已被证明生长在小而密集的聚集体中,这些聚合体为+10-1,000细胞,这些细胞在空间上组织起来,并表现出临床上相关表型,如抗菌耐受性。研究 Pa 聚合物如何应对不断变化的痰环境的最大挑战之一是缺乏营养相关和强大的系统,促进聚合形成。使用合成的 CF 痰介质 (SCFM2),可以使用共聚焦激光扫描显微镜 (CLSM) 和单个细胞分辨率的图像分析来观察 Pa 聚合物的寿命历史。这种 体外 系统允许实时、三维和微米尺度观察数千个大小不一的聚合物。在个体和人口层面,能够按表型和位置对聚合进行分组,有助于观察处于不同发育阶段的聚合物,并有助于精确区分它们对微环境变化的反应,如抗生素治疗。
Introduction
伪多莫纳斯航空(Pa)是一种机会型病原体,可在免疫受损个体中建立慢性感染。对于那些有遗传性疾病囊性纤维化 (CF) 的人,这些感染可以跨越一生的过程。CF导致气道中粘稠、营养丰富的痰的积聚,随着时间的推移,这种痰会被各种微生物病原体殖民。Pa是最常见的CF病原体之一,在幼儿期殖民气道,并建立难以治疗的感染1。Pa仍然是一个严重的临床问题,被认为是CF患者的主要死因,尽管近年来治疗方案有所改善。这种持续表型和增加抗生素耐受性使Pa在疾病控制中心(CDC)和世界卫生组织(WHO)确定为开发新治疗策略的研究重点的病原体中占有一席之地——ESKAPE病原体4。
像其他ESKAPE病原体一样,获得抗生素耐药性在Pa中很常见,但也有许多内在特性有助于Pa抗菌耐药性。其中包括Pa形成+10-1,000细胞的聚集高度密集的集群的能力,这可以在多种感染中观察到,包括CF患者痰5,6。与其他生物膜系统研究的Pa相似,Pa聚合显示临床相关表型,如对抗生素的耐药性增加和细胞细胞通信(量子传感(QS)的激活。例如,Pa的聚合已被证明使用QS调节的行为来对抗其他微生物,以及耐受抗菌治疗,如生产皮奥氰宁7。研究这些行为的能力为人们提供了对细菌生态系统的令人兴奋的洞察,其环境与它们存在于人体中的生态系统相似。
研究Pa聚合物如何应对不断变化的痰环境的最大挑战之一是缺乏营养相关和强大的系统,促进聚合形成。关于Pa的很多已知的东西都是使用体外系统发现的,其中细胞以浮子或在一个典型的表面附着的”蘑菇”结构中生长,而这种结构在体内8中是没有观察到的。虽然经典的生物膜生长模型,如流动细胞或固体阿加,已经产生了关于细菌行为和抗生素耐受机制的广泛和有价值的知识,这些发现并不总是在体内转化。许多体外模型模仿人类感染部位生长环境的能力有限,因此需要昂贵的体内研究。反过来,许多体内模型缺乏体外技术提供的灵活性和分辨率。
合成囊性纤维化痰 (SCFM2) 旨在为Pa生长提供类似于 CF 肺慢性感染期间的环境。SCFM2 包括预期 CF 痰中发现的营养来源,以及粘液、脂质和 DNA。PA生长在SCFM2需要一个几乎相同的基因集,需要在实际痰生长,并支持自然Pa聚合形成9,10。接种后,浮子细胞形成聚合体,通过膨胀增加体积。单个细胞(称为移民)从聚合体中释放出来,迁移到非殖民化地区,并形成新的聚合体10。使用 CLSM 和图像分析在单个细胞的分辨率下观察到此生命史。在SCFM2中形成的Pa的聚合体与CF肺10中观察到的体积大小相似。此模型允许在微米尺度上实时和三维观察不同大小的多个聚合体。延时显微镜可在一个实验中跟踪数千个(+50,000)的聚合物。使用图像分析软件可以从显微图中量化聚合表型,包括聚合体积、表面积和位置三个维度,以最近的 0.1 μm 为数,包括单个聚合度和人口水平。能够按表型和位置对聚合进行分组,可以精确区分不同发育阶段的聚合,以及它们对不断变化的微环境6、11的反应。
SCFM2 应用于低体积和高吞吐量检测中的Pa聚合物研究,使其成为一个灵活、经济高效的模型。作为一种定义的介质,SCFM2在多个平台上提供均匀性和可重复性,为研究Pa聚合体外9提供了营养和物理相关的方法。应用包括它与 CLSM 结合使用,以高分辨率观察空间组织和抗生素耐受性(如本方法本文所述)。能够进行实验,提供实时的,微米级的数据,允许研究物种内部和物种间相互作用,因为它们可能发生在体内。例如,SCFM2以前曾被用来研究细胞细胞通信的空间动力学在总人口通过一个系统网络利用Pa用于调节多个基因,导致毒性和发病机理6。
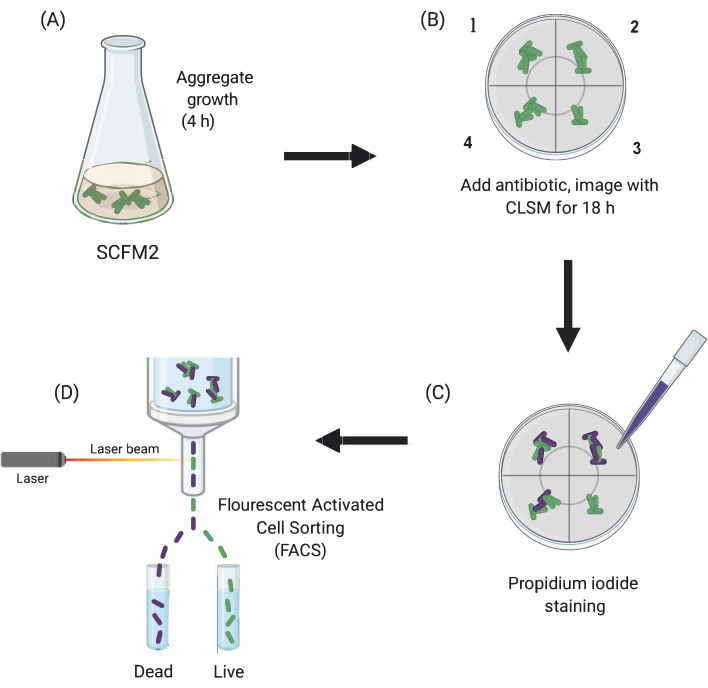
图1:主要实验步骤的图形描述。(A) SCFM2与Pa细胞一起接种,并允许在玻璃底培养皿中形成聚合物。(B) 聚合物被转移到共聚焦显微镜,并添加抗生素。描述是三个技术复制(室1-3)和控制井(4)接种SCFM2没有抗生素治疗。聚合物在初始 18 小时成像后使用 CLSM进行成像,用碘化物处理聚合物,以可视化死细胞,使用 CLSM(D)使用所需表型的聚合物使用 FACS 从 SCFM2 中分离图像。缩写:SCFM2 = 合成囊性纤维化痰介质;帕=伪多莫纳斯航空;CLSM = 聚焦激光扫描显微镜;FACS = 荧光激活细胞排序。请单击此处查看此图的较大版本。
在这里,SCFM2的效用,以研究抗生素治疗对Pa聚合物的实时影响,然后使用细胞分拣方法,以隔离人群与不同的表型进行下游分析(图1)。
Protocol
Representative Results
Discussion
这项工作引入了方法,可以结合研究细菌聚集人群的存在和缺乏抗生素治疗。高分辨率 CLSM 允许在接触抗生素时实时可视化聚合生物量的变化和聚合物的结构方向。此外,可以量化抗生素治疗后剩余生物量的物理和结构特征,目的是利用RNA-seq将这些观测结果与未来的基因表达研究联系起来。虽然使用GFP表达细胞可以可视化抗生素耐药细胞的集体和个人行为,但它只提供了整体情况的一部分。从没…
Divulgations
The authors have nothing to disclose.
Acknowledgements
S.E.D由南佛罗里达大学分子医学系提供的启动资金以及CFF研究赠款(DARCH19G0)N.I.H(5R21AI147654-02(PI,Chen)和美国F微生物群系研究所提供支持。我们感谢怀特利实验室持续合作,涉及与此手稿相关的数据集。我们感谢查尔斯·塞克斯博士为FACS分拣提供便利。数字是由A.D.G和S.E.D使用 Biorender.com 创建的。
Materials
| Amino acids | |||
| Alanine | Acr s Organics s Organics |
56-41-7 | |
| Arginine HCl | MP | 1119-34-2 | |
| Asparagine | Acr s Organics s Organics |
56-84-8 | Prepared in 0.5 M NaOH |
| Cystine HCl | Alfa Aesar | L06328 | |
| Glutamic acid HCl | Acr s Organics s Organics |
138-15-8 | |
| Glycine | Acr s Organics s Organics |
56-40-6 | |
| Histidine HCl H2O | Alfa Aesar | A17627 | |
| Isoleucine | Acr s Organics s Organics |
73-32-5 | |
| Leucine | Alfa Aesar | A12311 | |
| Lysine HCl | Alfa Aesar | J62099 | |
| Methionine | Acr s Organics s Organics |
63-68-3 | |
| Ornithine HCl | Alfa Aesar | A12111 | |
| Phenylalanine | Acr s Organics s Organics |
63-91-2 | |
| Proline | Alfa Aesar | A10199 | |
| Serine | Alfa Aesar | A11179 | |
| Threonine | Acr s Organics s Organics |
72-19-5 | |
| Tryptophan | Acr s Organics s Organics |
73-22-3 | Prepared in 0.2 M NaOH |
| Tyrosine | Alfa Aesar | A11141 | Prepared in 1.0 M NaOH |
| Valine | Acr s Organics s Organics |
72-18-4 | |
| Antibiotic | |||
| Carbenicillin | Alfa Aesar | J6194903 | |
| Day-of Stocks | |||
| CaCl2 * 2H2O | Fisher Chemical | C79-500 | |
| Dextrose (D-glucose) | Fisher Chemical | 50-99-7 | |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Fisher (Avanti Polar Lipids) | 4235-95-4 | shake 15-20 min at 37 °C to evaporate chloroform |
| FeSO4 * 7H2O | Acr s Organics s Organics |
7782-63-0 | this stock equals 1 mg/mL, MUST make fresh |
| L-lactic acid | Alfa Aesar | L13242 | pH stock to 7 with NaOH |
| MgCl2 * 6H2O | Acr s Organics s Organics |
7791-18-6 | |
| N-acetylglucosamine | TCI | A0092 | |
| Prepared solids | |||
| Porcine mucin | Sigma | M1778-100G | UV-sterilize |
| Salmon sperm DNA | Invitrogen | 15632-011 | |
| Stain | |||
| Propidium iodide | Alfa Aesar | J66764MC | |
| Salts | |||
| K2SO4 | Alfa Aesar | A13975 | |
| KCl | Alfa Aesar | J64189 | add solid directly to buffered base |
| KNO3 | Acr s Organics s Organics |
7757-79-1 | |
| MOPS | Alfa Aesar | A12914 | add solid directly to buffered base |
| NaCl | Fisher Chemical | S271-500 | add solid directly to buffered base |
| Na2HPO4 | RPI | S23100-500.0 | |
| NaH2PO4 | RPI | S23120-500.0 | |
| NH4Cl | Acr s Organics s Organics |
12125-02-9 | add solid directly to buffered base |
| Consumables | |||
| Conical tubes (15 mL) | Olympus plastics | 28-101 | |
| Conical tubes (50 mL) | Olympus plastics | 28-106 | |
| Culture tubes w/air flow cap | Olympus plastics | 21-129 | |
| 35 mm four chamber glass-bottom dish | CellVis | NC0600518 | |
| Luria Bertani (LB) broth | Genessee Scientific | 11-118 | |
| Phosphate-buffered saline (PBS) | Fisher Bioreagents | BP2944100 | |
| Pipet tips (p200) | Olympus plastics | 23-150RL | |
| Pipet tips (p1000) | Olympus plastics | 23-165RL | |
| Serological pipets (5 mL) | Olympus plastics | 12-102 | |
| Serological pipets (25 mL) | Olympus plastics | 12-106 | |
| Serological pipets (50 mL) | Olympus plastics | 12-107 | |
| Ultrapure water (RNAse/DNAse free); nanopure water | Genessee Scientific | 18-194 | Nanopure water used for preparation of solutions in Table 1 |
| Syringes (10 mL) | BD | 794412 | |
| Syringes (50 mL) | BD | 309653 | |
| 0.22 mm PES syringe filter | Olympus plastics | 25-244 | |
| PS cuvette semi-mico | Olympus plastics | 91-408 | |
| Software | |||
| Biorender | To prepare the figures | ||
| FacsDiva6.1.3 | Becton Dickinson, San Jose, CA | ||
| Imaris | Bitplane | version 9.6 | |
| Zen Black | |||
| Equipment | |||
| FacsAriallu | Becton Dickinson, San Jose, CA | ||
| LSM 880 confocal laser scanning microscope | Zeiss |
References
- Ramsay, K. A., et al. The changing prevalence of pulmonary infection in with fibrosis: A longitudinal analysis. Journal of Cystic Fibrosis. 16 (1), 70-77 (2017).
- Bessonova, L., et al. Data from the US and UK cystic fibrosis registries support disease modification by CFTR modulation with ivacaftor. Thorax. 73 (8), 731-740 (2018).
- Breuer, O., et al. Changing prevalence of lower airway infections in young children with cystic fibrosis. American Journal of Respiratory and Critical Care Medicine. 200 (5), 590-599 (2019).
- O’Donnell, J. N., Bidell, M. R., Lodise, T. P. Approach to the treatment of patients with serious multidrug-resistant Pseudomonas aeruginosa infections. Pharmacotherapy. 40 (9), 952-969 (2020).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Darch, S. E., et al. Spatial determinants of quorum signaling in a Pseudomonas aeruginosa infection model. Proceedings of the National Academy of Sciences of the United States of America. 115 (18), 4779-4784 (2018).
- Zhu, K., Chen, S., Sysoeva, T. A., You, L. Universal antibiotic tolerance arising from antibiotic-triggered accumulation of pyocyanin in Pseudomonas aeruginosa. PLoS Biology. 17 (12), 3000573 (2019).
- Ciofu, O., Tolker-Nielsen, T. Tolerance and resistance of Pseudomonas aeruginosa biofilms to antimicrobial agents-how P. aeruginosa can escape antibiotics. Frontiers in Microbiology. 10, 913 (2019).
- Turner, K. H., Wessel, A. K., Palmer, G. C., Murray, J. L., Whiteley, M. Essential genome of Pseudomonas aeruginosa in cystic fibrosis sputum. Proceedings of the National Academy of Sciences of the United States of America. 112 (13), 4110-4115 (2015).
- Darch, S. E., et al. Phage inhibit pathogen dissemination by targeting bacterial migrants in a chronic infection model. MBio. 8 (2), 00240 (2017).
- Jorth, P., et al. Regional isolation drives bacterial diversification within cystic fibrosis lungs. Cell Host & Microbe. 18 (3), 307-319 (2015).
- Palmer, K. L., Aye, L. M., Whiteley, M. Nutritional cues control Pseudomonas aeruginosa multicellular behavior in cystic fibrosis sputum. Journal of Bacteriology. 189 (22), 8079-8087 (2007).
- Davies, D. G., et al. The involvement of cell-to-cell signals in the development of a bacterial biofilm. Science. 280 (5361), 295-298 (1998).
- Hartmann, R., et al. Quantitative image analysis of microbial communities with BiofilmQ. Nature Microbiology. 6 (2), 151-156 (2021).
- Stacy, A., et al. Bacterial fight-and-flight responses enhance virulence in a polymicrobial infection. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7819-7824 (2014).

