Identifizierung von alternativem Spleißen und Polyadenylierung in RNA-seq-Daten
Summary
Alternative Spleißen (AS) und alternative Polyadenylierung (APA) erweitern die Vielfalt der Transkriptisoformen und ihrer Produkte. Hier beschreiben wir bioinformatische Protokolle zur Analyse von Bulk-RNA-seq und 3′-Endsequenzierungsassays, um AS und APA zu erkennen und zu visualisieren, die unter experimentellen Bedingungen variieren.
Abstract
Neben der typischen Analyse von RNA-Seq zur Messung der differentiellen Genexpression (DGE) unter experimentellen/biologischen Bedingungen können RNA-seq-Daten auch verwendet werden, um andere komplexe regulatorische Mechanismen auf Exon-Ebene zu erforschen. Alternative Spleißung und Polyadenylierung spielen eine entscheidende Rolle für die funktionelle Vielfalt eines Gens, indem sie verschiedene Isoformen erzeugen, um die Genexpression auf posttranskriptioneller Ebene zu regulieren, und die Beschränkung der Analysen auf die gesamte Genebene kann diese wichtige regulatorische Schicht übersehen. Hier demonstrieren wir detaillierte Schritt-für-Schritt-Analysen zur Identifizierung und Visualisierung der differentiellen Exon- und Polyadenylierungsstellennutzung über Bedingungen hinweg, wobei Bioconductor und andere Pakete und Funktionen wie DEXSeq, diffSplice aus dem Limma-Paket und rMATES verwendet werden.
Introduction
RNA-seq wurde im Laufe der Jahre häufig verwendet, typischerweise zur Schätzung der differentiellen Genexpression und Genentdeckung1. Darüber hinaus kann es auch verwendet werden, um die unterschiedliche Nutzung auf Exon-Ebene aufgrund der Genexprimierung verschiedener Isoformen abzuschätzen, was zu einem besseren Verständnis der Genregulation auf posttranskriptioneller Ebene beiträgt. Die Mehrheit der eukaryotischen Gene erzeugt verschiedene Isoformen durch alternatives Spleißen (AS), um die Vielfalt der mRNA-Expression zu erhöhen. AS-Ereignisse können in verschiedene Muster unterteilt werden: Überspringen vollständiger Exons (SE), bei denen ein (“Kassetten”) Exon zusammen mit seinen flankierenden Introns vollständig aus dem Transkript entfernt wird; alternative (Donor) 5′-Spleißstellenauswahl (A5SS) und alternative 3′ (Akzeptor) Spleißstellenauswahl (A3SS), wenn zwei oder mehr Spleißstellen an beiden Enden eines Exons vorhanden sind; Beibehaltung von Introns (RI), wenn ein Intron innerhalb des reifen mRNA-Transkripts beibehalten wird, und gegenseitiger Ausschluss der Exon-Nutzung (MXE), wobei nur eines der beiden verfügbaren Exons gleichzeitig beibehalten werden kann 2,3. Die alternative Polyadenylierung (APA) spielt auch eine wichtige Rolle bei der Regulierung der Genexpression unter Verwendung alternativer Poly(A)-Stellen, um mehrere mRNA-Isoformen aus einem einzigen Transkriptzu erzeugen 4. Die meisten Polyadenylierungsstellen (pAs) befinden sich in der 3′ untranslatierten Region (3′ UTRs) und erzeugen mRNA-Isoformen mit unterschiedlichen 3′ UTR-Längen. Da die 3′ UTR die zentrale Drehscheibe für die Erkennung regulatorischer Elemente ist, können unterschiedliche 3′ UTR-Längen die mRNA-Lokalisierung, Stabilität und Translation beeinflussen5. Es gibt eine Klasse von 3′-Endsequenzierungsassays, die für den Nachweis von APA optimiert sind und sich in den Details des Protokolls6 unterscheiden. Die hier beschriebene Pipeline ist für PolyA-seq ausgelegt, kann aber wie beschrieben für andere Protokolle angepasst werden.
In dieser Studie stellen wir eine Pipeline von differentiellen Exon-Analysemethoden7,8 (Abbildung 1) vor, die in zwei große Kategorien unterteilt werden können: exon-basiert (DEXSeq9, diffSplice 10) und ereignisbasiert (replicate Multivariate Analysis of Transcript Splicing (rMATS)11). Die Exon-basierten Methoden vergleichen die Faltenänderung über die Bedingungen einzelner Exons hinweg mit einem Maß für die gesamte Genfaltenänderung, um differentiell exprimierte Exon-Nutzung zu nennen, und berechnen daraus ein Maß für die AS-Aktivität auf Genebene. Ereignisbasierte Methoden verwenden Exon-Intron-Spanning-Junction-Lesevorgänge, um bestimmte Spleißereignisse wie Exon-Skipping oder Beibehaltung von Introns zu erkennen und zu klassifizieren und diese AS-Typen in Ausgabe3 zu unterscheiden. Somit bieten diese Methoden komplementäre Sichtweisen für eine vollständige Analyse von AS12,13. Wir haben DEXSeq (basierend auf dem DESeq214 DGE-Paket) und diffSplice (basierend auf dem Limma10 DGE-Paket) für die Studie ausgewählt, da sie zu den am häufigsten verwendeten Paketen für die differentielle Spleißanalyse gehören. rMATS wurde als beliebte Methode für die ereignisbasierte Analyse ausgewählt. Eine weitere beliebte ereignisbasierte Methode ist MISO (Mixture of Isoforms)1. Für APA adaptieren wir den Exon-basierten Ansatz.
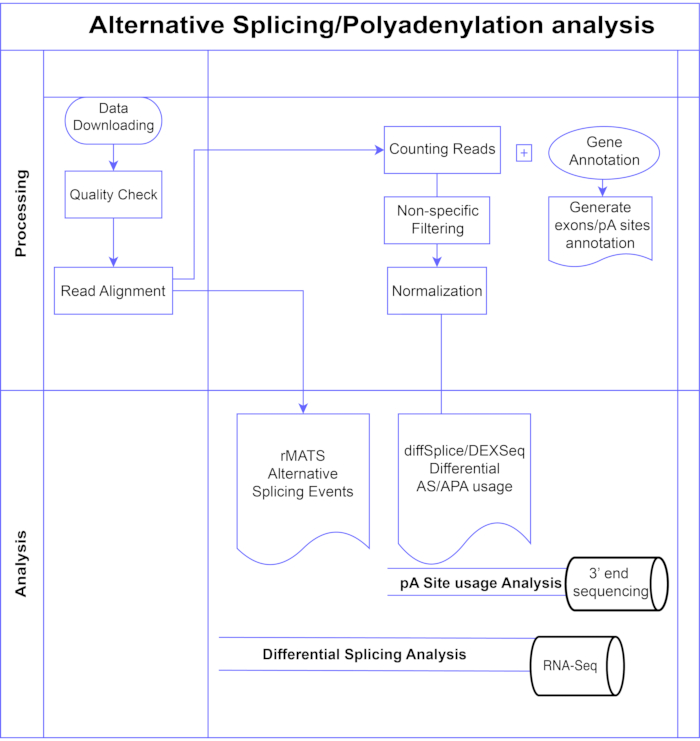
Abbildung 1. Analyse-Pipeline. Flussdiagramm der in der Analyse verwendeten Schritte. Zu den Schritten gehören: Abrufen der Daten, Durchführen von Qualitätsprüfungen und Leseausrichtung, gefolgt von Zählen von Lesevorgängen unter Verwendung von Anmerkungen für bekannte Exons, Introns und pA-Stellen, Filtern zur Entfernung niedriger Zählungen und Normalisierung. PolyA-seq-Daten wurden für alternative pA-Stellen mit diffSplice/DEXSeq-Methoden analysiert, Bulk-RNA-Seq wurde auf alternative Spleißung auf Exon-Ebene mit diffSplice/DEXseq-Methoden analysiert und AS-Ereignisse mit rMATS analysiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Die in dieser Umfrage verwendeten RNA-seq-Daten stammen von Gene Expression Omnibus (GEO) (GSE138691)15. Wir verwendeten Maus-RNA-seq-Daten aus dieser Studie mit zwei Bedingungsgruppen: Wildtyp (WT) und Muskelblind-ähnlicher Typ-1-Knockout (Mbnl1 KO) mit jeweils drei Replikaten. Um die Analyse der differentiellen Polyadenylierungsstandortnutzung zu demonstrieren, erhielten wir PolyA-seq-Daten von Mausembryo-Fibroblasten (MEFs) (GEO Accession GSE60487)16. Die Daten haben vier Bedingungsgruppen: Wild-Typ (WT), Muskelblind-Typ 1/Typ 2 Doppel-Knockout (Mbnl1/2 DKO), Mbnl 1/2 DKO mit Mbnl3-Knockdown (KD) und Mbnl1/2 DKO mit Mbnl3-Kontrolle (Strg). Jede Bedingungsgruppe besteht aus zwei Replikaten.
| GEO-Beitritt | SRA-Ausführungsnummer | Name des Beispiels | Zustand | Replizieren | Gewebe | Sequenzierung | Leselänge | |
| RNA-Seq | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 Knockout | Wiederholung 1 | Thymus | Gepaartes Ende | 100 bp |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 Knockout | Wiederholung 2 | Thymus | Gepaartes Ende | 100 bp | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 Knockout | Wiederholung 3 | Thymus | Gepaartes Ende | 100 bp | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | Wildtyp | Wiederholung 1 | Thymus | Gepaartes Ende | 100 bp | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | Wildtyp | Wiederholung 2 | Thymus | Gepaartes Ende | 100 bp | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | Wildtyp | Wiederholung 3 | Thymus | Gepaartes Ende | 100 bp | |
| 3P-Seq | GSM1480973 | SRR1553129 | WT_1 | Wildtyp (WT) | Wiederholung 1 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp |
| GSM1480974 | SRR1553130 | WT_2 | Wildtyp (WT) | Wiederholung 2 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 Doppel-Knockout (DKO) | Wiederholung 1 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 Doppel-Knockout (DKO) | Wiederholung 2 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 Doppel-Knockout mit Mbnl 3 siRNA (KD) | Wiederholung 1 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 Doppel-Knockout mit Mbnl 3 siRNA (KD) | Wiederholung 2 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 36 bp | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 Doppel-Knockout mit nicht-targetender siRNA (Ctrl) | Wiederholung 1 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 Doppel-Knockout mit nicht-targetender siRNA (Ctrl) | Wiederholung 2 | Embryonale Fibroblasten (MEFs) der Maus | Single-End | 40 bp |
Tabelle 1. Zusammenfassung der RNA-Seq- und PolyA-seq-Datensätze, die für die Analyse verwendet wurden.
Protocol
Representative Results
Discussion
In dieser Studie evaluierten wir Exon-basierte und ereignisbasierte Ansätze zum Nachweis von AS und APA in Bulk-RNA-Seq- und 3′-Endsequenzierungsdaten. Die Exon-basierten AS-Ansätze erzeugen sowohl eine Liste differentiell exprimierter Exons als auch ein Ranking auf Genebene, geordnet nach der statistischen Signifikanz der gesamten differentiellen Spleißaktivität auf Genebene (Tabellen 1-2, 4-5). Für das diffSplice-Paket wird die differentielle Nutzung bestimmt, indem gewichtete lineare Modelle auf …
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Diese Studie wurde von einem Australian Research Council (ARC) Future Fellowship (FT16010043) und ANU Futures Scheme unterstützt.
Riferimenti
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3′ end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).

