זיהוי שחבור חלופי ופוליאדנילציה בנתוני RNA-seq
Summary
שחבור חלופי (AS) ופוליאדנילציה חלופית (APA) מרחיבים את מגוון האיזופורמים של שעתוק ותוצרים. כאן אנו מתארים פרוטוקולים ביואינפורמטיים לניתוח מבחני RNA-seq בתפזורת ו-3′ של ריצוף קצה כדי לזהות ולהמחיש AS ו-APA המשתנים בין תנאי ניסוי.
Abstract
בנוסף לניתוח הטיפוסי של RNA-Seq למדידת ביטוי גנים דיפרנציאלי (DGE) בתנאים ניסיוניים/ביולוגיים, ניתן להשתמש בנתוני RNA-seq גם כדי לחקור מנגנוני ויסות מורכבים אחרים ברמת האקסון. שחבור חלופי ופוליאדנילציה ממלאים תפקיד מכריע במגוון התפקודי של גן על ידי יצירת איזופורמים שונים כדי לווסת את ביטוי הגנים ברמה שלאחר השעתוק, והגבלת הניתוחים לרמת הגן כולה עלולה לפספס את שכבת הוויסות החשובה הזו. כאן, אנו מדגימים ניתוחים מפורטים צעד אחר צעד לזיהוי והדמיה של שימוש באקסון דיפרנציאלי ובאתר פוליאדנילציה בתנאים שונים, תוך שימוש ב- Bioconductor ובחבילות ופונקציות אחרות, כולל DEXSeq, diffSplice מחבילת לימה ו- rMATS.
Introduction
RNA-seq היה בשימוש נרחב לאורך השנים בדרך כלל להערכת ביטוי גנים דיפרנציאליים וגילוי גנים1. בנוסף, ניתן להשתמש בו גם כדי להעריך שימוש משתנה ברמת אקסון עקב גנים המבטאים איזופורמים שונים, ובכך לתרום להבנה טובה יותר של ויסות גנים ברמה שלאחר השעתוק. רוב הגנים האאוקריוטים מייצרים איזופורמים שונים על ידי שחבור חלופי (AS) כדי להגדיל את המגוון של ביטוי mRNA. ניתן לחלק את אירועי AS לדפוסים שונים: דילוג על אקסונים שלמים (SE) שבהם אקסון (“קלטת”) מוסר לחלוטין מהתמליל יחד עם האינטרונים האגפים שלו; בחירת אתר שחבור חלופי (תורם) 5′ (A5SS) וחלופה 3′ (מקבל) בחירת אתר שחבור (A3SS) כאשר שני אתרי שחבור או יותר נמצאים משני קצותיו של אקסון; שמירה של אינטרונים (RI) כאשר אינטרון נשמר בתוך תעתיק mRNA בוגר והדרה הדדית של שימוש באקסון (MXE) כאשר רק אחד משני האקסונים הזמינים יכול להישמר בכל פעם 2,3. פוליאדנילציה חלופית (APA) ממלאת גם תפקיד חשוב בוויסות ביטוי גנים באמצעות אתרי פולי (A) חלופיים ליצירת איזופורמים מרובים של mRNA מתעתיק יחיד4. רוב אתרי הפוליאדנילציה (pAs) ממוקמים באזור 3′ לא מתורגם (3′ UTRs), ומייצרים איזופורמים של mRNA עם אורכי UTR מגוונים של 3′. מכיוון שה-UTR 3′ הוא הרכזת המרכזית לזיהוי אלמנטים רגולטוריים, אורכי UTR שונים של 3′ יכולים להשפיע על לוקליזציה, יציבות ותרגוםmRNA 5. ישנם סוגים של מבחני רצף קצה 3 ‘הממוטבים לזיהוי APA השונים בפרטי הפרוטוקול6. הצינור המתואר כאן מיועד ל- PolyA-seq, אך ניתן להתאים אותו לפרוטוקולים אחרים כמתואר.
במחקר זה אנו מציגים צינור של שיטות ניתוח אקסון דיפרנציאליות7,8 (איור 1), שניתן לחלק לשתי קטגוריות רחבות: מבוססות אקסון (DEXSeq9, diffSplice 10) ומבוססות אירועים (ניתוח רב-משתני משוכפל של שחבור תעתיק (rMATS)11). השיטות המבוססות על אקסון משוות את שינוי הקיפול על פני תנאים של אקסונים בודדים, כנגד מידה של שינוי קיפול גנים כולל כדי לקרוא לשימוש באקסון המתבטא באופן דיפרנציאלי, ומתוך כך מחשבים מדידה ברמת הגן של פעילות AS. שיטות מבוססות אירועים משתמשות בקריאות צומת exon-intron-spanning כדי לזהות ולסווג אירועי שחבור ספציפיים כגון דילוג אקסון או שמירה של אינטרונים, ולהבחין בין סוגי AS אלה בפלט3. לפיכך, שיטות אלה מספקות תצוגות משלימות לניתוח מלא של AS12,13. בחרנו ב- DEXSeq (בהתבסס על חבילת DESeq214 DGE) ו- diffSplice (בהתבסס על חבילת Limma10 DGE) למחקר מכיוון שהם בין החבילות הנפוצות ביותר לניתוח שחבור דיפרנציאלי. rMATS נבחרה כשיטה פופולרית לניתוח מבוסס אירועים. שיטה פופולרית נוספת המבוססת על אירועים היא MISO (תערובת של איזופורמים)1. עבור APA אנו מתאימים את הגישה מבוססת exon.
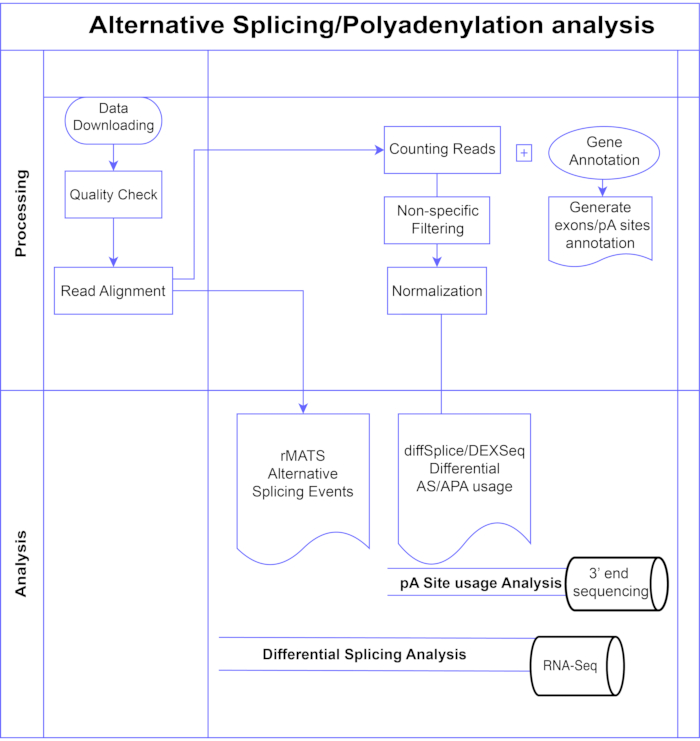
איור 1. צינור ניתוח. תרשים זרימה של השלבים המשמשים בניתוח. השלבים כוללים: קבלת הנתונים, ביצוע בדיקות איכות ויישור קריאה ולאחר מכן ספירת קריאות באמצעות ביאורים עבור exons, introns ואתרי pA ידועים, סינון כדי להסיר ספירות נמוכות ונורמליזציה. נתוני PolyA-seq נותחו עבור אתרי pA חלופיים באמצעות שיטות diffSplice/DEXSeq, RNA-Seq בתפזורת נותח עבור שחבור חלופי ברמת האקסון בשיטות diffSplice/DEXseq, ואירועי AS נותחו עם rMATS. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
נתוני ה-RNA-seq ששימשו בסקר זה נרכשו מביטוי גנים אומניבוס (GEO) (GSE138691)15. השתמשנו בנתוני RNA-seq של עכברים ממחקר זה עם שתי קבוצות מצב: נוקאאוט מסוג פראי (WT) ונוקאאוט דמוי שריר מסוג 1 (Mbnl1 KO) עם שלושה שכפולים כל אחד. כדי להדגים ניתוח שימוש באתר פוליאדנילציה דיפרנציאלית, השגנו נתוני פוליA-seq של עוברי עכברים (MEFs) (GEO Accession GSE60487)16. לנתונים יש ארבע קבוצות מצבים: סוג פראי (WT), דמוי שרירים מסוג 1/סוג 2 נוקאאוט כפול (Mbnl1/2 DKO), Mbnl 1/2 DKO עם נוקאאוט Mbnl3 (KD) ו- Mbnl1/2 DKO עם בקרת Mbnl3 (Ctrl). כל קבוצת תנאים מורכבת משני עותקים משוכפלים.
| הצטרפות גיאוגרפית | מספר הפעלה של SRA | שם לדוגמה | תנאי | לשכפל | טישו | רצף | אורך קריאה | |
| רנ”א-סק | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | נוקאאוט Mbnl1 | נציג 1 | התימוס | סוף מזווג | 100 כ”ס |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | נוקאאוט Mbnl1 | חזרה 2 | התימוס | סוף מזווג | 100 כ”ס | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | נוקאאוט Mbnl1 | חזרה 3 | התימוס | סוף מזווג | 100 כ”ס | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | סוג פראי | נציג 1 | התימוס | סוף מזווג | 100 כ”ס | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | סוג פראי | חזרה 2 | התימוס | סוף מזווג | 100 כ”ס | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | סוג פראי | חזרה 3 | התימוס | סוף מזווג | 100 כ”ס | |
| 3P-Seq | GSM1480973 | SRR1553129 | WT_1 | סוג פראי (WT) | נציג 1 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp |
| GSM1480974 | SRR1553130 | WT_2 | סוג פראי (WT) | חזרה 2 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 נוקאאוט כפול (DKO) | נציג 1 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 נוקאאוט כפול (DKO) | חזרה 2 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 נוקאאוט כפול עם Mbnl 3 siRNA (KD) | נציג 1 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 נוקאאוט כפול עם Mbnl 3 siRNA (KD) | חזרה 2 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 36 bp | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 נוקאאוט כפול עם siRNA ללא מיקוד (Ctrl) | נציג 1 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 נוקאאוט כפול עם siRNA ללא מיקוד (Ctrl) | חזרה 2 | פיברובלסטים עובריים לעכבר (MEFs) | קצה יחיד | 40 bp |
טבלה 1. סיכום של מערכי נתונים של RNA-Seq ו- PolyA-seq המשמשים לניתוח.
Protocol
Representative Results
Discussion
במחקר זה, הערכנו גישות מבוססות אקסון ומבוססות אירועים לזיהוי AS ו-APA בנתוני RNA-Seq בתפזורת ובנתוני ריצוף קצה של 3′. גישות ה-AS המבוססות על אקסון מייצרות הן רשימה של אקסונים המבוטאים באופן דיפרנציאלי והן דירוג ברמת הגן המסודר לפי המובהקות הסטטיסטית של פעילות השחבור הדיפרנציאלית הכוללת ברמת הגן (…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
מחקר זה נתמך על ידי מלגת העתיד של מועצת המחקר האוסטרלית (ARC) (FT16010043) ותוכנית החוזים העתידיים של ANU.
Riferimenti
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3′ end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).

