Et open-source rammeverk for masseberegning av antistoffbaserte terapeutiske molekyler
Summary
Denne artikkelen beskriver bruken av et program, mAbScale, for beregning av masser for monoklonale antistoffbaserte proteinterapier.
Abstract
Bioterapeutiske masser er et middel til å verifisere identitet og strukturell integritet. Massespektrometri (MS) av intakte proteiner eller proteinunderenheter gir et enkelt analyseverktøy for ulike stadier av biofarmasøytisk utvikling. Proteinets identitet bekreftes når den eksperimentelle massen fra MS er innenfor et forhåndsdefinert massefeilområde av den teoretiske massen. Mens det finnes flere beregningsverktøy for beregning av protein- og peptidmolekylvekter, ble de enten ikke designet for direkte anvendelse på bioterapeutiske enheter, har tilgangsbegrensninger på grunn av betalte lisenser, eller krever opplasting av proteinsekvenser til vertsservere.
Vi har utviklet en modulær masseberegningsrutine som gjør det enkelt å bestemme gjennomsnittlig eller monoisotopisk masse og elementære sammensetninger av terapeutiske glykoproteiner, inkludert monoklonale antistoffer (mAb), bispesifikke antistoffer (bsAb) og antistoff-legemiddelkonjugater (ADC). Den modulære naturen til dette Python-baserte beregningsrammeverket vil tillate utvidelse av denne plattformen til andre modaliteter som vaksiner, fusjonsproteiner og oligonukleotider i fremtiden, og dette rammeverket kan også være nyttig for undersøkelse av ovenfra og ned massespektrometridata. Ved å lage et frittstående skrivebordsprogram med åpen kildekode med et grafisk brukergrensesnitt (GUI), håper vi å overvinne begrensningene rundt bruk i miljøer der proprietær informasjon ikke kan lastes opp til nettbaserte verktøy. Denne artikkelen beskriver algoritmene og anvendelsen av dette verktøyet, mAbScale, på forskjellige antistoffbaserte terapeutiske modaliteter.
Introduction
I løpet av de siste to tiårene har bioterapi utviklet seg til å bli en bærebjelke i den moderne farmasøytiske industrien. SARS-CoV2-pandemien og andre livstruende tilstander har ytterligere økt behovet for raskere og bredere utvikling av biofarmasøytiske molekyler 1,2,3.
Den bioterapeutiske molekylvekten er kritisk for identifisering av molekylet, i kombinasjon med andre analytiske analyser. De intakte og reduserte underenhetsmassene brukes gjennom hele oppdagelses- og utviklingslivssyklusen som en del av kontrollstrategier som tar sikte på å opprettholde kvaliteten, som beskrevet i QTPP (Quality Target Product Profile)4.
Analytisk utvikling i biofarmasøytisk industri er avhengig av massemålinger for intakt masseanalyse og dyp karakterisering ved hjelp av peptidkartlegging eller multiattributtmetode (MAM) overvåking. I sentrum av disse teknikkene som benytter moderne massespektrometri (MS) plattformer er evnen til å gi høyoppløselige nøyaktige masse (HR / AM) målinger. De fleste HR / AM-instrumenter gir massenøyaktigheter i området 0,5-5 ppm, som skalerer med masseområdet. Evnen til å måle masser nøyaktig for intakte store molekyler muliggjør rask og trygg identifisering av stormolekylære terapier. Siden isotopoppløsning ikke kan oppnås ved bruk av typiske eksperimentelle betingelser for store molekyler (>10 kDa), må gjennomsnittsmasser beregnes for sammenligning og identifisering 5,6.
Et typisk intakt eller subenhetsproteinmassespektrum representerer den overordnede proteoformprofilen, som inneholder sammensatt informasjon om de forskjellige molekylære formene som følge av posttranslasjonelle modifikasjoner (PTM) og eventuelle primære strukturforskjeller, for eksempel klipp eller sekvensvarianter. Den relativt enkle og høye gjennomstrømningen av disse målingene gjør dem attraktive for karakterisering og som overvåkingskontroller i prosessen 7,8. Dataanalyse for disse eksperimentene krever vanligvis at brukeren definerer søkeområdet for molekylære former (utvalg av PTM eller andre molekylære former). For glykosylerte proteiner er dette søkeområdet i stor grad drevet av glykoformheterogenitet. Kombinasjoner av flere PTM-er, disulfidbindingskonfigurasjoner og andre variasjoner langs primærstrukturen gjør beregning av alle mulige molekylære former til en kjedelig oppgave. Derfor er manuell beregning av mulige molekylære former en tid- og ressurskrevende prosess med høyt potensial for menneskelige feil.
Her presenterer vi et masseberegningsverktøy som ble utviklet med tanke på de viktigste egenskapene til bioterapeutiske molekyler, som mAbs, bsAbs, ADC, etc. Verktøyet gjør det enkelt å innlemme søkeromvariabler for konsistent beregning av masser og elementære komposisjoner. Den modulære naturen til dette verktøyet vil gjøre det mulig å videreutvikle og bruke til masseberegning og massematching for andre modaliteter.
GUI-modulen lar brukeren spesifisere inngangen for masseberegningen, som vist i figur 1; Spesielt går brukeren inn i aminosyresekvenser med én bokstav for lette og tunge antistoffkjeder. Vanlige modifikasjoner for tungkjedet N-terminal syklisering og C-terminal lysinklipping er inkludert som avmerkingsbokser. Videre kan den kjemiske formelen / elementsammensetningen legges til / trekkes fra disse proteinkjedene gjennom den respektive Chem Mod-tekstboksen . Dette gir brukeren fleksibiliteten til å legge til en elementær sammensetning som inkluderer flere posttranslasjonelle modifikasjoner eller en nyttelast med lite molekyl i tilfelle av en ADC. Siden de fleste terapeutiske mAbs er konstruert for å fjerne glykosyleringsstedene i lyskjeden, er glykosylering i lyskjeden valgfri og kan spesifiseres ved hjelp av en avmerkingsboks på GUI.
En typisk variasjon på intakt masseanalyse for antistoffer er en redusert subenhetsmasseanalyse, hvor den lette kjeden løsnes fra den tunge kjeden ved å redusere disulfidbindingene mellom kjedene. Avhengig av styrken av reduksjonsmidlet som brukes, kan de intrakjede disulfidbindingene spaltes eller ikke. Brukerne har fleksibilitet til å angi totalt antall disulfidbindinger avhengig av IgG-subtypen eller i tilfelle en cysteinkonjugert ADC9.
Applikasjonen beregner masser nedenfra og opp, der elementsammensetningene først beregnes for de enkelte tunge kjeder og lette kjeder. Deretter regnskapsføres tungkjede (HC) N-terminal syklisering Lys-klipping ved å justere de beregnede elementære sammensetningene. Eventuelle spesifiserte kjemiske modifikasjoner blir deretter påført de tunge og / eller lette kjedene. Avhengig av typen analyse og disulfidbindingsmønstrene spesifisert av brukeren, justeres antall hydrogener for de to polypeptidkjedene. De glykosylerte HC- og lettkjedemassene (LC) (valgfritt) beregnes basert på brukerens inndata. Til slutt kombineres flere HC- og LC-masser, og disulfidbindingsnumrene oppdateres automatisk for intakt masseberegning.
Med større molekyler som intakte proteiner kan monoisotopiske masser ikke måles på grunn av additivmassedefekten ved bruk av massespektrometre med typisk oppløsningskraft. I stedet måles eller rapporteres nominelle eller gjennomsnittlige masser 5,10,11,12,13. De gjennomsnittlige elementmassene kan variere basert på kilden som brukes til de kuraterte massene14,15. Mens forskjellene i elementmasser kan være små, kan de legge opp til signifikante verdier for molekylvektberegninger med store molekyler. De gjennomsnittlige elementmassene som brukes som standard i programvaren, er vist i tilleggstabell 1. For regulerte miljøer som biofarmasøytisk forskning og utvikling (FoU) -feltet, er det viktig å opprettholde konsistente molekylmasser fordi endringer i massene kan innebære endringer i molekylenheten under regulatoriske innleveringer. For å muliggjøre konsistens i bruken av elementmasser, følger en ordbok med elementmasser med programvareverktøyet som en kommaseparert tekstfil (csv): Element_Mass.csv (Supplementary Coding File 1). På samme måte er en kuratert liste over glykankomposisjoner som vanligvis ses på mAbs inkludert: Glykan.csv (Supplementary Coding File 2). Begge filene lagres på samme mappeplassering som et kjørbart program og kan endres av brukeren for å bruke en bestemt elementær masseliste eller glykanbibliotek.
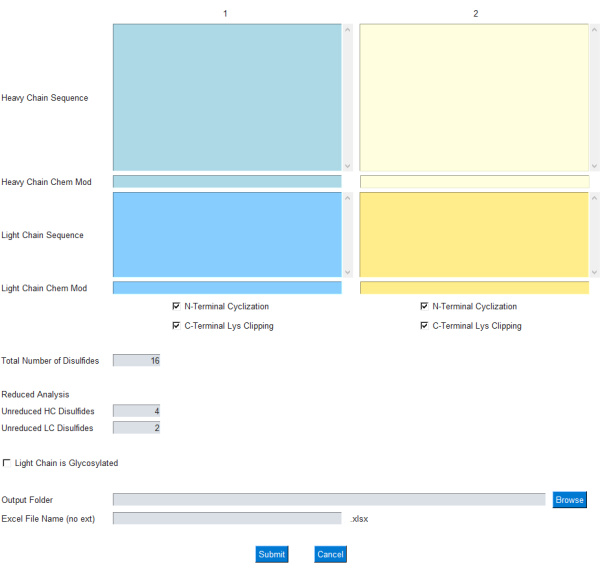
Figur 1: GUI-grensesnitt for mAbScale-programmet. GUI-modulen lar brukeren spesifisere inngangen for masseberegningen. Brukeren legger inn aminosyresekvenser på én bokstav for de lette og tunge antistoffkjedene. Vanlige modifikasjoner for tungkjedet N-terminal syklisering og C-terminal lysinklipping er inkludert som avmerkingsbokser. Kjemiske formler / elementære komposisjoner kan legges til / trekkes fra gjennom den respektive Chem Mod-tekstboksen . Klikk her for å se en større versjon av denne figuren.
Protocol
Representative Results
Discussion
mAbScale gir et intuitivt brukergrensesnitt med fleksibilitet til å endre byggesteinene for masse- og elementberegninger. Brukerne forventes å ha en grunnleggende forståelse av målmolekylet for å bruke applikasjonen, utlede korrekte masser og tolke resultatene. For eksempel kan det intakte eller reduserte masseutgangsarket være overveldende på grunn av de mange radene med intakte eller reduserte masser, siden standard glykandatabase inneholder 88 N-koblede glykaner som ofte finnes i Fc-delen av terapeutiske antist…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Forfatterne takker Robert Schuster for hjelp med dataverifisering.
Materials
| Acquity UPLC system | Waters Corp., Milford, MA | N/A | Modular system |
| Antibody-drug conjugate (ADC) | GlaxoSmithKline | N/A | Proprietory molecule |
| BEH 200 SEC column | Waters Corp., Milford, MA | 176003904 | |
| Bispecific mAb | GlaxoSmithKline | N/A | Proprietory molecule |
| Byos | Protein Metrics, Cupertino, CA | https://proteinmetrics.com/byos/ Version 4.5 |
|
| GPMAW | GPMAW | http://www.gpmaw.com/ | |
| LC-MS grade water | Thermo Fisher Scientific, Waltham, MA | W6-1 | |
| mAb standard | Waters Corp., Milford, MA | 186009125 | Waters Humanized mAb Mass Check Standard |
| mAbScale | GlaxoSmithKline | Apache License, Version 2.0 | |
| Xevo G2 Q-TOF mass spectrometer | Waters Corp., Milford, MA | N/A | Modular system |
Riferimenti
- Reichert, J. M., Valge-Archer, V. E. Development trends for monoclonal antibody cancer therapeutics. Nature Reviews Drug Discovery. 6 (5), 349-356 (2007).
- Kintzing, J. R., Filsinger Interrante, M. V., Cochran, J. R. Emerging strategies for developing next-generation protein therapeutics for cancer treatment. Trends in Pharmacological Sciences. 37 (12), 993-1008 (2016).
- Wang, M. -. Y., et al. SARS-CoV-2: Structure, biology, and structure-based therapeutics development. Frontiers in Cellular and Infection Microbiology. 10, 587269 (2020).
- ICH Q8 (R2) Pharmaceutical Development – Scientific Guideline. European Medicines Agency Available from: https://www.ema.europa.eu/en/ch-q8-r2-pharmaceutical-development-scientific-guideline (2018)
- Donnelly, D. P., et al. Best practices and benchmarks for intact protein analysis for top-down mass spectrometry. Nature Methods. 16 (7), 587-594 (2019).
- Gadgil, H. S., Pipes, G. D., Dillon, T. M., Treuheit, M. J., Bondarenko, P. V. Improving mass accuracy of high performance liquid chromatography/electrospray ionization time-of-flight mass spectrometry of intact antibodies. Journal of the American Society for Mass Spectrometry. 17 (6), 867-872 (2006).
- Beck, A., Sanglier-Cianférani, S., Van Dorsselaer, A. Biosimilar, biobetter, and next generation antibody characterization by mass spectrometry. Analytical Chemistry. 84 (11), 4637-4646 (2012).
- Camperi, J., Goyon, A., Guillarme, D., Zhang, K., Stella, C. Multi-dimensional LC-MS: the next generation characterization of antibody-based therapeutics by unified online bottom-up, middle-up and intact approaches. Analyst. 146 (3), 747-769 (2021).
- Liu, H., May, K. Disulfide bond structures of IgG molecules. mAbs. 4 (1), 17-23 (2012).
- Jakes, C., Füssl, F., Zaborowska, I., Bones, J. Rapid analysis of biotherapeutics using protein a chromatography coupled to orbitrap mass spectrometry. Analytical Chemistry. 93 (40), 13505-13512 (2021).
- Robotham, A. C., Kelly, J. F., Matte, A. Chapter 1 – LC-MS characterization of antibody-based therapeutics: Recent highlights and future prospects. Approaches to the Purification, Analysis and Characterization of Antibody-Based Therapeutics. , 1-33 (2020).
- Valeja, S. G., et al. Unit mass baseline resolution for an intact 148 kDa therapeutic monoclonal antibody by fourier transform ion cyclotron resonance mass spectrometry. Analytical Chemistry. 83 (22), 8391-8395 (2011).
- Fornelli, L., Ayoub, D., Aizikov, K., Beck, A., Tsybin, Y. O. Middle-down analysis of monoclonal antibodies with electron transfer dissociation orbitrap fourier transform mass spectrometry. Analytical Chemistry. 86 (6), 3005-3012 (2014).
- Berglund, M., Wieser, M. E. Isotopic compositions of the elements 2009 (IUPAC Technical Report). Pure and Applied Chemistry. 83 (2), 397-410 (2011).
- Wang, M., et al. The Ame2012 atomic mass evaluation. Chinese Physics C. 36 (12), 1603-2014 (2012).
- Peri, S., Steen, H., Pandey, A. GPMAW–A software tool for analyzing proteins and peptides. Trends in Biochemical Sciences. 26 (11), 687-689 (2001).
- Tipton, J. D., et al. Analysis of intact protein isoforms by mass spectrometry. The Journal of Biological Chemistry. 286 (29), 25451-25458 (2011).
- De Leoz, M. L. A., et al. interlaboratory study on glycosylation analysis of monoclonal antibodies: Comparison of results from diverse analytical methods. Molecular & Cellular Proteomics. 19 (1), 11-30 (2020).
- Cymer, F., Beck, H., Rohde, A., Reusch, D. Therapeutic monoclonal antibody N-glycosylation – Structure, function and therapeutic potential. Biologicals. 52, 1-11 (2018).
- Baker, P. R., Trinidad, J. C., Chalkley, R. J. Modification site localization scoring integrated into a search engine. Molecular & Cellular Proteomics. 10 (7), (2011).
- Chalkley, R. J., Clauser, K. R. Modification site localization scoring: Strategies and performance. Molecular & Cellular Proteomics. 11 (5), 3-14 (2012).

