إطار مفتوح المصدر لحساب الكتلة للجزيئات العلاجية القائمة على الأجسام المضادة
Summary
توضح هذه المقالة استخدام تطبيق برمجي، mAbScale، لحساب الكتل لعلاجات البروتين القائمة على الأجسام المضادة وحيدة النسيلة.
Abstract
كتل العلاج الحيوي هي وسيلة للتحقق من الهوية والسلامة الهيكلية. يوفر قياس الطيف الكتلي (MS) للبروتينات السليمة أو الوحدات الفرعية للبروتين أداة تحليلية سهلة لمراحل مختلفة من تطوير المستحضرات الصيدلانية الحيوية. يتم تأكيد هوية البروتين عندما تكون الكتلة التجريبية من MS ضمن نطاق خطأ الكتلة المحدد مسبقا للكتلة النظرية. في حين توجد العديد من الأدوات الحسابية لحساب الأوزان الجزيئية للبروتين والببتيد ، إلا أنها إما لم تكن مصممة للتطبيق المباشر على كيانات العلاج الحيوي ، أو لديها قيود على الوصول بسبب التراخيص المدفوعة ، أو تتطلب تحميل تسلسلات البروتين إلى خوادم المضيفة.
لقد طورنا روتينا لحساب الكتلة المعيارية يتيح التحديد السهل للكتل المتوسطة أو أحادية النظائر والتركيبات الأولية للبروتينات السكرية العلاجية ، بما في ذلك الأجسام المضادة وحيدة النسيلة (mAb) والأجسام المضادة ثنائية النوعية (bsAb) واتحادات الأدوية والأجسام المضادة (ADC). ستسمح الطبيعة المعيارية لإطار الحساب المستند إلى Python بتوسيع هذه المنصة لتشمل طرائق أخرى مثل اللقاحات وبروتينات الاندماج وقليل النيوكليوتيدات في المستقبل ، ويمكن أن يكون هذا الإطار مفيدا أيضا لاستجواب بيانات قياس الطيف الكتلي من أعلى إلى أسفل. من خلال إنشاء تطبيق سطح مكتب مستقل مفتوح المصدر بواجهة مستخدم رسومية (GUI) ، نأمل في التغلب على القيود المتعلقة بالاستخدام في البيئات التي لا يمكن فيها تحميل معلومات الملكية إلى الأدوات المستندة إلى الويب. توضح هذه المقالة خوارزميات وتطبيق هذه الأداة ، mAbScale ، على طرق علاجية مختلفة قائمة على الأجسام المضادة.
Introduction
على مدى العقدين الماضيين ، تطورت العلاجات الحيوية لتصبح الدعامة الأساسية لصناعة الأدوية الحديثة. أدى جائحة SARS-CoV2 والظروف الأخرى التي تهدد الحياة إلى زيادة الحاجة إلى تطوير أسرع وأوسع لجزيئات المستحضرات الصيدلانية الحيوية1،2،3.
الوزن الجزيئي للعلاج الحيوي أمر بالغ الأهمية لتحديد الجزيء ، بالاقتران مع المقايسات التحليلية الأخرى. يتم استخدام كتل الوحدات الفرعية السليمة والمخفضة طوال دورات حياة الاكتشاف والتطوير كجزء من استراتيجيات التحكم التي تهدف إلى الحفاظ على الجودة ، كما هو موضح في QTPP (ملف تعريف المنتج المستهدف للجودة)4.
يعتمد التطور التحليلي في صناعة المستحضرات الصيدلانية البيولوجية بشكل كبير على قياسات الكتلة لتحليل الكتلة السليمة والتوصيف العميق باستخدام رسم خرائط الببتيد أو مراقبة طريقة متعددة السمات (MAM). في قلب هذه التقنيات التي تستخدم منصات قياس الطيف الكتلي الحديثة (MS) هي القدرة على توفير قياسات كتلة دقيقة عالية الدقة (HR / AM). تنتج معظم أدوات HR / AM دقة جماعية في نطاق 0.5-5 جزء في المليون ، والتي تتناسب مع نطاق الكتلة. تتيح القدرة على قياس الكتل بدقة للجزيئات الكبيرة السليمة التعرف السريع والواثق على علاجات الجزيئات الكبيرة. وبما أنه لا يمكن تحقيق الاستبانة النظيرية باستخدام الظروف التجريبية النموذجية للجزيئات الكبيرة (>10 كيلو دالتون)، يجب حساب متوسط الكتل للمقارنة وتحديد 5,6.
يمثل طيف كتلة البروتين النموذجي السليم أو الفرعي ملف تعريف البروتيوفورم العام ، والذي يحتوي على معلومات مركبة عن الأشكال الجزيئية المختلفة الناتجة عن تعديلات ما بعد الترجمة (PTM) وأي اختلافات في البنية الأولية ، مثل المقاطع أو متغيرات التسلسل. إن الطبيعة السهلة نسبيا وعالية الإنتاجية لهذه القياسات تجعلها جذابة للتوصيف وكضوابط مراقبة أثناء العملية 7,8. عادة ما يتطلب تحليل البيانات لهذه التجارب من المستخدم تحديد مساحة البحث للأشكال الجزيئية (نطاق PTMs أو الأشكال الجزيئية الأخرى). بالنسبة للبروتينات الغليكوزيلاتية ، فإن مساحة البحث هذه مدفوعة إلى حد كبير بعدم تجانس الجليكوفورم. إن مجموعات من PTMs المتعددة ، وتكوينات رابطة ثاني كبريتيد ، والاختلافات الأخرى على طول الهيكل الأساسي تجعل حساب جميع الأشكال الجزيئية الممكنة مهمة شاقة. لذلك ، فإن الحساب اليدوي للأشكال الجزيئية المحتملة هو عملية مستهلكة للوقت والموارد مع احتمال كبير للخطأ البشري.
هنا ، نقدم أداة حساب الكتلة التي تم تطويرها مع الأخذ في الاعتبار أهم ميزات جزيئات العلاج الحيوي ، مثل mAbs و bsAbs و ADCs وما إلى ذلك. تتيح الأداة سهولة دمج متغيرات مساحة البحث من أجل الحساب المتسق للكتل والتراكيب الأولية. وستمكن الطبيعة المعيارية لهذه الأداة من مواصلة تطويرها وتطبيقها على حساب الكتلة ومطابقة الكتلة بالنسبة للطرائق الأخرى.
تسمح وحدة واجهة المستخدم الرسومية للمستخدم بتحديد مدخلات حساب الكتلة ، كما هو موضح في الشكل 1 ؛ على وجه التحديد ، يقوم المستخدم بإدخال تسلسلات الأحماض الأمينية المكونة من حرف واحد لسلاسل الأجسام المضادة الخفيفة والثقيلة. يتم تضمين التعديلات الشائعة لتدوير N-terminal الثقيل وقص اللايسين C-terminal كخانات اختيار. علاوة على ذلك ، يمكن إضافة / طرح الصيغة الكيميائية / التركيب الأولي من سلاسل البروتين هذه من خلال مربع نص Chem Mod المعني. يتيح ذلك للمستخدم المرونة في إضافة تركيبة عنصرية تتضمن تعديلات متعددة بعد الترجمة أو حمولة جزيء صغير في حالة ADC. نظرا لأن معظم mAbs العلاجية مصممة لإزالة مواقع الجليكوزيل في سلسلة الضوء ، يتم ترك الجليكوزيل في سلسلة الضوء اختياريا ويمكن تحديده باستخدام خانة اختيار في واجهة المستخدم الرسومية.
الاختلاف النموذجي في تحليل الكتلة السليمة للأجسام المضادة هو تحليل كتلة الوحدة الفرعية المختزلة ، حيث يتم فصل السلسلة الخفيفة عن السلسلة الثقيلة عن طريق تقليل روابط ثاني كبريتيد السلاسل البينية. اعتمادا على قوة عامل الاختزال المستخدم ، قد تكون أو لا تكون روابط ثاني كبريتيد داخل السلسلة مشقوقة. يتمتع المستخدمون بالمرونة في إدخال العدد الإجمالي لروابط ثاني كبريتيد اعتمادا على النوع الفرعي IgG أو في حالةADC 9 المترافق بالسيستين.
يحسب التطبيق الكتل بطريقة من أسفل إلى أعلى ، حيث يتم حساب التراكيب الأولية أولا للسلاسل الثقيلة الفردية والسلاسل الخفيفة. بعد ذلك ، يتم حساب Lys-Clipping ذات السلسلة الثقيلة (HC) N-terminal cyclization عن طريق ضبط التراكيب الأولية المحسوبة. ثم يتم تطبيق أي تعديلات كيميائية محددة على السلاسل الثقيلة و / أو الخفيفة. اعتمادا على نوع التحليل وأنماط رابطة ثاني كبريتيد المحددة من قبل المستخدم ، يتم ضبط عدد الهيدروجين لسلسلتي عديد الببتيد. يتم حساب كتل HC الغليكوزيلاتي والسلسلة الخفيفة (LC) (اختيارية) بناء على مدخلات المستخدم. أخيرا ، يتم دمج كتل HC و LC متعددة ، ويتم تحديث أرقام روابط ثاني كبريتيد تلقائيا لحساب الكتلة السليمة.
مع الجزيئات الأكبر مثل البروتينات السليمة ، لا يمكن قياس الكتل أحادية النظائر بسبب عيب الكتلة المضافة عند استخدام مطياف الكتلة مع قوة حل نموذجية. بدلا من ذلك ، يتم قياس الكتل الاسمية أو المتوسطة أو الإبلاغ عنها5،10،11،12،13. يمكن أن يختلف متوسط الكتل الأولية بناء على المصدر المستخدم للكتل المنسقة14,15. في حين أن الاختلافات في الكتل الأولية قد تكون صغيرة ، إلا أنها يمكن أن تضيف ما يصل إلى قيم مهمة لحسابات الوزن الجزيئي للجزيء الكبير. يوضح الجدول التكميلي 1 متوسط الكتل الأولية المستخدمة افتراضيا في تطبيق البرنامج. بالنسبة للبيئات الخاضعة للتنظيم مثل مجال البحث والتطوير في مجال المستحضرات الصيدلانية الحيوية (R&D) ، من المهم الحفاظ على كتل جزيئية متسقة لأن التغييرات في الكتل قد تعني تغييرات في الكيان الجزيئي أثناء الإيداعات التنظيمية. لتمكين الاتساق في استخدام الكتل الأولية ، يتم تضمين قاموس للكتل الأولية مع أداة البرنامج كملف نصي لقيمة مفصولة بفواصل (csv): Element_Mass.csv (ملف الترميز التكميلي 1). وبالمثل ، يتم تضمين قائمة منسقة من تركيبات الجليكان التي تظهر عادة على mAbs: Glycan .csv (ملف الترميز التكميلي 2). يتم حفظ كلا الملفين في نفس موقع المجلد كتطبيق قابل للتنفيذ ويمكن للمستخدم تعديلهما لاستخدام قائمة كتلة عنصرية محددة أو مكتبة جليكان.
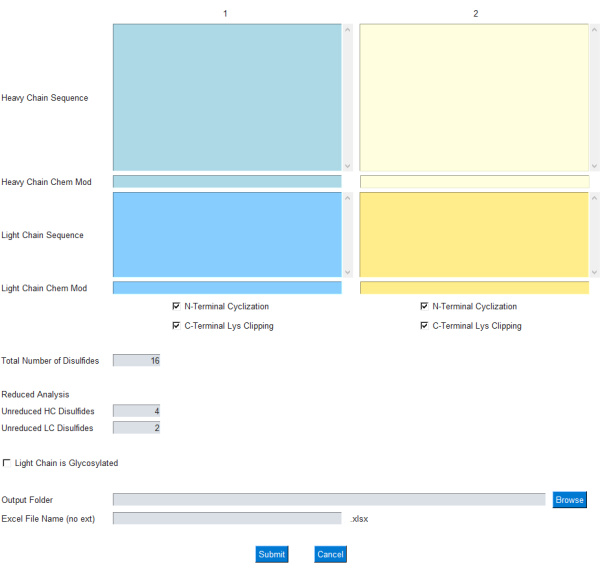
الشكل 1: واجهة المستخدم الرسومية لتطبيق mAbScale. تسمح وحدة واجهة المستخدم الرسومية للمستخدم بتحديد الإدخال لحساب الكتلة. يقوم المستخدم بإدخال تسلسلات الأحماض الأمينية المكونة من حرف واحد لسلاسل الأجسام المضادة الخفيفة والثقيلة. يتم تضمين التعديلات الشائعة لتدوير N-terminal الثقيل وقص ليسين C-terminal كخانات اختيار. يمكن إضافة / طرح الصيغ الكيميائية / التركيبات الأولية من خلال مربع نص Chem Mod المعني. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
Representative Results
Discussion
يوفر mAbScale واجهة مستخدم سهلة الاستخدام مع المرونة لتغيير اللبنات الأساسية لحسابات الكتلة والعناصر. من المتوقع أن يكون لدى المستخدمين فهم أساسي للجزيء المستهدف لاستخدام التطبيق ، واشتقاق الكتل الصحيحة ، وتفسير النتائج. على سبيل المثال ، يمكن أن تكون ورقة إخراج الكتلة السليمة أو المخفضة سا…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
يشكر المؤلفون روبرت شوستر على المساعدة في التحقق من البيانات.
Materials
| Acquity UPLC system | Waters Corp., Milford, MA | N/A | Modular system |
| Antibody-drug conjugate (ADC) | GlaxoSmithKline | N/A | Proprietory molecule |
| BEH 200 SEC column | Waters Corp., Milford, MA | 176003904 | |
| Bispecific mAb | GlaxoSmithKline | N/A | Proprietory molecule |
| Byos | Protein Metrics, Cupertino, CA | https://proteinmetrics.com/byos/ Version 4.5 |
|
| GPMAW | GPMAW | http://www.gpmaw.com/ | |
| LC-MS grade water | Thermo Fisher Scientific, Waltham, MA | W6-1 | |
| mAb standard | Waters Corp., Milford, MA | 186009125 | Waters Humanized mAb Mass Check Standard |
| mAbScale | GlaxoSmithKline | Apache License, Version 2.0 | |
| Xevo G2 Q-TOF mass spectrometer | Waters Corp., Milford, MA | N/A | Modular system |
Riferimenti
- Reichert, J. M., Valge-Archer, V. E. Development trends for monoclonal antibody cancer therapeutics. Nature Reviews Drug Discovery. 6 (5), 349-356 (2007).
- Kintzing, J. R., Filsinger Interrante, M. V., Cochran, J. R. Emerging strategies for developing next-generation protein therapeutics for cancer treatment. Trends in Pharmacological Sciences. 37 (12), 993-1008 (2016).
- Wang, M. -. Y., et al. SARS-CoV-2: Structure, biology, and structure-based therapeutics development. Frontiers in Cellular and Infection Microbiology. 10, 587269 (2020).
- ICH Q8 (R2) Pharmaceutical Development – Scientific Guideline. European Medicines Agency Available from: https://www.ema.europa.eu/en/ch-q8-r2-pharmaceutical-development-scientific-guideline (2018)
- Donnelly, D. P., et al. Best practices and benchmarks for intact protein analysis for top-down mass spectrometry. Nature Methods. 16 (7), 587-594 (2019).
- Gadgil, H. S., Pipes, G. D., Dillon, T. M., Treuheit, M. J., Bondarenko, P. V. Improving mass accuracy of high performance liquid chromatography/electrospray ionization time-of-flight mass spectrometry of intact antibodies. Journal of the American Society for Mass Spectrometry. 17 (6), 867-872 (2006).
- Beck, A., Sanglier-Cianférani, S., Van Dorsselaer, A. Biosimilar, biobetter, and next generation antibody characterization by mass spectrometry. Analytical Chemistry. 84 (11), 4637-4646 (2012).
- Camperi, J., Goyon, A., Guillarme, D., Zhang, K., Stella, C. Multi-dimensional LC-MS: the next generation characterization of antibody-based therapeutics by unified online bottom-up, middle-up and intact approaches. Analyst. 146 (3), 747-769 (2021).
- Liu, H., May, K. Disulfide bond structures of IgG molecules. mAbs. 4 (1), 17-23 (2012).
- Jakes, C., Füssl, F., Zaborowska, I., Bones, J. Rapid analysis of biotherapeutics using protein a chromatography coupled to orbitrap mass spectrometry. Analytical Chemistry. 93 (40), 13505-13512 (2021).
- Robotham, A. C., Kelly, J. F., Matte, A. Chapter 1 – LC-MS characterization of antibody-based therapeutics: Recent highlights and future prospects. Approaches to the Purification, Analysis and Characterization of Antibody-Based Therapeutics. , 1-33 (2020).
- Valeja, S. G., et al. Unit mass baseline resolution for an intact 148 kDa therapeutic monoclonal antibody by fourier transform ion cyclotron resonance mass spectrometry. Analytical Chemistry. 83 (22), 8391-8395 (2011).
- Fornelli, L., Ayoub, D., Aizikov, K., Beck, A., Tsybin, Y. O. Middle-down analysis of monoclonal antibodies with electron transfer dissociation orbitrap fourier transform mass spectrometry. Analytical Chemistry. 86 (6), 3005-3012 (2014).
- Berglund, M., Wieser, M. E. Isotopic compositions of the elements 2009 (IUPAC Technical Report). Pure and Applied Chemistry. 83 (2), 397-410 (2011).
- Wang, M., et al. The Ame2012 atomic mass evaluation. Chinese Physics C. 36 (12), 1603-2014 (2012).
- Peri, S., Steen, H., Pandey, A. GPMAW–A software tool for analyzing proteins and peptides. Trends in Biochemical Sciences. 26 (11), 687-689 (2001).
- Tipton, J. D., et al. Analysis of intact protein isoforms by mass spectrometry. The Journal of Biological Chemistry. 286 (29), 25451-25458 (2011).
- De Leoz, M. L. A., et al. interlaboratory study on glycosylation analysis of monoclonal antibodies: Comparison of results from diverse analytical methods. Molecular & Cellular Proteomics. 19 (1), 11-30 (2020).
- Cymer, F., Beck, H., Rohde, A., Reusch, D. Therapeutic monoclonal antibody N-glycosylation – Structure, function and therapeutic potential. Biologicals. 52, 1-11 (2018).
- Baker, P. R., Trinidad, J. C., Chalkley, R. J. Modification site localization scoring integrated into a search engine. Molecular & Cellular Proteomics. 10 (7), (2011).
- Chalkley, R. J., Clauser, K. R. Modification site localization scoring: Strategies and performance. Molecular & Cellular Proteomics. 11 (5), 3-14 (2012).

