Фреймворк с открытым исходным кодом для расчета массы терапевтических молекул на основе антител
Summary
В этой статье описывается использование программного приложения mAbScale для расчета масс для белковой терапии на основе моноклональных антител.
Abstract
Биотерапевтические массы являются средством проверки идентичности и структурной целостности. Масс-спектрометрия (МС) интактных белков или белковых субъединиц обеспечивает простой аналитический инструмент для различных стадий биофармацевтических разработок. Идентичность белка подтверждается, когда экспериментальная масса из МС находится в пределах заданного диапазона погрешности массы от теоретической массы. Несмотря на то, что существует несколько вычислительных инструментов для вычисления молекулярных масс белков и пептидов, они либо не были разработаны для непосредственного применения в биотерапевтических учреждениях, либо имеют ограничения доступа из-за платных лицензий, либо требуют загрузки белковых последовательностей на хост-серверы.
Мы разработали модульную процедуру расчета массы, которая позволяет легко определять средние или моноизотопные массы и элементный состав терапевтических гликопротеинов, включая моноклональные антитела (mAb), биспецифические антитела (bsAb) и конъюгаты антитело-лекарство (ADC). Модульный характер этой вычислительной платформы на основе Python позволит в будущем распространить эту платформу на другие модальности, такие как вакцины, гибридные белки и олигонуклеотиды, и эта структура также может быть полезна для изучения данных масс-спектрометрии сверху вниз. Создав автономное настольное приложение с открытым исходным кодом и графическим пользовательским интерфейсом (GUI), мы надеемся преодолеть ограничения, связанные с использованием в средах, где конфиденциальная информация не может быть загружена в веб-инструменты. В данной статье описаны алгоритмы и применение этого инструмента, mAbScale, к различным терапевтическим методам на основе антител.
Introduction
За последние два десятилетия биотерапевтические препараты превратились в основу современной фармацевтической промышленности. Пандемия SARS-CoV2 и другие опасные для жизни состояния еще больше увеличили потребность в более быстрой и широкой разработке биофармацевтических молекул 1,2,3.
Биотерапевтическая молекулярная масса имеет решающее значение для идентификации молекулы в сочетании с другими аналитическими анализами. Неповрежденные и уменьшенные массы субъединиц используются на протяжении всего жизненного цикла открытия и разработки в рамках стратегий контроля, направленных на поддержание качества, как описано в QTPP (Quality Target Product Profile)4.
Аналитические разработки в биофармацевтической промышленности в значительной степени опираются на измерения массы для анализа интактной массы и глубокой характеристики с использованием пептидного картирования или мониторинга многоатрибутного метода (MAM). В основе этих методов, использующих современные платформы масс-спектрометрии (МС), лежит возможность обеспечения точных измерений массы (ЧСС/АМ) с высоким разрешением. Большинство приборов ЧСС/АМ дают точность измерения массы в диапазоне 0,5-5 ppm, которая зависит от диапазона масс. Возможность точного измерения масс неповрежденных больших молекул позволяет быстро и уверенно идентифицировать крупномолекулярные терапевтические средства. Поскольку изотопное разрешение не может быть достигнуто в типичных экспериментальных условиях для больших молекул (>10 кДа), для сравнения и идентификации необходимо рассчитать средние массы 5,6.
Типичный масс-спектр интактного или субъединичного белка представляет собой общий профиль протеоформы, который содержит сводную информацию о различных молекулярных формах, возникающих в результате посттрансляционных модификаций (ПТМ), и любых первичных структурных различиях, таких как клипсы или варианты последовательностей. Относительная простота и высокая производительность этих измерений делают их привлекательными для определения характеристик и в качестве средств контроля в процессе производства 7,8. Анализ данных для этих экспериментов обычно требует от пользователя определения пространства поиска молекулярных форм (диапазона ПТМ или других молекулярных форм). Для гликозилированных белков это пространство поиска в значительной степени обусловлено гетерогенностью гликоформа. Комбинации нескольких ПТМ, конфигураций дисульфидных связей и других вариаций вдоль первичной структуры делают расчет всех возможных молекулярных форм утомительной задачей. Поэтому ручной расчет возможных молекулярных форм является трудоемким и ресурсоемким процессом с высокой вероятностью человеческой ошибки.
Здесь мы представляем инструмент расчета массы, который был разработан с учетом наиболее важных характеристик биотерапевтических молекул, таких как мАТ, бсАБ, АЦП и т.д. Инструмент позволяет легко вводить переменные пространства поиска для согласованного вычисления масс и элементного состава. Модульный характер этого инструмента позволит в дальнейшем развивать его и применять для вычисления массы и сопоставления масс для других модальностей.
Модуль GUI позволяет пользователю указать входные данные для расчета массы, как показано на рисунке 1; В частности, пользователь вводит однобуквенные аминокислотные последовательности для легких и тяжелых цепочек антител. Общие модификации для циклизации N-конца с тяжелой цепью и клипирования лизина C-концевой включены в качестве флажков. Кроме того, химическая формула/элементный состав могут быть добавлены/вычтены из этих белковых цепей через соответствующее текстовое поле Chem Mod . Это позволяет пользователю гибко добавлять элементный состав, который включает в себя несколько посттрансляционных модификаций или полезную нагрузку из малых молекул в случае АЦП. Поскольку большинство терапевтических мАТ спроектированы таким образом, чтобы удалять сайты гликозилирования в легкой цепи, гликозилирование в легкой цепи остается необязательным и может быть задано с помощью флажка в графическом интерфейсе.
Типичным вариантом анализа интактной массы на антитела является анализ приведенной субъединичной массы, при котором легкая цепь отделяется от тяжелой цепи путем уменьшения межцепочечных дисульфидных связей. В зависимости от силы используемого восстановителя внутрицепочечные дисульфидные связи могут расщепляться или не расщепляться. Пользователи могут вводить общее количество дисульфидных связей в зависимости от подтипа IgG или в случае цистеин-конъюгированного АЦП9.
Приложение рассчитывает массы по принципу «снизу вверх», при этом сначала рассчитываются элементные составы для отдельных тяжелых и легких цепей. Далее N-концевая циклизация тяжелой цепи (HC) Lys-клиппинг учитывается путем корректировки рассчитанного элементного состава. Любые указанные химические модификации затем наносятся на тяжелые и/или легкие цепи. В зависимости от типа анализа и характера дисульфидной связи, заданного пользователем, количество водорода корректируется для двух полипептидных цепей. Массы гликозилированного HC и легкой цепи (LC) (опционально) рассчитываются на основе вводимых пользователем данных. Наконец, несколько масс HC и LC объединяются, и номера дисульфидных связей автоматически обновляются для расчета неповрежденной массы.
С более крупными молекулами, такими как интактные белки, моноизотопные массы не могут быть измерены из-за дефекта аддитивной массы при использовании масс-спектрометров с типичной разрешающей способностью. Вместо этого измеряются номинальные или средние массы 5,10,11,12,13. Средние массы элементов могут варьироваться в зависимости от источника, используемого для курируемых масс14,15. Хотя различия в массах элементов могут быть небольшими, они могут складываться в значительные значения для расчетов молекулярной массы больших молекул. Средние массы элементов, используемые по умолчанию в программном приложении, приведены в Дополнительной таблице 1. Для регулируемых сред, таких как биофармацевтические исследования и разработки (НИОКР), важно поддерживать постоянную молекулярную массу, поскольку изменения масс могут повлечь за собой изменения молекулярной сущности во время подачи заявок в регулирующие органы. Чтобы обеспечить единообразие в использовании элементарных масс, словарь элементарных масс включен в программный инструмент в виде текстового файла с разделителями-запятыми (csv): Element_Mass.csv (Дополнительный файл кодирования 1). Аналогичным образом, включен курируемый список гликановых композиций, обычно встречающихся на мАТ: Glycan.csv (Дополнительный файл кодирования 2). Оба файла сохраняются в той же папке, что и исполняемое приложение, и могут быть изменены пользователем для использования определенного списка элементных масс или библиотеки гликанов.
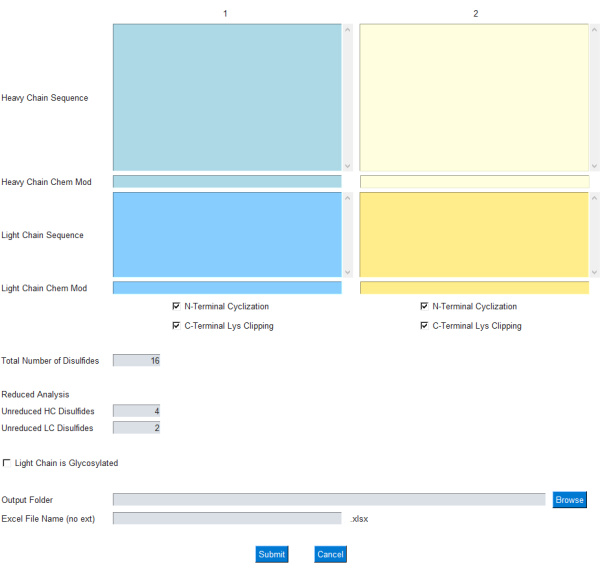
Рисунок 1: Графический интерфейс для приложения mAbScale. Модуль GUI позволяет пользователю указать входные данные для расчета массы. Пользователь вводит однобуквенные аминокислотные последовательности для легких и тяжелых цепочек антител. Общие модификации для циклизации N-конца тяжелой цепи и клипирования лизина C-концевой включены в качестве флажков. Химические формулы/элементные составы могут быть добавлены/вычтены через соответствующее текстовое поле Chem Mod . Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
Protocol
Representative Results
Discussion
mAbScale предоставляет интуитивно понятный пользовательский интерфейс с гибкостью для изменения строительных блоков для расчетов массы и элементов. Ожидается, что пользователи будут иметь базовое представление о целевой молекуле, чтобы использовать приложение, получать правильные масс…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Авторы благодарят Роберта Шустера за помощь в проверке данных.
Materials
| Acquity UPLC system | Waters Corp., Milford, MA | N/A | Modular system |
| Antibody-drug conjugate (ADC) | GlaxoSmithKline | N/A | Proprietory molecule |
| BEH 200 SEC column | Waters Corp., Milford, MA | 176003904 | |
| Bispecific mAb | GlaxoSmithKline | N/A | Proprietory molecule |
| Byos | Protein Metrics, Cupertino, CA | https://proteinmetrics.com/byos/ Version 4.5 |
|
| GPMAW | GPMAW | http://www.gpmaw.com/ | |
| LC-MS grade water | Thermo Fisher Scientific, Waltham, MA | W6-1 | |
| mAb standard | Waters Corp., Milford, MA | 186009125 | Waters Humanized mAb Mass Check Standard |
| mAbScale | GlaxoSmithKline | Apache License, Version 2.0 | |
| Xevo G2 Q-TOF mass spectrometer | Waters Corp., Milford, MA | N/A | Modular system |
Riferimenti
- Reichert, J. M., Valge-Archer, V. E. Development trends for monoclonal antibody cancer therapeutics. Nature Reviews Drug Discovery. 6 (5), 349-356 (2007).
- Kintzing, J. R., Filsinger Interrante, M. V., Cochran, J. R. Emerging strategies for developing next-generation protein therapeutics for cancer treatment. Trends in Pharmacological Sciences. 37 (12), 993-1008 (2016).
- Wang, M. -. Y., et al. SARS-CoV-2: Structure, biology, and structure-based therapeutics development. Frontiers in Cellular and Infection Microbiology. 10, 587269 (2020).
- ICH Q8 (R2) Pharmaceutical Development – Scientific Guideline. European Medicines Agency Available from: https://www.ema.europa.eu/en/ch-q8-r2-pharmaceutical-development-scientific-guideline (2018)
- Donnelly, D. P., et al. Best practices and benchmarks for intact protein analysis for top-down mass spectrometry. Nature Methods. 16 (7), 587-594 (2019).
- Gadgil, H. S., Pipes, G. D., Dillon, T. M., Treuheit, M. J., Bondarenko, P. V. Improving mass accuracy of high performance liquid chromatography/electrospray ionization time-of-flight mass spectrometry of intact antibodies. Journal of the American Society for Mass Spectrometry. 17 (6), 867-872 (2006).
- Beck, A., Sanglier-Cianférani, S., Van Dorsselaer, A. Biosimilar, biobetter, and next generation antibody characterization by mass spectrometry. Analytical Chemistry. 84 (11), 4637-4646 (2012).
- Camperi, J., Goyon, A., Guillarme, D., Zhang, K., Stella, C. Multi-dimensional LC-MS: the next generation characterization of antibody-based therapeutics by unified online bottom-up, middle-up and intact approaches. Analyst. 146 (3), 747-769 (2021).
- Liu, H., May, K. Disulfide bond structures of IgG molecules. mAbs. 4 (1), 17-23 (2012).
- Jakes, C., Füssl, F., Zaborowska, I., Bones, J. Rapid analysis of biotherapeutics using protein a chromatography coupled to orbitrap mass spectrometry. Analytical Chemistry. 93 (40), 13505-13512 (2021).
- Robotham, A. C., Kelly, J. F., Matte, A. Chapter 1 – LC-MS characterization of antibody-based therapeutics: Recent highlights and future prospects. Approaches to the Purification, Analysis and Characterization of Antibody-Based Therapeutics. , 1-33 (2020).
- Valeja, S. G., et al. Unit mass baseline resolution for an intact 148 kDa therapeutic monoclonal antibody by fourier transform ion cyclotron resonance mass spectrometry. Analytical Chemistry. 83 (22), 8391-8395 (2011).
- Fornelli, L., Ayoub, D., Aizikov, K., Beck, A., Tsybin, Y. O. Middle-down analysis of monoclonal antibodies with electron transfer dissociation orbitrap fourier transform mass spectrometry. Analytical Chemistry. 86 (6), 3005-3012 (2014).
- Berglund, M., Wieser, M. E. Isotopic compositions of the elements 2009 (IUPAC Technical Report). Pure and Applied Chemistry. 83 (2), 397-410 (2011).
- Wang, M., et al. The Ame2012 atomic mass evaluation. Chinese Physics C. 36 (12), 1603-2014 (2012).
- Peri, S., Steen, H., Pandey, A. GPMAW–A software tool for analyzing proteins and peptides. Trends in Biochemical Sciences. 26 (11), 687-689 (2001).
- Tipton, J. D., et al. Analysis of intact protein isoforms by mass spectrometry. The Journal of Biological Chemistry. 286 (29), 25451-25458 (2011).
- De Leoz, M. L. A., et al. interlaboratory study on glycosylation analysis of monoclonal antibodies: Comparison of results from diverse analytical methods. Molecular & Cellular Proteomics. 19 (1), 11-30 (2020).
- Cymer, F., Beck, H., Rohde, A., Reusch, D. Therapeutic monoclonal antibody N-glycosylation – Structure, function and therapeutic potential. Biologicals. 52, 1-11 (2018).
- Baker, P. R., Trinidad, J. C., Chalkley, R. J. Modification site localization scoring integrated into a search engine. Molecular & Cellular Proteomics. 10 (7), (2011).
- Chalkley, R. J., Clauser, K. R. Modification site localization scoring: Strategies and performance. Molecular & Cellular Proteomics. 11 (5), 3-14 (2012).

