Protocole générique pour l’optimisation de la Production de protéines hétérologues à l’aide de la technologie automatisée de MICROBIOREACTEUR
Summary
Ce manuscrit décrit une approche générique pour une conception sur mesure de supports de culture microbienne. Cette option est activée par un flux de travail itératif combinant krigeage experimental design et MICROBIOREACTEUR technologie pour un débit suffisant la culture, qui est pris en charge par robotics lab pour augmenter la fiabilité et la rapidité dans la manipulation des médias de liquides préparation.
Abstract
Des aspects essentiels en matière de biotechnologie industrielle utilisant des usines de cellules de production microbienne sont le processus itératif de l’ingénierie de la souche et l’optimisation des conditions de bioprocédés. Un aspect important est l’amélioration du milieu de culture pour fournir un environnement optimal pour la formation microbienne du produit d’intérêt. Il est bien établi que la composition de médias peut influencer considérablement la performance globale de bioprocédés. Optimisation moyenne nutrition est connue pour améliorer la protéine recombinante production avec les systèmes microbiens et donc, c’est une étape enrichissante dans le développement de bioprocédés. Toutefois, les recettes des médias très souvent standard proviennent de littérature, depuis la conception sur mesure de la milieu de culture est une tâche fastidieuse qui exige la technologie MICROBIOREACTEUR pour un débit suffisant culture, analytique rapide produit, ainsi que soutien par robotique de laboratoire pour permettre la fiabilité dans les étapes de manipulation de liquides. En outre, des méthodes mathématiques avancées sont nécessaires pour analyser rationnellement les données de mesure et concevoir efficacement des expériences parallèles comme pour atteindre le contenu de l’information optimale.
Le caractère générique du protocole présenté permet une adaptation facile au matériel de laboratoire différents hôtes de l’expression et autres protéines cibles d’intérêt, ainsi que d’autres paramètres de bioprocédés. En outre, autres objectifs d’optimisation comme taux de production de protéines, le rendement spécifique ou la qualité des produits peuvent être choisies pour s’adapter à la portée des autres études d’optimisation. La Toolbox de krigeage appliquée (KriKit) est un outil général pour conception d’expériences (DOE) qui contribue à l’optimisation de bioprocédés globale améliorée. Il soutient également l’optimisation multi-objectif qui peut être importante dans l’optimisation des processus en amont et en aval.
Introduction
Technologie moderne gène recombinant permet l’utilisation de techniques enzymes pour différentes applications dans l’industrie pharmaceutique, l’alimentation animale, chimie organique et l’Agroalimentaire1,2,3. La production d’enzymes techniques en vrac est un thème majeur pour la biotechnologie industrielle et la production de protéines recombinantes optimisé et les deux de la souche et génie des bioprocédés est nécessaire. Pour la génération des souches efficacement ingénierie production, différentes bibliothèques génétiques sont disponibles, par exemple, pour équilibré gene expression4 ou augmentation de la sécrétion efficacité5.
Corynebacterium glutamicum est un important producteur d’acides aminés à l’échelle industrielle6,7 et représente un hôte d’expressions non conventionnelles attrayant pour la production de sécrétion de protéines recombinantes8 ,,9. Voie de translocation à arginine (Tat) tant générales sécrétoire (s) sont présents dans c. glutamicum et ont été appliqués avec succès pour la sécrétion de protéine recombinante10. Une vaste expérience en génie des bioprocédés concernant la production d’acides aminés à l’échelle industrielle, ainsi que la capacité de sécréter des protéines g/L montants11 et grande robustesse concernant des inhomogénéités de bioprocédés trouvées à grande échelle cultures12,13, faire glutamicum c. un organisme de plate-forme prometteuse pour la production de sécrétion de protéines hétérologues à l’échelle industrielle.
Optimisation moyenne nutrition est connue pour améliorer la production de protéines recombinantes avec systèmes microbiens14,15,16,17 et par conséquent, l’adaptation du milieu la composition est une étape enrichissante en bioprocédés relativement à une productivité optimale18,19,20,21. D’intenses recherches sur l’application de plaques de microtitration (MTP) pour culture microbienne22,23,24 a ouvert la voie pour le développement et la conception de MTP pour culture microbienne25 ,26 et le développement de systèmes axés sur les MTP MICROBIOREACTEUR (MBR) avec suivi en ligne et de l’environnement de contrôle27,28. MBRs permettent une augmentation significative en matière de culture expérimentale. En outre, les systèmes MBR découlant des autres types de bioréacteurs, par exemple, les colonnes de bulles ou réservoir agité réacteurs, sont disponibles pour microbial bioprocess optimisation29,30,31, 32.
En règle générale, études d’optimisation de bénéficient d’un débit plus élevé expérimental, qui devient encore plus puissant en combinaison avec des méthodologies DOE, telles qu’évaluer les interactions entre variables de conception ou de réduire les espaces de grande dimension recherche. Par conséquent, l’utilisation combinée de systèmes MBR, l’automatisation de laboratoire et DOE s’est avérée pour être une méthode puissante en biotechnologie8,16,33,34,35.
Un protocole pour l’optimisation des médias est présenté combinant state-of-the-art lab d’automatisation, technologie MBR avec surveillance des processus en ligne et données orientées sur le krigeage analyse/experimental design. La méthodologie de krigeage est implémentée dans une boîte à outils MATLAB (« KriKit ») qui peut être téléchargé et utilisé gratuitement frais36. Comme exemple d’application, la maximisation de la production de protéine (GFP) sécrétrices fluorescentes vert avec c. glutamicum est montrée en optimisant la composition d’un milieu minimal CgXII. Titre de la GFP a été choisi comme l’objectif d’optimisation car il peut être quantifié facilement et elle est largement appliquée comme protéine modèle pour les études sur le MBR systèmes37,38,39.
Le cadre présenté est divisé en quatre étapes, qui sont illustrées dans la Figure 1. Les étapes sont indiquées par des cadres de la boîte et correspondent aux sections du protocole. La première étape (Figure 1 a) est de définir les objectifs du projet et de déterminer les méthodes requises. La combinaison de méthodologies DOE, la technologie MBR et automatisation de laboratoire permet un débit accru expérimental qui exige le traitement de données puissant. La deuxième étape (Figure 1 b) a pour but de détecter les variables de conception sensible (c’est-à-direles composants moyennes) avec une forte influence sur l’objectif d’optimisation. Cela conduit à un nombre réduit de variables de conception d’intérêt. La troisième étape (Figure 1) comprend une optimisation itérative pour une étude plus détaillée de la relation fonctionnelle entre les variables restantes de la conception et l’objectif d’intérêt. En utilisant le jeu de données successivement étendu, l’approche de krigeage est appliquée pour prédire le résultat expérimental aux endroits non mesurés. Le cycle itératif s’arrête dès que le modèle de krigeage prédit un optimum ou un plateau avec une précision suffisante. Les résultats sont vérifiés dans la quatrième étape (Figure 1), commençant par une autre analyse de sensibilité autour de l’optimum identifié. Si au départ, les composants insensibles se trouvent être insensible aussi dans la région optimale, il est raisonnable de supposer que c’est le cas au cours de la procédure d’optimisation itérative dans la troisième étape. Par la suite, il est conseillé de vérifier les résultats de l’optimisation par l’application de méthodes orthogonales, comme une analyse de l’activité ou SDS-Page.
Le caractère générique du protocole présenté permet adaptation facile au matériel de laboratoire différents, autres hôtes expression et protéines cibles de choix, mais aussi des bioprocédés autres variables comme la température de culture ou de la valeur pH.En outre, les autres objectifs d’optimisation comme taux de production de protéines, le rendement spécifique ou la qualité des produits peuvent être choisies pour s’adapter à la portée des autres études d’optimisation.
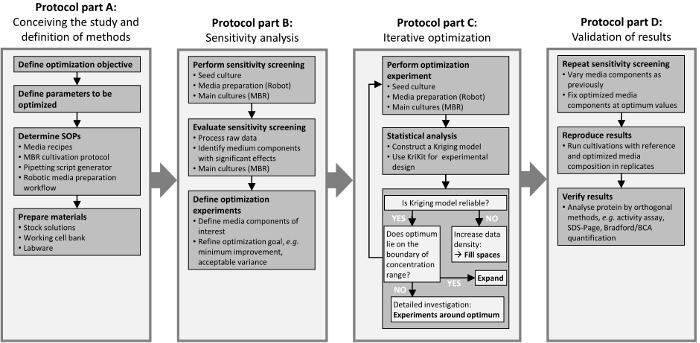
Figure 1 : Flux de travail d’étude d’optimisation. Les boîtes de quatre châssis correspondent aux sections du protocole, « Concevoir l’étude et définition de méthodes » (Section 1), « Sensitivity Analysis » (article 2), « Optimisation itérative » (article 3) et « Validation » (article 4). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Protocol
Representative Results
Discussion
Le caractère générique du protocole présenté permet diverses adaptations, par exemple, pour étudier d’autres expression microbienne hôtes9,47,48,49,50, 51, ou pour optimiser les autres propriétés de la protéine cible, comme les obligations de motif ou de bisulfure de glycosylation. Le protocole devrez peut-être aussi être adapté à l’équipement de laboratoire disponibles. L’intégration d’un système MBR permet d’augmenter le débit expérimental, qui permet d’importantes économies dans le temps. Cependant, lors du remplacement des bioréacteurs entièrement instrumentés et contrôlables par les systèmes MBR, évolutivité des résultats doit considérer8,37,52,53. L’utilisation de méthodologies DOE et la modélisation mathématique permet de maximiser l’information contenue dans les données de mesure en ce qui concerne l’ objectif étudiés54 par efficace de planification expérimentale et interprétation de données basées sur le modèle15.
Modifications à la méthode
À côté de polyvalent et extensible robotic manipulation de liquides systèmes comme celui utilisé dans cette étude, il convient de mentionner qu’il y a plusieurs liquides plus petits systèmes disponibles dans le commerce qui sont capables d’accomplir cette tâche et peuvent être placés à l’intérieur des bancs de travail à flux laminaire. Si aucun système de pipetage automatisée n’est disponible, des compositions de différents supports selon le régime DOE également peuvent être réalisées que par pipetage manuel à l’aide de pipettes unique ou multi canaux. Puisque la préparation manuelle est plus sujette aux erreurs et exigera des travaux très ciblé pour un temps assez long, il est conseillé de préparer un nombre inférieur de compositions de différents médias.
Selon les capacités du système MBR indépendant, le protocole de culture correspondant peut varier. Par exemple, si aucune mesure en ligne de formation de la biomasse est disponible, il peut être suffisant mesurer la concentration de la biomasse issue de l’expérience de la croissance. En combinaison avec un suivi en ligne du pH et l’oxygène dissous, qui est implémenté dans plusieurs systèmes de MBR, la saturation de la croissance peut être déterminée en toute sécurité. En principe, les expériences de croissance peuvent être effectuées en MTP seul placé à l’intérieur de secouer les incubateurs, sans l’utilisation d’un système MBR. Dans ce cas, les conditions de culture bon doivent être assurés : (1) cultures limitées en oxygène peuvent être évité en utilisant MTP avec des géométries adaptés, en combinaison avec bon secouant les fréquences et secouant les diamètres, par exemple, carré 96 ou 24 ” profondeur plaques bien exploité à 1 000 tr/min à 3 mm jet ou à 250 tr/min à 25 mm jet, respectivement. Ce qui est important, plus les taux de transfert d’oxygène maximale réalisable, plus la source principale de carbone devrait être concentrée. Comme mentionné ci-dessus, pour cette étude, l’utilisation de 10 g/L de glucose a été adaptée afin d’éviter la prescription d’oxygène pour les conditions de culture indépendants ; (2) l’échantillonnage des cultures du PSG pour la quantification de la biomasse et les produits doit être réduit au minimum. Chaque fois que le PSG est retiré de l’incubateur agitateur, transfert d’oxygène sera immédiatement panne qui peut aboutir à des conditions de culture défavorables ; (3) de l’avis des auteurs, l’utilisation de lecteurs du PSG comme dispositifs de culture n’est pas recommandée car ces appareils n’a été élaborés à cet effet. Par exemple, agitation mécanique ont été construite pour le mélange occasionnelle de microplaques après l’addition de réactif et ainsi, manque souvent de robustesse pour de longues distances de continu durée secouer pendant jours. En outre, suffisamment puissance d’entrée nécessaire pour les cultures microbiennes ne peut être réalisée dans ces lecteurs. L’intégration de lectures de densité optique en bref des intervalles de temps nécessite l’arrêt de la motion secousse, résultant en des périodes répétées de limitation de l’oxygène. En outre, évaporation dans de tels systèmes sur une période longue culture faussera les résultats. Pour plus de détails sur le sujet étonnamment complexe sur l’utilisation de MTP pour cultures microbiennes, on se reportera à la littérature citée22,23,24,25,26 et références.
D’autres considérations
Aux étapes d’optimisation itérative de Speed-up, il est conseillé de sélectionner avec soin la méthode d’analyse pour le dosage du produit. Rapides et simple des méthodes doivent être privilégiées au détriment de la précision et l’exactitude, comme la stratégie de conception expérimentale itératif tolère des imprécisions expérimentales. Toutefois, les résultats définitifs doivent être confrontées avec des méthodes de quantification suffisamment précise et exacte de produit qui pourraient être plus compliqués. En général, une évaluation soigneuse et prise de décisions sur les procédures d’étude nécessitent un effort au début de l’étude, mais payent à long terme, après que des méthodes de routine ont été établis.
Il est fortement recommandé de définir une expérience de référence qui est comparée à toutes les expériences pendant l’optimisation. Autrement dit, la composante moyenne appliquée concentrations ainsi que sortie mesuré est normalisée par divisant par des valeurs de référence. De cette façon, chacune appliquée et la valeur mesurée peut être interprété comme le x-giron de la valeur de référence. Afin de tenir compte des variations entre les plaques, cinq expériences de référence sont effectués sur chaque plaque. La valeur moyenne des résultats mesurés est utilisée pour la normalisation.
Il généralement ne peut être garantie que le milieu développé est également optimal pour les autres souches. Cependant, le support amélioré aussi sera probablement approprié pour la culture des souches d’expression avec de petites différences génétiques, par exemple, lors de produire des variantes de l’enzyme avec des substitutions d’acides aminés unique obtenue (études de mutagenèse même de simples mutations ponctuelles ont été décrits au métabolisme cellulaire effet et expression hétérologue performance55,,56). Dans ce cas, le protocole présenté peut être une première étape, suivie des protocoles haut débit expression projections57. Si le protocole est utilisé pour le développement moyen avec intensification ultérieure aux cultures fed-batch, le milieu optimisé doit être vérifié pour les conditions correspondantes de bioprocédés, comme clone dépistage des campagnes à la micro-échelle identifié différent haut artistes interprètes ou exécutants pour différentes stratégies d’alimentation et de la culture médiatique52,58. En outre, l’introduites KriKit36 peut généralement contribuer à optimisation améliorée bioprocédés holistique.Récemment encore, les capacités de l’outil ont été étendues pour prendre également en charge optimisation multi-objectif40, qui peut être important pour optimiser les processus en amont et en aval59,60.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Les activités scientifiques du Bioeconomy Science Center étaient soutenues financièrement par le ministère de l’Innovation, la Science et la recherche dans le cadre de la BioSC NRW-Strategieprojekt (no. 13 313/323-400-002). Les auteurs tiennent à remercier le ministère de l’Innovation, la Science et recherche de Rhénanie du Nord-Westphalie et l’Heinrich Heine Université Düsseldorf une bourse d’études pour Lars Freier au sein de la biotechnologie industrielle du Cluster CLIB-diplômé. Un financement supplémentaire a été reçu de l’activation du programme espaces « Helmholtz Innovation Labs » de l’Association allemande Helmholtz pour soutenir le « Microbial Bioprocess Lab – A Helmholtz Innovation Lab ».
Materials
| BioLector | m2p-labs | G-BL-100 | |
| Flowerplate | m2p-labs | MTP-48-BOH | For cultivation in the BioLector device |
| Sealing foil | m2p-labs | F-GP-10 | Sterile sealing for Flowerplate |
| MATLAB | Mathworks | 2016b | |
| KriKit | Forschungszentrum Jülich | n/a | Freely available, MATLAB installation required |
| Janus pipetting robot | Perkin Elmer | n/a | Includes "WinPrep" software installation |
| 12-column deep well microplate | E&K Scientific | EK-2034 | Container for medium stock solutions |
| 96 well microplates, transparent, F-bottom | Greiner | 655101 | For Bradford protein assay |
| µclear 96 well microplates, black body, transparent F-bottom | Greiner | 655087 | For flourescence measurement in cell-free supernatants |
| Pipette Research plus multi-channel pipettes | Eppendorf | n/a | Facilitates manual liquid handling with microplates |
| TruPAG Precast Gels | Sigma | PCG2002 | For SDS-Page analysis of cell-free supernantants |
| Bradford Reagent | Sigma | B6916 | |
| C. glutamicum pCGPhoDBs-GFP | n/a | n/a | Carries pEKEx2 plasmid with fusion of GFP gene and PhoD signal peptide from B.subtilis as expression insert. Plasmid provides kanamycin resistance. Described and published by Meissner et al. Appl Microbiol Biotechnol 76 (3), 633–42 (2007) |
References
- Choi, J. -. M., Han, S. -. S., Kim, H. -. S. Industrial applications of enzyme biocatalysis: Current status and future aspects. Biotechnol Adv. 33 (7), 1443-1454 (2015).
- Li, S., Yang, X., Yang, S., Zhu, M., Wang, X. Technology Prospecting on Enzymes: Application, Marketing and Engineering. Comput Struct Biotechnol J. 2 (3), (2012).
- Adrio, J. L., Demain, A. L. Microbial enzymes: tools for biotechnological processes. Biomolecules. 4 (1), 117-139 (2014).
- Yim, S. S., An, S. J., Kang, M., Lee, J., Jeong, K. J. Isolation of fully synthetic promoters for high-level gene expression in Corynebacterium glutamicum. Biotechnol Bioeng. 110 (11), 2959-2969 (2013).
- Hemmerich, J., et al. Use of a Sec signal peptide library from Bacillus subtilis.for the optimization of cutinase secretion in Corynebacterium glutamicum. Microb Cell Fact. 15 (1), 208 (2016).
- Wendisch, V. F. Microbial production of amino acids and derived chemicals: synthetic biology approaches to strain development. Curr Opin Biotechnol. 30, 51-58 (2014).
- Lee, J. -. Y., Na, Y. -. A., Kim, E., Lee, H. -. S., Kim, P. The Actinobacterium Corynebacterium glutamicum, an Industrial Workhorse. J Microbiol Biotechnol. 26 (5), 807-822 (2016).
- Rohe, P., Venkanna, D., Kleine, B., Freudl, R., Oldiges, M. An automated workflow for enhancing microbial bioprocess optimization on a novel microbioreactor platform. Microb Cell Fact. 11 (1), 144 (2012).
- Liu, X., et al. Expression of recombinant protein using Corynebacterium glutamicum.: progress, challenges and applications. Crit Rev Biotechnol. 36 (4), 652-664 (2016).
- Freudl, R., Burkovski, A. Corynebacterium glutamicum as a Platform Organism for the Secretory Production of Heterologous Proteins. Corynebacterium glutamicum: From systems biology to biotechnological applications. , 161-177 (2015).
- Yim, S. S., et al. Development of a new platform for secretory production of recombinant proteins in Corynebacterium glutamicum. Biotechnol Bioeng. 113 (1), 163-172 (2015).
- Limberg, M. H., et al. Metabolic profile of 1,5-diaminopentane producing Corynebacterium glutamicum under scale-down conditions: Blueprint for robustness to bioreactor inhomogeneities. Biotechnol Bioeng. 114 (3), 560-575 (2016).
- Käβ, F. Assessment of robustness against dissolved oxygen/substrate oscillations for C. glutamicum DM1933 in two-compartment bioreactor. Bioprocess Biosyst Eng. 37 (6), 1151-1162 (2014).
- Teramoto, H., Watanabe, K., Suzuki, N., Inui, M., Yukawa, H. High yield secretion of heterologous proteins in Corynebacterium glutamicum.using its own Tat-type signal sequence. Appl Microbiol Biotechnol. 91 (3), 677-687 (2011).
- Freier, L., Hemmerich, J., Schöler, K., Wiechert, W., Oldiges, M., von Lieres, E. Framework for Kriging-based iterative experimental analysis and design: Optimization of secretory protein production in Corynebacterium glutamicum. Eng Life Sci. 16 (6), 538-549 (2016).
- Huber, R., Roth, S., Rahmen, N., Büchs, J. Utilizing high-throughput experimentation to enhance specific productivity of an E. coli. T7 expression system by phosphate limitation. BMC Biotechnol. 11 (1), 22 (2011).
- Kottmeier, K., Müller, C., Huber, R., Büchs, J. Increased product formation induced by a directed secondary substrate limitation in a batch Hansenula polymorpha culture. Appl Microbiol Biotechnol. 86 (1), 93-101 (2010).
- Kennedy, M. J., Krouse, D. Strategies for improving fermentation medium performance: A review. J Ind Microbiol Biotechnol. 23 (6), 456-475 (1999).
- Kleman, G. L., Strohl, W. R. Developments in high cell density and high productivity microbial fermentation. Curr Opin Biotechnol. 5 (2), 180-186 (1994).
- Jones, R., Gadd, G. Ionic nutrition of yeast: Physiological mechanisms involved and implications for biotechnology. Enzy Microb Technol. 12 (6), 402-418 (1990).
- Zhang, J., Greasham, R. Chemically defined media for commercial fermentations. Appl Microbiol Biotechnol. 51 (4), 407-421 (1999).
- Kensy, F., et al. Oxygen transfer phenomena in 48-well microtiter plates: Determination by optical monitoring of sulfite oxidation and verification by real-time measurement during microbial growth. Biotechnol Bioeng. 89 (6), 698-708 (2005).
- Hermann, R., Lehmann, M., Büchs, J. Characterization of gas-liquid mass transfer phenomena in microtiter plates. Biotechnol Bioeng. 81 (2), 178-186 (2002).
- Duetz, W. A. Microtiter plates as mini-bioreactors: Miniaturization of fermentation methods. Trends Microbiol. 15 (10), 469-475 (2007).
- Funke, M., Diederichs, S., Kensy, F., Müller, C., Büchs, J. The baffled microtiter plate: Increased oxygen transfer and improved online monitoring in small scale fermentations. Biotechnol Bioeng. 103 (6), 1118-1128 (2009).
- Lattermann, C., Funke, M., Hansen, S., Diederichs, S., Büchs, J. Cross-section perimeter is a suitable parameter to describe the effects of different baffle geometries in shaken microtiter plates. J Biol Eng. 8, 18 (2014).
- Samorski, M., Müller-Newen, G., Büchs, J. Quasi-continuous combined scattered light and fluorescence measurements: A novel measurement technique for shaken microtiter plates. Biotechnol Bioeng. 92 (1), 61-68 (2005).
- Kensy, F., Zang, E., Faulhammer, C., Tan, R. -. K., Büchs, J. Validation of a high-throughput fermentation system based on online monitoring of biomass and fluorescence in continuously shaken microtiter plates. Microb Cell Fact. 8 (1), 31 (2009).
- Puskeiler, R., Kaufmann, K., Weuster-Botz, D. Development, parallelization, and automation of a gas-inducing milliliter-scale bioreactor for high-throughput bioprocess design (HTBD). Biotechnol Bioeng. 89 (5), 512-523 (2005).
- Bareither, R., Pollard, D. A review of advanced small-scale parallel bioreactor technology for accelerated process development: Current state and future need. Biotechnol Prog. 27 (1), 2-14 (2011).
- Hortsch, R., Stratmann, A., Weuster-Botz, D. New milliliter-scale stirred tank bioreactors for the cultivation of mycelium forming microorganisms. Biotechnol Bioeng. 106 (3), 443-451 (2010).
- Isett, K., George, H., Herber, W., Amanullah, A. Twenty-four-well plate miniature bioreactor high-throughput system: Assessment for microbial cultivations. Biotechnol Bioeng. 98 (5), 1017-1028 (2007).
- Motta Dos Santos, L. F., Coutte, F., Ravallec, R., Dhulster, P., Tournier-Couturier, L., Jacques, P. An improvement of surfactin production by B. subtilis BBG131 using design of experiments in microbioreactors and continuous process in bubbleless membrane bioreactor. Bioresour Technol. 218, 944-952 (2016).
- Islam, R. S., Tisi, D., Levy, M. S., Lye, G. J. Framework for the Rapid Optimization of Soluble Protein Expression in Escherichia coli Combining Microscale Experiments and Statistical Experimental Design. Biotechnol Prog. 23 (4), 785-793 (2007).
- Huber, R., et al. Robo-Lector: A novel platform for automated high-throughput cultivations in microtiter plates with high information content. Microb Cell Fact. 8 (1), 42 (2009).
- . Kriging toolKit (KriKit) Available from: https://github.com/modsim/KriKit (2017)
- Kensy, F., Engelbrecht, C., Büchs, J. Scale-up from microtiter plate to laboratory fermenter: evaluation by online monitoring techniques of growth and protein expression in Escherichia coli.and Hansenula polymorpha fermentations. Microb Cell Fact. 8 (1), 68 (2009).
- Lu, C., Bentley, W. E., Rao, G. A high-throughput approach to promoter study using green fluorescent protein. Biotechnol Prog. 20 (6), 1634-1640 (2004).
- Zanzotto, A., Boccazzi, P., Gorret, N., van Dyk, T. K., Sinskey, A. J., Jensen, K. F. In situ measurement of bioluminescence and fluorescence in an integrated microbioreactor. Biotechnol Bioeng. 93 (1), 40-47 (2006).
- Freier, L., von Lieres, E. Multi-Objective Global Optimization (MOGO): Algorithm and Case Study in Gradient Elution Chromatography. Biotechnol J. , (2016).
- Keilhauer, C., Eggeling, L., Sahm, H. Isoleucine synthesis in Corynebacterium glutamicum.: molecular analysis of the ilvB-ilvN-ilvC operon. J Bacteriol. 175 (17), 5595-5603 (1993).
- Weuster-Botz, D., Kelle, R., Frantzen, M., Wandrey, C. Substrate Controlled Fed-Batch Production of L-Lysine with Corynebacterium glutamicum. Biotechnol Prog. 13 (4), 387-393 (1997).
- Meissner, D., Vollstedt, A., van Dijl, J. M., Freudl, R. Comparative analysis of twin-arginine (Tat)-dependent protein secretion of a heterologous model protein (GFP) in three different Gram-positive bacteria. Appl Microbiol Biotechnol. 76 (3), 633-642 (2007).
- Morschett, H., Wiechert, W., Oldiges, M. Automation of a Nile red staining assay enables high throughput quantification of microalgal lipid production. Microb Cell Fact. 15, 34 (2016).
- Fisher, R. A. . The design of experiments. , (1935).
- Morschett, H., Freier, L., Rohde, J., Wiechert, W., von Lieres, E., Oldiges, M. A framework for accelerated phototrophic bioprocess development: Integration of parallelized microscale cultivation, laboratory automation and Kriging-assisted experimental design. Biotechnol Biofuels. 10, 26 (2017).
- Le Loir, Y., et al. Protein secretion in Lactococcus lactis: An efficient way to increase the overall heterologous protein production. Microb Cell Fact. 4 (1), 2 (2005).
- Anné, J., Vrancken, K., van Mellaert, L., van Impe, J., Bernaerts, K. Protein secretion biotechnology in Gram-positive bacteria with special emphasis on Streptomyces lividans. Biochim Biophys Acta. 1843 (8), 1750-1761 (2014).
- Gupta, S. K., Shukla, P. Advanced technologies for improved expression of recombinant proteins in bacteria: perspectives and applications. Crit Rev Biotechnol. 36 (6), 1089-1098 (2016).
- Fu, L. L., Xu, Z. R., Li, W. F., Shuai, J. B., Lu, P., Hu, C. X. Protein secretion pathways in Bacillus subtilis: implication for optimization of heterologous protein secretion. Biotechnol Adv. 25 (1), 1-12 (2007).
- Yoon, S., Kim, S., Kim, J. Secretory Production of Recombinant Proteins in Escherichia coli. Rec Pat Biotechnol. 4 (1), 23-29 (2010).
- Hemmerich, J., et al. Comprehensive clone screening and evaluation of fed-batch strategies in a microbioreactor and lab scale stirred tank bioreactor system: application on Pichia pastoris producing Rhizopus oryzae lipase. Microb Cell Fact. 13 (1), 36 (2014).
- Glazyrina, J., Krause, M., Junne, S., Glauche, F., Strom, D., Neubauer, P. Glucose-limited high cell density cultivations from small to pilot plant scale using an enzyme-controlled glucose delivery system. New Biotechnol. 29 (2), 235-242 (2012).
- Gernaey, K. V., et al. Monitoring and control of microbioreactors: An expert opinion on development needs. Biotechnol J. 7 (10), 1308-1314 (2012).
- Rahmen, N., Fulton, A., Ihling, N., Magni, M., Jaeger, K. -. E., Büchs, J. Exchange of single amino acids at different positions of a recombinant protein affects metabolic burden in Escherichia coli. Microb Cell Fact. 14, 10 (2015).
- Rahmen, N., et al. A particular silent codon exchange in a recombinant gene greatly influences host cell metabolic activity. Microb Cell Fact. 14, 156 (2015).
- Saez, N. J., Nozach, H., Blemont, M., Vincentelli, R. High throughput quantitative expression screening and purification applied to recombinant disulfide-rich venom proteins produced in E. coli. J Vis Exp. (89), e51464 (2014).
- Scheidle, M., et al. High-throughput screening of Hansenula polymorpha clones in the batch compared with the controlled-release fed-batch mode on a small scale. FEMS Yeast Res. 10 (1), 83-92 (2010).
- Baumann, P., Bluthardt, N., Renner, S., Burghardt, H., Osberghaus, A., Hubbuch, J. Integrated development of up- and downstream processes supported by the Cherry-Tag™ for real-time tracking of stability and solubility of proteins. J Biotechnol. 200, 27-37 (2015).
- Baumann, P., Hahn, T., Hubbuch, J. High-throughput micro-scale cultivations and chromatography modeling: Powerful tools for integrated process development. Biotechnol Bioeng. 112 (10), 2123-2133 (2015).

