Protocolo genérico para otimização da produção de proteína heteróloga usando tecnologia automatizada de Microbioreactor
Summary
Este manuscrito descreve uma abordagem genérica de design à medida dos meios de cultivo microbiano. Isso é ativado por um fluxo de trabalho iterativo combinando Kriging experimental design e microbioreactor tecnologia para throughput de cultivo suficientes, que é apoiado pela robótica de laboratório para aumentar a confiabilidade e a velocidade em mídia de manuseio de líquidos preparação.
Abstract
Um negócio em biotecnologia industrial usando fábricas de célula de produção microbiana é o processo iterativo de tensão de engenharia e otimização das condições de Bioprocessos. Um aspecto importante é a melhoria do meio de cultivo para fornecer um ambiente ideal para formação microbiana do produto de interesse. É bem aceite que a composição de mídia dramaticamente pode influenciar o desempenho geral de Bioprocessos. Otimização de média nutrição é conhecida por melhorar a proteína recombinante produção com sistemas microbianos e, portanto, este é um gratificante passo no desenvolvimento de Bioprocessos. No entanto, receitas de mídia padrão muitas vezes são tomadas a partir de literatura, desde o design à medida do meio de cultivo é uma tarefa tediosa que exige tecnologia microbioreactor para a taxa de transferência de cultivo suficientes, análise de produto rápido, bem como suporte por robótica de laboratório para permitir confiabilidade nas etapas de manuseio de líquidos. Além disso, métodos matemáticos avançados são necessários para racionalmente analisando dados de medição e projetar eficientemente experiências paralelas tais como conseguir conteúdo de informação ideal.
A natureza genérica do protocolo apresentado permite fácil adaptação ao equipamento de laboratório diferentes, outros hosts de expressão e proteínas alvo de interesse, bem como parâmetros de Bioprocessos ainda mais. Além disso, outros objetivos de otimização como taxa de produção de proteína, rendimento específico ou qualidade do produto podem ser escolhidos para caber o escopo de outros estudos de otimização. O Toolbox de Kriging aplicada (KriKit) é uma ferramenta geral para Design de experimentos (DOE) que contribui para a optimização de Bioprocessos holística melhorada. Ele também suporta otimização multi-objetiva que pode ser importante na otimização de processos de upstream e downstream.
Introduction
Tecnologia moderna genética recombinante permite a ampla utilização de enzimas técnicas para diversas aplicações na indústria farmacêutica, animal, alimentação, química orgânica e1,2,3de processamento de alimentos. A produção de enzimas técnicas em grandes quantidades é um tópico importante para a biotecnologia industrial e para a produção de proteínas recombinantes otimizado e ambos Coe e engenharia de Bioprocessos é necessária. Para a geração de cepas de produção eficientemente projetado, bibliotecas genéticas diferentes estão disponíveis, por exemplo, para a expressão de gene equilibrada4 ou aumento da secreção de eficiência5.
Corynebacterium glutamicum é um importante produtor de aminoácidos em escala industrial6,7 e representa um host atraente expressão não-convencionais para a produção de secreção de proteínas recombinantes8 ,9. O general secretora (Sec) e via de translocação gêmeo-arginina (Tat) estão presentes em c. glutamicum e foram aplicadas com êxito para a secreção de proteína recombinante10. Vasta experiência em engenharia de Bioprocessos em matéria de produção de aminoácidos em escala industrial, bem como a capacidade de secretar proteínas g/L de montantes11 e grande robustez relativa heterogeneidades de Bioprocessos, encontradas em larga escala cultivos12,13, faça glutamicum c. um organismo plataforma promissora para a produção de secreção de proteínas heterólogos em escala industrial.
Otimização de média nutrição é conhecida para melhorar a produção de proteína recombinante com sistemas microbianos14,15,16,17 e consequentemente, o ajustamento de médio composição é um passo gratificante em Bioprocessos desenvolvimento no que diz respeito a produtividade ideal18,19,20,21. Intensas pesquisas sobre a aplicação de placas de microtitulação (MTPs) para cultivo microbiano22,23,24 pavimentaram o caminho para o desenvolvimento e design de MTPs para cultivo microbiano25 ,26 e o desenvolvimento de sistemas baseados em MTP microbioreactor (MBR) com monitoramento on-line e ambiental controle27,28. MBR permite um aumento significativo na produtividade do cultivo experimental. Além disso, sistemas MBR decorrentes de outros tipos de biorreatores, por exemplo, colunas de bolhas ou mexido tanque reatores, estão disponíveis para Bioprocessos microbiana otimização29,30,31, 32.
Em geral, estudos de otimização beneficiam maior throughput experimental, que se torna ainda mais poderoso em combinação com metodologias DOE, como para avaliar as interações entre as variáveis de projeto ou reduzir os espaços de busca de alta dimensão. Consequentemente, o uso combinado de MBR sistemas, automação de laboratório e DOE provou para ser um método poderoso em biotecnologia8,16,33,34,35.
Apresenta-se um protocolo para otimização de mídia combinando a automação de laboratório de estado-da-arte, tecnologia MBR com acompanhamento de processo on-line e análise/experimental design Kriging-base de dados. A metodologia de Kriging é implementada em um Toolbox MATLAB (“KriKit”), que pode ser baixado e usado livre de carga36. Como exemplo de aplicação, maximização da produção de proteína (GFP) secretoras fluorescent verde com c. glutamicum é mostrada através da otimização da composição do meio mínimo de CgXII. Título GFP foi escolhido como o objetivo de otimização, como isso pode ser quantificado facilmente e é aplicado extensamente como proteína de modelo para estudos sobre MBR sistemas37,38,39.
O quadro apresentado é dividido em quatro etapas, que são ilustradas na Figura 1. As etapas são indicadas por quadros de caixa e correspondem às seções do protocolo. A primeira etapa (figura 1A) é definir os objetivos do projeto e para determinar os métodos necessários. A combinação de metodologias DOE, MBR tecnologia e automação de laboratório permite uma maior throughput experimental que exige poderosa de processamento de dados. A segunda etapa (figura 1B) visa detectar variáveis de projeto sensível (isto é, componentes médios) com alta influência sobre o objetivo de otimização. Isto leva a um número reduzido de variáveis de projeto de interesse. A terceira etapa (Figura 1) é composto por uma otimização iterativa para uma investigação mais detalhada da relação funcional entre as restantes variáveis de projeto e o objetivo de interesse. Usando o conjunto de dados sucessivamente estendido, a abordagem de Kriging é aplicada para prever o resultado experimental em locais não mensuráveis. O ciclo iterativo é interrompida assim que o modelo de Kriging prevê um ideal ou um planalto com uma precisão suficiente. Os resultados são verificados na quarta etapa (Figura 1), começando com uma nova análise de sensibilidade em torno do optimum identificado. Se inicialmente, diferenciação de componentes são encontrados para ser insensível também na região ideal, é razoável supor que isto prende verdadeiro durante o procedimento iterativo de otimização na terceira etapa. Depois disso, é aconselhável verificar resultados de otimização da aplicação dos métodos ortogonais, como um ensaio de atividade ou SDS-Page.
A natureza genérica do protocolo apresentado permite fácil adaptação ao equipamento de laboratório diferentes, outros hosts de expressão e proteínas alvo de escolha, bem como mais Bioprocessos variáveis como a temperatura de pH valor ou cultivo.Além disso, outros objetivos de otimização como taxa de produção de proteína, rendimento específico ou qualidade do produto podem ser escolhidos para caber o escopo de outros estudos de otimização.
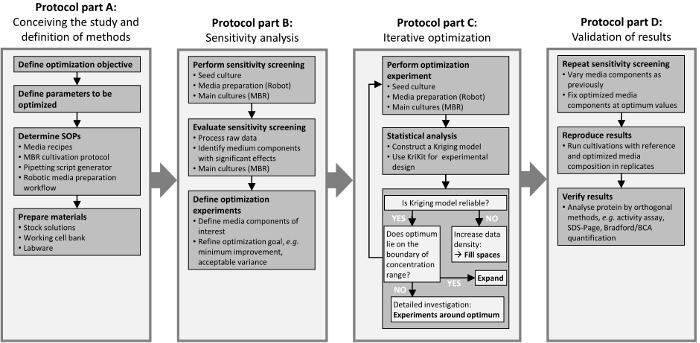
Figura 1 : Fluxo de trabalho de estudo de otimização. As caixas de quatro quadros correspondem às seções do protocolo, “Conceber o estudo e definição de métodos” (secção 1), “Análise de sensibilidade” (secção 2), “Optimization iterativo” (secção 3) e “Validação” (secção 4). Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
A natureza genérica do protocolo apresentado permite várias adaptações, por exemplo, para estudar a expressão microbiana anfitriões9,47,,48,49,50, 51, ou para otimizar a outras propriedades da proteína alvo, como títulos do padrão ou dissulfureto de glicosilação. O protocolo também pode precisar de ser adaptado para o equipamento de laboratório disponíveis. A integração de um sistema MBR permite aumentando o throughput experimental, que permite grande economia no tempo. No entanto, quando substituindo biorreatores totalmente instrumentados e controláveis pelos sistemas MBR, escalabilidade dos resultados deve ser considerada8,37,52,53. O uso de metodologias DOE e modelagem matemática ajuda a maximizar o conteúdo de informação de dados de medição em relação a objetiva estudados54 por eficiente planejamento experimental e de interpretação de dados baseados em modelo15.
Modificações para o método
Ao lado de múltiplos propósito e expansível robótico líquido manipulação de sistemas como o usado neste estudo, deve-se mencionar que existem vários líquido menor manipulação sistemas comercialmente disponíveis, que são capazes de executar esta tarefa e podem ser colocados dentro bancada de trabalho de fluxo laminar. Se nenhum sistema automatizado de pipetagem é disponível, composições de meios diferentes de acordo com o plano DOE também podem ser realizadas por pipetagem manual usando pipetas única e/ou multi-canal. Desde a preparação manual é mais propenso a erros e exigirá trabalho altamente concentrado por muito tempo, é aconselhável preparar um número menor de composições diferentes mídias.
Dependendo dos recursos do sistema MBR independente, o protocolo de cultivo correspondente irá variar. Por exemplo, se nenhuma medição on-line de formação de biomassa está disponível, pode ser suficiente medir a concentração de biomassa após a conclusão do experimento crescimento. Em combinação com monitoramento on-line do pH e oxigênio dissolvido, que é implementado em vários sistemas MBR, a saturação de crescimento pode ser determinada com segurança. Em princípio, as experiências de crescimento podem ser conduzidas no MTPs sozinho colocados dentro de incubadoras, sem o uso de um sistema MBR a tremer. Neste caso, as condições de cultivo adequado tem que ser assegurada: cultivos de oxigênio-limitada (1) podem ser evitado pelo uso do MSDN com geometrias adequadas, em combinação com adequada frequências a tremer e tremer diâmetros, por exemplo, quadrado 96 ou 24 profunda placas bem operado a 1.000 rpm em jogar apenas 3 mm ou a 250 rpm em passos de 25 mm, respectivamente. Importante, quanto mais baixo as taxas de transferência de oxigênio máxima atingível, menor a fonte principal de carbono deve ser concentrada. Como mencionado acima, para este estudo, o uso de glicose 10 g/L foi adequado para evitar a limitação de oxigênio para as condições de cultivo empregados; (2) a amostragem das culturas MTP para quantificação de biomassa e o produto deve ser reduzida ao mínimo. Cada vez que o MTP é retirado da incubadora tremendo, transferência de oxigênio será imediatamente avaria que pode resultar em condições desfavoráveis de cultivo; (3) na opinião dos autores, o uso de leitores MTP como dispositivos de cultivo não é recomendado como esses dispositivos não foram desenvolvidos para esta finalidade. Por exemplo, agitação mecânica foram construídas para a mistura ocasionais de microplacas após a adição do reagente e, muitas vezes falta-lhes robustez para corridas longas de duração agitação contínua por dias. Além disso, entrada de alimentação suficiente necessária para cultivos microbianos não pode ser realizada nesses leitores. A integração das leituras de densidade óptica em breve intervalos de tempo requer parada do movimento de agitação, resultando em períodos repetidos de limitação de oxigênio. Além disso, a evaporação em tais sistemas, durante períodos de longo período de cultivo vai distorcer os resultados. Para obter mais detalhes sobre o tema surpreendentemente complexo sobre o uso do MSDN para cultivos microbianos, o leitor é referido a literatura citada22,23,24,25,26 e referências ali.
Considerações adicionais
A passos de otimização iterativo de acelerar, é aconselhável selecionar cuidadosamente o método analítico para quantificação do produto. Rápido e simples métodos devem ser preferidos ao custo de precisão e exatidão, como a estratégia de projeto experimental iterativo tolera imprecisão experimental. No entanto, os resultados finais devem ser cotejados com métodos de quantificação do produto suficientemente precisa e exata que pode ser mais complicado. Em geral, cuidadosa avaliação e tomada de decisão sobre os procedimentos de estudo requerem esforço no início do estudo, mas pagam a longo prazo, depois que se estabeleceram métodos de rotina.
É altamente recomendável para definir um experimento de referência é comparado a todas as experiências durante a otimização. Ou seja, as concentrações de componente média aplicada, bem como saída medida é normalizada através de dividindo por valores de referência. Desta forma, cada um aplicado e o valor medido pode ser interpretado como a x-dobra o valor de referência. Para tomar em conta as variações entre as placas, cinco experimentos de referência são executados em cada prato. O valor médio dos resultados medidos é usado para a normalização.
Ele pode geralmente não ser garantido que o médio desenvolvido também é ideal para outras tensões. No entanto, o melhor médio provavelmente também será apropriado para cultivar variedades de expressão com pequenas diferenças genéticas, por exemplo, quando produzir variantes da enzima com substituições de aminoácidos única obtida (estudos de mutagênese Embora ainda única ponto foram descritas mutações para o metabolismo celular de efeito e expressão heteróloga desempenho55,56). Neste caso, o protocolo apresentado pode ser um primeiro passo, seguido por protocolos de expressão do elevado-throughput seleções57. Se o protocolo é usado para o desenvolvimento médio com escala-up subsequente para cultivos alimentados em lotes, o médio otimizado deve ser verificado para as respectivas condições de Bioprocessos, clone rastreio campanhas na microescala identificados diferente top artistas de diferentes estratégias de alimentação e de52,de meios de cultivo58. Além disso, o Danilo introduzido36 geralmente pode contribuir para otimização de Bioprocessos holística melhorada.Apenas recentemente, as capacidades da ferramenta foram estendidas para também oferecer suporte a multi-objetivo de otimização40, que pode ser importante para otimizar os dois processos de upstream e downstream59,60.
Disclosures
The authors have nothing to disclose.
Acknowledgements
As atividades científicas do centro de ciência de bioeconomia foram apoiadas financeiramente pelo Ministério da inovação, ciência e pesquisa no âmbito da BioSC NRW-Strategieprojekt (n º 313/323-400-002-13). Os autores agradecer o Ministério da inovação, ciência e pesquisa do Norte-Vestfália e a Heinrich Heine University Düsseldorf para uma bolsa de estudos para Lars Freier dentro Cluster CLIB-pós-graduação Biotecnologia Industrial. Mais financiamento foi recebido do programa espaços habilitando “Laboratórios de inovação de Helmholtz” da associação alemã de Helmholtz para apoiar o “microbiana Bioprocessos laboratório – A Helmholtz laboratório de inovação”.
Materials
| BioLector | m2p-labs | G-BL-100 | |
| Flowerplate | m2p-labs | MTP-48-BOH | For cultivation in the BioLector device |
| Sealing foil | m2p-labs | F-GP-10 | Sterile sealing for Flowerplate |
| MATLAB | Mathworks | 2016b | |
| KriKit | Forschungszentrum Jülich | n/a | Freely available, MATLAB installation required |
| Janus pipetting robot | Perkin Elmer | n/a | Includes "WinPrep" software installation |
| 12-column deep well microplate | E&K Scientific | EK-2034 | Container for medium stock solutions |
| 96 well microplates, transparent, F-bottom | Greiner | 655101 | For Bradford protein assay |
| µclear 96 well microplates, black body, transparent F-bottom | Greiner | 655087 | For flourescence measurement in cell-free supernatants |
| Pipette Research plus multi-channel pipettes | Eppendorf | n/a | Facilitates manual liquid handling with microplates |
| TruPAG Precast Gels | Sigma | PCG2002 | For SDS-Page analysis of cell-free supernantants |
| Bradford Reagent | Sigma | B6916 | |
| C. glutamicum pCGPhoDBs-GFP | n/a | n/a | Carries pEKEx2 plasmid with fusion of GFP gene and PhoD signal peptide from B.subtilis as expression insert. Plasmid provides kanamycin resistance. Described and published by Meissner et al. Appl Microbiol Biotechnol 76 (3), 633–42 (2007) |
References
- Choi, J. -. M., Han, S. -. S., Kim, H. -. S. Industrial applications of enzyme biocatalysis: Current status and future aspects. Biotechnol Adv. 33 (7), 1443-1454 (2015).
- Li, S., Yang, X., Yang, S., Zhu, M., Wang, X. Technology Prospecting on Enzymes: Application, Marketing and Engineering. Comput Struct Biotechnol J. 2 (3), (2012).
- Adrio, J. L., Demain, A. L. Microbial enzymes: tools for biotechnological processes. Biomolecules. 4 (1), 117-139 (2014).
- Yim, S. S., An, S. J., Kang, M., Lee, J., Jeong, K. J. Isolation of fully synthetic promoters for high-level gene expression in Corynebacterium glutamicum. Biotechnol Bioeng. 110 (11), 2959-2969 (2013).
- Hemmerich, J., et al. Use of a Sec signal peptide library from Bacillus subtilis.for the optimization of cutinase secretion in Corynebacterium glutamicum. Microb Cell Fact. 15 (1), 208 (2016).
- Wendisch, V. F. Microbial production of amino acids and derived chemicals: synthetic biology approaches to strain development. Curr Opin Biotechnol. 30, 51-58 (2014).
- Lee, J. -. Y., Na, Y. -. A., Kim, E., Lee, H. -. S., Kim, P. The Actinobacterium Corynebacterium glutamicum, an Industrial Workhorse. J Microbiol Biotechnol. 26 (5), 807-822 (2016).
- Rohe, P., Venkanna, D., Kleine, B., Freudl, R., Oldiges, M. An automated workflow for enhancing microbial bioprocess optimization on a novel microbioreactor platform. Microb Cell Fact. 11 (1), 144 (2012).
- Liu, X., et al. Expression of recombinant protein using Corynebacterium glutamicum.: progress, challenges and applications. Crit Rev Biotechnol. 36 (4), 652-664 (2016).
- Freudl, R., Burkovski, A. Corynebacterium glutamicum as a Platform Organism for the Secretory Production of Heterologous Proteins. Corynebacterium glutamicum: From systems biology to biotechnological applications. , 161-177 (2015).
- Yim, S. S., et al. Development of a new platform for secretory production of recombinant proteins in Corynebacterium glutamicum. Biotechnol Bioeng. 113 (1), 163-172 (2015).
- Limberg, M. H., et al. Metabolic profile of 1,5-diaminopentane producing Corynebacterium glutamicum under scale-down conditions: Blueprint for robustness to bioreactor inhomogeneities. Biotechnol Bioeng. 114 (3), 560-575 (2016).
- Käβ, F. Assessment of robustness against dissolved oxygen/substrate oscillations for C. glutamicum DM1933 in two-compartment bioreactor. Bioprocess Biosyst Eng. 37 (6), 1151-1162 (2014).
- Teramoto, H., Watanabe, K., Suzuki, N., Inui, M., Yukawa, H. High yield secretion of heterologous proteins in Corynebacterium glutamicum.using its own Tat-type signal sequence. Appl Microbiol Biotechnol. 91 (3), 677-687 (2011).
- Freier, L., Hemmerich, J., Schöler, K., Wiechert, W., Oldiges, M., von Lieres, E. Framework for Kriging-based iterative experimental analysis and design: Optimization of secretory protein production in Corynebacterium glutamicum. Eng Life Sci. 16 (6), 538-549 (2016).
- Huber, R., Roth, S., Rahmen, N., Büchs, J. Utilizing high-throughput experimentation to enhance specific productivity of an E. coli. T7 expression system by phosphate limitation. BMC Biotechnol. 11 (1), 22 (2011).
- Kottmeier, K., Müller, C., Huber, R., Büchs, J. Increased product formation induced by a directed secondary substrate limitation in a batch Hansenula polymorpha culture. Appl Microbiol Biotechnol. 86 (1), 93-101 (2010).
- Kennedy, M. J., Krouse, D. Strategies for improving fermentation medium performance: A review. J Ind Microbiol Biotechnol. 23 (6), 456-475 (1999).
- Kleman, G. L., Strohl, W. R. Developments in high cell density and high productivity microbial fermentation. Curr Opin Biotechnol. 5 (2), 180-186 (1994).
- Jones, R., Gadd, G. Ionic nutrition of yeast: Physiological mechanisms involved and implications for biotechnology. Enzy Microb Technol. 12 (6), 402-418 (1990).
- Zhang, J., Greasham, R. Chemically defined media for commercial fermentations. Appl Microbiol Biotechnol. 51 (4), 407-421 (1999).
- Kensy, F., et al. Oxygen transfer phenomena in 48-well microtiter plates: Determination by optical monitoring of sulfite oxidation and verification by real-time measurement during microbial growth. Biotechnol Bioeng. 89 (6), 698-708 (2005).
- Hermann, R., Lehmann, M., Büchs, J. Characterization of gas-liquid mass transfer phenomena in microtiter plates. Biotechnol Bioeng. 81 (2), 178-186 (2002).
- Duetz, W. A. Microtiter plates as mini-bioreactors: Miniaturization of fermentation methods. Trends Microbiol. 15 (10), 469-475 (2007).
- Funke, M., Diederichs, S., Kensy, F., Müller, C., Büchs, J. The baffled microtiter plate: Increased oxygen transfer and improved online monitoring in small scale fermentations. Biotechnol Bioeng. 103 (6), 1118-1128 (2009).
- Lattermann, C., Funke, M., Hansen, S., Diederichs, S., Büchs, J. Cross-section perimeter is a suitable parameter to describe the effects of different baffle geometries in shaken microtiter plates. J Biol Eng. 8, 18 (2014).
- Samorski, M., Müller-Newen, G., Büchs, J. Quasi-continuous combined scattered light and fluorescence measurements: A novel measurement technique for shaken microtiter plates. Biotechnol Bioeng. 92 (1), 61-68 (2005).
- Kensy, F., Zang, E., Faulhammer, C., Tan, R. -. K., Büchs, J. Validation of a high-throughput fermentation system based on online monitoring of biomass and fluorescence in continuously shaken microtiter plates. Microb Cell Fact. 8 (1), 31 (2009).
- Puskeiler, R., Kaufmann, K., Weuster-Botz, D. Development, parallelization, and automation of a gas-inducing milliliter-scale bioreactor for high-throughput bioprocess design (HTBD). Biotechnol Bioeng. 89 (5), 512-523 (2005).
- Bareither, R., Pollard, D. A review of advanced small-scale parallel bioreactor technology for accelerated process development: Current state and future need. Biotechnol Prog. 27 (1), 2-14 (2011).
- Hortsch, R., Stratmann, A., Weuster-Botz, D. New milliliter-scale stirred tank bioreactors for the cultivation of mycelium forming microorganisms. Biotechnol Bioeng. 106 (3), 443-451 (2010).
- Isett, K., George, H., Herber, W., Amanullah, A. Twenty-four-well plate miniature bioreactor high-throughput system: Assessment for microbial cultivations. Biotechnol Bioeng. 98 (5), 1017-1028 (2007).
- Motta Dos Santos, L. F., Coutte, F., Ravallec, R., Dhulster, P., Tournier-Couturier, L., Jacques, P. An improvement of surfactin production by B. subtilis BBG131 using design of experiments in microbioreactors and continuous process in bubbleless membrane bioreactor. Bioresour Technol. 218, 944-952 (2016).
- Islam, R. S., Tisi, D., Levy, M. S., Lye, G. J. Framework for the Rapid Optimization of Soluble Protein Expression in Escherichia coli Combining Microscale Experiments and Statistical Experimental Design. Biotechnol Prog. 23 (4), 785-793 (2007).
- Huber, R., et al. Robo-Lector: A novel platform for automated high-throughput cultivations in microtiter plates with high information content. Microb Cell Fact. 8 (1), 42 (2009).
- . Kriging toolKit (KriKit) Available from: https://github.com/modsim/KriKit (2017)
- Kensy, F., Engelbrecht, C., Büchs, J. Scale-up from microtiter plate to laboratory fermenter: evaluation by online monitoring techniques of growth and protein expression in Escherichia coli.and Hansenula polymorpha fermentations. Microb Cell Fact. 8 (1), 68 (2009).
- Lu, C., Bentley, W. E., Rao, G. A high-throughput approach to promoter study using green fluorescent protein. Biotechnol Prog. 20 (6), 1634-1640 (2004).
- Zanzotto, A., Boccazzi, P., Gorret, N., van Dyk, T. K., Sinskey, A. J., Jensen, K. F. In situ measurement of bioluminescence and fluorescence in an integrated microbioreactor. Biotechnol Bioeng. 93 (1), 40-47 (2006).
- Freier, L., von Lieres, E. Multi-Objective Global Optimization (MOGO): Algorithm and Case Study in Gradient Elution Chromatography. Biotechnol J. , (2016).
- Keilhauer, C., Eggeling, L., Sahm, H. Isoleucine synthesis in Corynebacterium glutamicum.: molecular analysis of the ilvB-ilvN-ilvC operon. J Bacteriol. 175 (17), 5595-5603 (1993).
- Weuster-Botz, D., Kelle, R., Frantzen, M., Wandrey, C. Substrate Controlled Fed-Batch Production of L-Lysine with Corynebacterium glutamicum. Biotechnol Prog. 13 (4), 387-393 (1997).
- Meissner, D., Vollstedt, A., van Dijl, J. M., Freudl, R. Comparative analysis of twin-arginine (Tat)-dependent protein secretion of a heterologous model protein (GFP) in three different Gram-positive bacteria. Appl Microbiol Biotechnol. 76 (3), 633-642 (2007).
- Morschett, H., Wiechert, W., Oldiges, M. Automation of a Nile red staining assay enables high throughput quantification of microalgal lipid production. Microb Cell Fact. 15, 34 (2016).
- Fisher, R. A. . The design of experiments. , (1935).
- Morschett, H., Freier, L., Rohde, J., Wiechert, W., von Lieres, E., Oldiges, M. A framework for accelerated phototrophic bioprocess development: Integration of parallelized microscale cultivation, laboratory automation and Kriging-assisted experimental design. Biotechnol Biofuels. 10, 26 (2017).
- Le Loir, Y., et al. Protein secretion in Lactococcus lactis: An efficient way to increase the overall heterologous protein production. Microb Cell Fact. 4 (1), 2 (2005).
- Anné, J., Vrancken, K., van Mellaert, L., van Impe, J., Bernaerts, K. Protein secretion biotechnology in Gram-positive bacteria with special emphasis on Streptomyces lividans. Biochim Biophys Acta. 1843 (8), 1750-1761 (2014).
- Gupta, S. K., Shukla, P. Advanced technologies for improved expression of recombinant proteins in bacteria: perspectives and applications. Crit Rev Biotechnol. 36 (6), 1089-1098 (2016).
- Fu, L. L., Xu, Z. R., Li, W. F., Shuai, J. B., Lu, P., Hu, C. X. Protein secretion pathways in Bacillus subtilis: implication for optimization of heterologous protein secretion. Biotechnol Adv. 25 (1), 1-12 (2007).
- Yoon, S., Kim, S., Kim, J. Secretory Production of Recombinant Proteins in Escherichia coli. Rec Pat Biotechnol. 4 (1), 23-29 (2010).
- Hemmerich, J., et al. Comprehensive clone screening and evaluation of fed-batch strategies in a microbioreactor and lab scale stirred tank bioreactor system: application on Pichia pastoris producing Rhizopus oryzae lipase. Microb Cell Fact. 13 (1), 36 (2014).
- Glazyrina, J., Krause, M., Junne, S., Glauche, F., Strom, D., Neubauer, P. Glucose-limited high cell density cultivations from small to pilot plant scale using an enzyme-controlled glucose delivery system. New Biotechnol. 29 (2), 235-242 (2012).
- Gernaey, K. V., et al. Monitoring and control of microbioreactors: An expert opinion on development needs. Biotechnol J. 7 (10), 1308-1314 (2012).
- Rahmen, N., Fulton, A., Ihling, N., Magni, M., Jaeger, K. -. E., Büchs, J. Exchange of single amino acids at different positions of a recombinant protein affects metabolic burden in Escherichia coli. Microb Cell Fact. 14, 10 (2015).
- Rahmen, N., et al. A particular silent codon exchange in a recombinant gene greatly influences host cell metabolic activity. Microb Cell Fact. 14, 156 (2015).
- Saez, N. J., Nozach, H., Blemont, M., Vincentelli, R. High throughput quantitative expression screening and purification applied to recombinant disulfide-rich venom proteins produced in E. coli. J Vis Exp. (89), e51464 (2014).
- Scheidle, M., et al. High-throughput screening of Hansenula polymorpha clones in the batch compared with the controlled-release fed-batch mode on a small scale. FEMS Yeast Res. 10 (1), 83-92 (2010).
- Baumann, P., Bluthardt, N., Renner, S., Burghardt, H., Osberghaus, A., Hubbuch, J. Integrated development of up- and downstream processes supported by the Cherry-Tag™ for real-time tracking of stability and solubility of proteins. J Biotechnol. 200, 27-37 (2015).
- Baumann, P., Hahn, T., Hubbuch, J. High-throughput micro-scale cultivations and chromatography modeling: Powerful tools for integrated process development. Biotechnol Bioeng. 112 (10), 2123-2133 (2015).

