Protocollo generico per l'ottimizzazione della produzione di proteine eterologhe utilizzando tecnologia automatizzata di Microbioreactor
Summary
Questo manoscritto descrive un approccio generico per la progettazione su misura di media di coltura microbica. Questa opzione è attivata da un flusso di lavoro iterativo che unisce Kriging sperimentale design e microbioreactor tecnologia per una velocità effettiva coltivazione sufficiente, che è supportata da robotica lab per aumentare affidabilità e velocità nella gestione dei mezzi liquidi preparazione.
Abstract
Un core business in biotecnologie industriali utilizzando fabbriche di produzione microbica delle cellule è il processo iterativo di ceppo ingegneria e ottimizzazione delle condizioni di bioprocessi. Un aspetto importante è il miglioramento di medium di coltivazione per fornire un ambiente ottimale per la formazione microbica del prodotto di interesse. È ben accettato che la composizione media può influire notevolmente le prestazioni complessive di bioprocessi. Ottimizzazione media nutrizione è noto per migliorare la proteina ricombinante produzione con sistemi microbici e, quindi, questo è un gratificante passo nello sviluppo di bioprocessi. Tuttavia, ricette molto spesso standard multimediali sono tratti dalla letteratura, poiché la progettazione su misura di medium di coltivazione è un compito noioso che richiede tecnologia microbioreactor per una velocità effettiva coltivazione sufficiente, analitica rapida del prodotto, così come supporto di robotica lab per abilitare affidabilità nelle operazioni di gestione dei liquidi. Inoltre, metodi matematici avanzati sono necessari per analizzare razionalmente i dati di misura e in modo efficiente progettazione di esperimenti paralleli come raggiungere contenuto informativo ottimale.
Il carattere generico del protocollo presentato consente facile adattamento alla strumentazione di laboratorio diversi, altri host espressione e proteine bersaglio di interesse, così come ulteriori parametri di bioprocessi. Inoltre, altri obiettivi di ottimizzazione come tasso di produzione di proteina, resa specifica o qualità del prodotto possono essere scelti per adattarsi l’ambito di altri studi di ottimizzazione. Il Toolbox di Kriging applicata (KriKit) è uno strumento generale per progettazione di esperimenti (DOE) che contribuisce alla ottimizzazione migliorata bioprocessi olistico. Supporta anche ottimizzazione multi-obiettivo che può essere importante nell’ottimizzare processi upstream e downstream.
Introduction
La tecnologia moderna gene ricombinante consente l’ampio uso di enzimi tecnici per varie applicazioni nell’industria farmaceutica, zootecnia, chimica organica e1,2,3di trasformazione alimentare. La produzione di enzimi tecnici allo stato sfuso è un argomento importante per la biotecnologia industriale e per la produzione di proteine ricombinanti ottimizzato ed entrambi colare e ingegneria di bioprocess è necessaria. Per la generazione di ceppi ingegnerizzati in modo efficiente produzione, librerie genetiche differenti sono disponibili, ad esempio, per equilibrato gene expression4 o secrezione aumentata efficienza5.
Glutamicum del corinebatterio è un importante produttore di aminoacidi alla scala industriale6,7 e rappresenta un host attraente espressione non convenzionali per la produzione di secrezione di proteine ricombinanti8 ,9. Sia il generale secretiva (Sec) e la via di twin-arginina traslocazione (Tat) sono presenti in c. glutamicum e sono state applicate correttamente per proteina ricombinante secrezione10. Vasta esperienza nell’ingegneria dei bioprocessi per quanto riguarda la produzione dell’amminoacido a scala industriale, come pure la capacità di secernere proteine g/L importi11 e grande robustezza relativa disomogeneità di bioprocessi trovato in larga scala coltivazioni12,13, effettuare c. glutamicum un organismo piattaforma promettente per la produzione di secrezione delle proteine eterologhe in scala industriale.
Ottimizzazione media nutrizione è noto per migliorare la produzione di proteine ricombinanti con sistemi microbici14,15,16,17 e di conseguenza, la regolazione del mezzo composizione è un passo gratificante in bioprocessi sviluppo per quanto riguarda la produttività ottimale18,19,20,21. Intense ricerche sull’applicazione delle piastre di microtitolazione (MTPs) per coltura microbica22,23,24 ha spianato la strada per lo sviluppo e la progettazione di MTPs per coltura microbica25 ,26 e lo sviluppo di sistemi basati su MTP microbioreactor (MBR) con monitoraggio on-line e ambientale controllo27,28. MBR permettono un significativo aumento della velocità effettiva coltivazione sperimentale. Inoltre, sono disponibili per bioprocessi microbica ottimizzazione29,30,31, sistemi MBR derivanti da altri tipi di bioreattori, ad esempio, bolla colonne o agitata serbatoio reattori, 32.
In generale, gli studi di ottimizzazione beneficiano della maggiore velocità effettiva sperimentale, che diventa ancora più potente in combinazione con metodologie DOE, ad esempio per valutare le interazioni tra le variabili di progetto o ridurre gli spazi di ricerca alto-dimensionali. Di conseguenza, l’uso combinato di sistemi MBR, automazione di laboratorio e DOE ha dimostrato di essere un metodo efficace nella biotecnologia8,16,33,34,35.
Un protocollo per l’ottimizzazione dei media è presentato combinando automazione di laboratorio state-of-the-art, tecnologia MBR con monitoraggio online di processo e progettazione sperimentale/analisi dati basati su Kriging. La metodologia di Kriging è implementata in una Toolbox di MATLAB (“KriKit”) che può essere scaricato e utilizzato gratuitamente carica36. Come esempio di applicazione, massimizzazione della produzione di proteine (GFP) secretiva fluo verde con c. glutamicum è mostrato ottimizzando la composizione del terreno minimo CgXII. Titolo GFP è stato scelto come l’obiettivo di ottimizzazione come esso può essere facilmente quantificato e ampiamente si applica come proteina di modello per lo studio del MBR sistemi37,38,39.
Il quadro presentato è diviso in quattro fasi, che sono illustrati nella Figura 1. I passaggi sono indicati da cornici di casella e corrispondono alle sezioni del protocollo. Il primo passo (Figura 1A) è di definire gli obiettivi di progetto e di determinare i metodi richiesti. La combinazione di metodologie DOE, tecnologia MBR e automazione di laboratorio consente un aumentare il throughput sperimentale che richiede potente elaborazione di dati. Il secondo passo (Figura 1B) mira a rilevare le variabili design sensibile (cioè, componenti medie) con alta influenza sull’obiettivo ottimizzazione. Questo porta ad un ridotto numero di variabili di progetto di interesse. Il terzo passo (Figura 1) comprende un’ottimizzazione iterativa per una ricerca più dettagliata del rapporto funzionale tra le restanti variabili di progetto e l’obiettivo di interesse. Utilizzando il set di dati successivamente estesi, l’approccio di Kriging è applicato per prevedere il risultato sperimentale alle posizioni non misurati. Il ciclo iterativo si arresta non appena il modello Kriging predice un’ottimale o altopiano con sufficiente precisione. I risultati sono verificati nel quarto passaggio (Figura 1), a partire da un’ulteriore analisi di sensibilità intorno l’optimum identificato. Se inizialmente, insensibile componenti si trovano ad per essere insensibili anche nella regione ottima, è ragionevole supporre che questo vale durante la procedura di ottimizzazione iterativa nel terzo passo. In seguito, si consiglia di verificare i risultati di ottimizzazione di applicazione dei metodi ortogonali, come un saggio di attività o SDS-Page.
Il carattere generico del protocollo presentato consente facile adattamento di attrezzature di laboratorio diversi, altri host espressione e proteine bersaglio di scelta, così come ulteriori bioprocessi variabili come la temperatura di valore o coltivazione di pH.Inoltre, altri obiettivi di ottimizzazione come tasso di produzione di proteina, resa specifica o qualità del prodotto possono essere scelti per adattarsi l’ambito di altri studi di ottimizzazione.
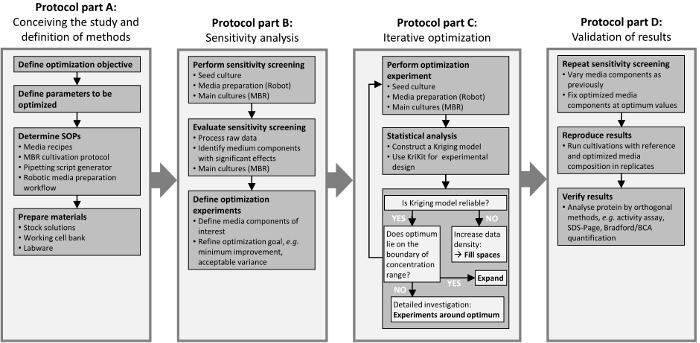
Figura 1 : Flusso di lavoro di studio di ottimizzazione. I quattro riquadri corrispondono alle sezioni del protocollo, “Concepire the studio e definizione di metodi” (sezione 1), “Sensitivity Analysis” (sezione 2), “Ottimizzazione iterativa” (sezione 3) e “Convalida” (sezione 4). Clicca qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Il carattere generico del protocollo presentato permette vari adattamenti, ad esempio, per studiare altri espressione microbica ospita9,47,48,49,50, 51, o per ottimizzare altre proprietà della proteina dell’obiettivo, come le obbligazioni di pattern o bisolfuro di glicosilazione. Il protocollo inoltre devono essere adattati per l’attrezzatura di laboratorio disponibili. L’integrazione di un sistema MBR permette di aumentare il throughput sperimentale, che consente un grande risparmio nel tempo. Tuttavia, quando sostituisce completamente strumentati e controllabile bioreattori di sistemi MBR, scalabilità dei risultati deve essere considerata8,37,52,53. L’utilizzo di metodologie DOE e modellazione matematica aiuta a massimizzare il contenuto informativo dei dati di misurazione rispetto l’ oggettiva studiati54 da un’efficiente pianificazione sperimentale e interpretazione di dati basati su modello15.
Modifiche al metodo
Accanto a multi-purpose ed espandibile robotica manipolazione dei liquidi sistemi come quello utilizzato in questo studio, va ricordato che ci sono parecchi più piccolo liquido sistemi disponibili in commercio che sono in grado di eseguire questa operazione e possono essere inseriti all’interno di movimentazione di banchi di lavoro a flusso laminare. Se nessun sistema di pipettaggio automatizzato è disponibile, composizioni di diversi media secondo il disegno DOE possono essere realizzate anche pipettando manuale utilizzando pipette mono e/o multi-canale. Poiché la preparazione manuale è più soggetto a errori e richiederà un lavoro altamente concentrato per un tempo abbastanza lungo, si consiglia di preparare un numero inferiore di composizioni di diversi media.
A seconda delle capacità del sistema MBR autonomo, varierà il corrispondente protocollo di coltivazione. Ad esempio, se non è disponibile alcuna misurazione online di formazione di biomassa, può essere sufficiente a misurare la concentrazione di biomassa dopo il completamento dell’esperimento di crescita. In combinazione con monitoraggio on-line il pH e l’ossigeno disciolto, che viene implementato in diversi sistemi MBR, la saturazione di crescita può essere determinata in modo sicuro. In linea di principio, gli esperimenti di crescita possono essere condotti in MTPs da solo collocato all’interno di agitazione incubatori, senza l’uso di un sistema MBR. In questo caso, le condizioni di corretta coltivazione necessario garantire: (1) ossigeno limitato coltivazioni possono essere evitato utilizzando MTPs con geometrie adatte, in combinazione con adeguata agitazione frequenze e scuotendo diametri, ad esempio, Piazza 96 o 24 profonda piatti ben operato a 1.000 giri/min a tiro di 3 mm o a 250 rpm a passi di 25 mm, rispettivamente. D’importanza, più basso i tassi di trasferimento di ossigeno massimo realizzabile, minore sarà la fonte principale di carbonio deve essere concentrata. Come accennato in precedenza, per questo studio, l’uso di 10 g/L glucosio era idoneo a prevenire la limitazione di ossigeno per le condizioni di coltivazione impiegato; (2) il campionamento delle culture MTP per quantificazione biomassa e prodotto dovrebbe essere ridotto al minimo. Ogni volta che il MTP è rimosso dall’incubatrice agitazione, trasferimento di ossigeno sarà immediatamente ripartizione che può provocare condizioni di coltivazione sfavorevole; (3) nel parere degli autori, l’uso dei lettori MTP come dispositivi di coltivazione non è raccomandato come questi dispositivi non sono stati sviluppati per questo scopo. Ad esempio, agitazione meccanica sono stati costruiti per la miscelazione occasionale di micropiastre dopo l’aggiunta del reagente e così, spesso mancanza di robustezza per lunghe percorrenze di agitazione continua durare per giorni. Inoltre, non può essere realizzato sufficienti input di alimentazione necessario per colture microbiche in questi lettori. L’integrazione delle letture di densità ottica in breve intervalli di tempo richiede arresto del movimento d’agitazione, conseguente a ripetuti periodi di prescrizione di ossigeno. Inoltre, evaporazione in tali sistemi su periodi lunghi coltivazione sarà falsare i risultati. Per maggiori dettagli sull’argomento sorprendentemente complesso sull’utilizzo MTPs per colture microbiche, si rimanda il lettore alla letteratura citata22,23,24,25,26 e bibliografia citata.
Ulteriori considerazioni
Per fasi di ottimizzazione iterativa di Speed-up, si consiglia di selezionare con attenzione il metodo analitico per la quantificazione del prodotto. Veloci e semplici metodi dovrebbero essere preferiti al costo di precisione e accuratezza, come la strategia di disegno sperimentale iterativo tollera sperimentale inesattezza. Tuttavia, i risultati finali devono essere verificati contro metodi di quantificazione del prodotto sufficientemente precise ed accurate che potrebbero essere più complicati. In generale, un’attenta valutazione e il processo decisionale circa le procedure di studio richiedono impegno all’inizio dello studio, ma pagare a lungo termine, dopo i metodi di routine sono stati stabiliti.
Si consiglia di definire un esperimento di riferimento che è rispetto a tutti gli esperimenti durante l’ottimizzazione. Cioè, le concentrazioni medie applicate componente così come uscita misurata è normalizzato tramite dividendo per i valori di riferimento. In questo modo, ciascuno applicato e il valore misurato può essere interpretato come la x-piega del valore di riferimento. Per tener conto delle variazioni tra le piastre, cinque esperimenti di riferimento vengono eseguiti su ogni piatto. Il valore medio dei risultati misurati viene utilizzato per la normalizzazione.
Non può generalmente essere garantito che il mezzo sviluppato è anche ottimo per altri ceppi. Tuttavia, il mezzo migliore probabilmente sarà anche appropriato per coltivare ceppi di espressione con piccole differenze genetiche, per esempio, quando produrre varianti di enzima con singolo amminoacido sostituzioni ottenuti da (studi di mutagenesi anche se per effetto di metabolismo cellulare e l’espressione eterologa prestazioni55,56, sono state descritte anche singole mutazioni puntiformi). In questo caso, il protocollo presentato può essere un primo passo, seguito da protocolli per espressione high-throughput screening57. Se il protocollo viene utilizzato per lo sviluppo medio con successiva scala-fino a coltivazioni di fed-batch, il supporto ottimizzato deve essere verificato per le corrispondenti condizioni di bioprocessi, come clone campagne a Microscala di screening identificato diversi top interpreti per diverse strategie d’alimentazione e coltivazione media52,58. Inoltre, l’introdotto KriKit36 generalmente possono contribuire alla ottimizzazione migliorata bioprocessi olistico.Solo di recente, le abilità di strumento sono estesi per supportare anche ottimizzazione multi-obiettivo40, che può essere importante per l’ottimizzazione di entrambi i processi a Monte e a valle59,60.
Disclosures
The authors have nothing to disclose.
Acknowledgements
L’attività scientifica del centro di scienza di bioeconomia era sostenuto finanziariamente dal Ministero dell’innovazione, scienza e della ricerca nell’ambito della BioSC NRW-Strategieprojekt (n. 313/323-400-002 13). Gli autori ringraziano il Ministero dell’innovazione, scienza e ricerca della Renania settentrionale-Vestfalia e Heinrich Heine University Düsseldorf per una borsa di studio a Lars Freier entro la biotecnologia industriale di Cluster CLIB-Graduate. Ulteriori finanziamenti è stato ricevuto dal programma di spazi di abilitazione “Helmholtz Innovation Labs” dell’Associazione Helmholtz tedesco per sostenere la “microbica Bioprocess Lab – A Helmholtz Innovation Lab”.
Materials
| BioLector | m2p-labs | G-BL-100 | |
| Flowerplate | m2p-labs | MTP-48-BOH | For cultivation in the BioLector device |
| Sealing foil | m2p-labs | F-GP-10 | Sterile sealing for Flowerplate |
| MATLAB | Mathworks | 2016b | |
| KriKit | Forschungszentrum Jülich | n/a | Freely available, MATLAB installation required |
| Janus pipetting robot | Perkin Elmer | n/a | Includes "WinPrep" software installation |
| 12-column deep well microplate | E&K Scientific | EK-2034 | Container for medium stock solutions |
| 96 well microplates, transparent, F-bottom | Greiner | 655101 | For Bradford protein assay |
| µclear 96 well microplates, black body, transparent F-bottom | Greiner | 655087 | For flourescence measurement in cell-free supernatants |
| Pipette Research plus multi-channel pipettes | Eppendorf | n/a | Facilitates manual liquid handling with microplates |
| TruPAG Precast Gels | Sigma | PCG2002 | For SDS-Page analysis of cell-free supernantants |
| Bradford Reagent | Sigma | B6916 | |
| C. glutamicum pCGPhoDBs-GFP | n/a | n/a | Carries pEKEx2 plasmid with fusion of GFP gene and PhoD signal peptide from B.subtilis as expression insert. Plasmid provides kanamycin resistance. Described and published by Meissner et al. Appl Microbiol Biotechnol 76 (3), 633–42 (2007) |
References
- Choi, J. -. M., Han, S. -. S., Kim, H. -. S. Industrial applications of enzyme biocatalysis: Current status and future aspects. Biotechnol Adv. 33 (7), 1443-1454 (2015).
- Li, S., Yang, X., Yang, S., Zhu, M., Wang, X. Technology Prospecting on Enzymes: Application, Marketing and Engineering. Comput Struct Biotechnol J. 2 (3), (2012).
- Adrio, J. L., Demain, A. L. Microbial enzymes: tools for biotechnological processes. Biomolecules. 4 (1), 117-139 (2014).
- Yim, S. S., An, S. J., Kang, M., Lee, J., Jeong, K. J. Isolation of fully synthetic promoters for high-level gene expression in Corynebacterium glutamicum. Biotechnol Bioeng. 110 (11), 2959-2969 (2013).
- Hemmerich, J., et al. Use of a Sec signal peptide library from Bacillus subtilis.for the optimization of cutinase secretion in Corynebacterium glutamicum. Microb Cell Fact. 15 (1), 208 (2016).
- Wendisch, V. F. Microbial production of amino acids and derived chemicals: synthetic biology approaches to strain development. Curr Opin Biotechnol. 30, 51-58 (2014).
- Lee, J. -. Y., Na, Y. -. A., Kim, E., Lee, H. -. S., Kim, P. The Actinobacterium Corynebacterium glutamicum, an Industrial Workhorse. J Microbiol Biotechnol. 26 (5), 807-822 (2016).
- Rohe, P., Venkanna, D., Kleine, B., Freudl, R., Oldiges, M. An automated workflow for enhancing microbial bioprocess optimization on a novel microbioreactor platform. Microb Cell Fact. 11 (1), 144 (2012).
- Liu, X., et al. Expression of recombinant protein using Corynebacterium glutamicum.: progress, challenges and applications. Crit Rev Biotechnol. 36 (4), 652-664 (2016).
- Freudl, R., Burkovski, A. Corynebacterium glutamicum as a Platform Organism for the Secretory Production of Heterologous Proteins. Corynebacterium glutamicum: From systems biology to biotechnological applications. , 161-177 (2015).
- Yim, S. S., et al. Development of a new platform for secretory production of recombinant proteins in Corynebacterium glutamicum. Biotechnol Bioeng. 113 (1), 163-172 (2015).
- Limberg, M. H., et al. Metabolic profile of 1,5-diaminopentane producing Corynebacterium glutamicum under scale-down conditions: Blueprint for robustness to bioreactor inhomogeneities. Biotechnol Bioeng. 114 (3), 560-575 (2016).
- Käβ, F. Assessment of robustness against dissolved oxygen/substrate oscillations for C. glutamicum DM1933 in two-compartment bioreactor. Bioprocess Biosyst Eng. 37 (6), 1151-1162 (2014).
- Teramoto, H., Watanabe, K., Suzuki, N., Inui, M., Yukawa, H. High yield secretion of heterologous proteins in Corynebacterium glutamicum.using its own Tat-type signal sequence. Appl Microbiol Biotechnol. 91 (3), 677-687 (2011).
- Freier, L., Hemmerich, J., Schöler, K., Wiechert, W., Oldiges, M., von Lieres, E. Framework for Kriging-based iterative experimental analysis and design: Optimization of secretory protein production in Corynebacterium glutamicum. Eng Life Sci. 16 (6), 538-549 (2016).
- Huber, R., Roth, S., Rahmen, N., Büchs, J. Utilizing high-throughput experimentation to enhance specific productivity of an E. coli. T7 expression system by phosphate limitation. BMC Biotechnol. 11 (1), 22 (2011).
- Kottmeier, K., Müller, C., Huber, R., Büchs, J. Increased product formation induced by a directed secondary substrate limitation in a batch Hansenula polymorpha culture. Appl Microbiol Biotechnol. 86 (1), 93-101 (2010).
- Kennedy, M. J., Krouse, D. Strategies for improving fermentation medium performance: A review. J Ind Microbiol Biotechnol. 23 (6), 456-475 (1999).
- Kleman, G. L., Strohl, W. R. Developments in high cell density and high productivity microbial fermentation. Curr Opin Biotechnol. 5 (2), 180-186 (1994).
- Jones, R., Gadd, G. Ionic nutrition of yeast: Physiological mechanisms involved and implications for biotechnology. Enzy Microb Technol. 12 (6), 402-418 (1990).
- Zhang, J., Greasham, R. Chemically defined media for commercial fermentations. Appl Microbiol Biotechnol. 51 (4), 407-421 (1999).
- Kensy, F., et al. Oxygen transfer phenomena in 48-well microtiter plates: Determination by optical monitoring of sulfite oxidation and verification by real-time measurement during microbial growth. Biotechnol Bioeng. 89 (6), 698-708 (2005).
- Hermann, R., Lehmann, M., Büchs, J. Characterization of gas-liquid mass transfer phenomena in microtiter plates. Biotechnol Bioeng. 81 (2), 178-186 (2002).
- Duetz, W. A. Microtiter plates as mini-bioreactors: Miniaturization of fermentation methods. Trends Microbiol. 15 (10), 469-475 (2007).
- Funke, M., Diederichs, S., Kensy, F., Müller, C., Büchs, J. The baffled microtiter plate: Increased oxygen transfer and improved online monitoring in small scale fermentations. Biotechnol Bioeng. 103 (6), 1118-1128 (2009).
- Lattermann, C., Funke, M., Hansen, S., Diederichs, S., Büchs, J. Cross-section perimeter is a suitable parameter to describe the effects of different baffle geometries in shaken microtiter plates. J Biol Eng. 8, 18 (2014).
- Samorski, M., Müller-Newen, G., Büchs, J. Quasi-continuous combined scattered light and fluorescence measurements: A novel measurement technique for shaken microtiter plates. Biotechnol Bioeng. 92 (1), 61-68 (2005).
- Kensy, F., Zang, E., Faulhammer, C., Tan, R. -. K., Büchs, J. Validation of a high-throughput fermentation system based on online monitoring of biomass and fluorescence in continuously shaken microtiter plates. Microb Cell Fact. 8 (1), 31 (2009).
- Puskeiler, R., Kaufmann, K., Weuster-Botz, D. Development, parallelization, and automation of a gas-inducing milliliter-scale bioreactor for high-throughput bioprocess design (HTBD). Biotechnol Bioeng. 89 (5), 512-523 (2005).
- Bareither, R., Pollard, D. A review of advanced small-scale parallel bioreactor technology for accelerated process development: Current state and future need. Biotechnol Prog. 27 (1), 2-14 (2011).
- Hortsch, R., Stratmann, A., Weuster-Botz, D. New milliliter-scale stirred tank bioreactors for the cultivation of mycelium forming microorganisms. Biotechnol Bioeng. 106 (3), 443-451 (2010).
- Isett, K., George, H., Herber, W., Amanullah, A. Twenty-four-well plate miniature bioreactor high-throughput system: Assessment for microbial cultivations. Biotechnol Bioeng. 98 (5), 1017-1028 (2007).
- Motta Dos Santos, L. F., Coutte, F., Ravallec, R., Dhulster, P., Tournier-Couturier, L., Jacques, P. An improvement of surfactin production by B. subtilis BBG131 using design of experiments in microbioreactors and continuous process in bubbleless membrane bioreactor. Bioresour Technol. 218, 944-952 (2016).
- Islam, R. S., Tisi, D., Levy, M. S., Lye, G. J. Framework for the Rapid Optimization of Soluble Protein Expression in Escherichia coli Combining Microscale Experiments and Statistical Experimental Design. Biotechnol Prog. 23 (4), 785-793 (2007).
- Huber, R., et al. Robo-Lector: A novel platform for automated high-throughput cultivations in microtiter plates with high information content. Microb Cell Fact. 8 (1), 42 (2009).
- . Kriging toolKit (KriKit) Available from: https://github.com/modsim/KriKit (2017)
- Kensy, F., Engelbrecht, C., Büchs, J. Scale-up from microtiter plate to laboratory fermenter: evaluation by online monitoring techniques of growth and protein expression in Escherichia coli.and Hansenula polymorpha fermentations. Microb Cell Fact. 8 (1), 68 (2009).
- Lu, C., Bentley, W. E., Rao, G. A high-throughput approach to promoter study using green fluorescent protein. Biotechnol Prog. 20 (6), 1634-1640 (2004).
- Zanzotto, A., Boccazzi, P., Gorret, N., van Dyk, T. K., Sinskey, A. J., Jensen, K. F. In situ measurement of bioluminescence and fluorescence in an integrated microbioreactor. Biotechnol Bioeng. 93 (1), 40-47 (2006).
- Freier, L., von Lieres, E. Multi-Objective Global Optimization (MOGO): Algorithm and Case Study in Gradient Elution Chromatography. Biotechnol J. , (2016).
- Keilhauer, C., Eggeling, L., Sahm, H. Isoleucine synthesis in Corynebacterium glutamicum.: molecular analysis of the ilvB-ilvN-ilvC operon. J Bacteriol. 175 (17), 5595-5603 (1993).
- Weuster-Botz, D., Kelle, R., Frantzen, M., Wandrey, C. Substrate Controlled Fed-Batch Production of L-Lysine with Corynebacterium glutamicum. Biotechnol Prog. 13 (4), 387-393 (1997).
- Meissner, D., Vollstedt, A., van Dijl, J. M., Freudl, R. Comparative analysis of twin-arginine (Tat)-dependent protein secretion of a heterologous model protein (GFP) in three different Gram-positive bacteria. Appl Microbiol Biotechnol. 76 (3), 633-642 (2007).
- Morschett, H., Wiechert, W., Oldiges, M. Automation of a Nile red staining assay enables high throughput quantification of microalgal lipid production. Microb Cell Fact. 15, 34 (2016).
- Fisher, R. A. . The design of experiments. , (1935).
- Morschett, H., Freier, L., Rohde, J., Wiechert, W., von Lieres, E., Oldiges, M. A framework for accelerated phototrophic bioprocess development: Integration of parallelized microscale cultivation, laboratory automation and Kriging-assisted experimental design. Biotechnol Biofuels. 10, 26 (2017).
- Le Loir, Y., et al. Protein secretion in Lactococcus lactis: An efficient way to increase the overall heterologous protein production. Microb Cell Fact. 4 (1), 2 (2005).
- Anné, J., Vrancken, K., van Mellaert, L., van Impe, J., Bernaerts, K. Protein secretion biotechnology in Gram-positive bacteria with special emphasis on Streptomyces lividans. Biochim Biophys Acta. 1843 (8), 1750-1761 (2014).
- Gupta, S. K., Shukla, P. Advanced technologies for improved expression of recombinant proteins in bacteria: perspectives and applications. Crit Rev Biotechnol. 36 (6), 1089-1098 (2016).
- Fu, L. L., Xu, Z. R., Li, W. F., Shuai, J. B., Lu, P., Hu, C. X. Protein secretion pathways in Bacillus subtilis: implication for optimization of heterologous protein secretion. Biotechnol Adv. 25 (1), 1-12 (2007).
- Yoon, S., Kim, S., Kim, J. Secretory Production of Recombinant Proteins in Escherichia coli. Rec Pat Biotechnol. 4 (1), 23-29 (2010).
- Hemmerich, J., et al. Comprehensive clone screening and evaluation of fed-batch strategies in a microbioreactor and lab scale stirred tank bioreactor system: application on Pichia pastoris producing Rhizopus oryzae lipase. Microb Cell Fact. 13 (1), 36 (2014).
- Glazyrina, J., Krause, M., Junne, S., Glauche, F., Strom, D., Neubauer, P. Glucose-limited high cell density cultivations from small to pilot plant scale using an enzyme-controlled glucose delivery system. New Biotechnol. 29 (2), 235-242 (2012).
- Gernaey, K. V., et al. Monitoring and control of microbioreactors: An expert opinion on development needs. Biotechnol J. 7 (10), 1308-1314 (2012).
- Rahmen, N., Fulton, A., Ihling, N., Magni, M., Jaeger, K. -. E., Büchs, J. Exchange of single amino acids at different positions of a recombinant protein affects metabolic burden in Escherichia coli. Microb Cell Fact. 14, 10 (2015).
- Rahmen, N., et al. A particular silent codon exchange in a recombinant gene greatly influences host cell metabolic activity. Microb Cell Fact. 14, 156 (2015).
- Saez, N. J., Nozach, H., Blemont, M., Vincentelli, R. High throughput quantitative expression screening and purification applied to recombinant disulfide-rich venom proteins produced in E. coli. J Vis Exp. (89), e51464 (2014).
- Scheidle, M., et al. High-throughput screening of Hansenula polymorpha clones in the batch compared with the controlled-release fed-batch mode on a small scale. FEMS Yeast Res. 10 (1), 83-92 (2010).
- Baumann, P., Bluthardt, N., Renner, S., Burghardt, H., Osberghaus, A., Hubbuch, J. Integrated development of up- and downstream processes supported by the Cherry-Tag™ for real-time tracking of stability and solubility of proteins. J Biotechnol. 200, 27-37 (2015).
- Baumann, P., Hahn, T., Hubbuch, J. High-throughput micro-scale cultivations and chromatography modeling: Powerful tools for integrated process development. Biotechnol Bioeng. 112 (10), 2123-2133 (2015).

