Uma estratégia de enriquecimento de ponta de spin para análise simultânea de N-glicopeptídeos e Fosfopeptídeos de Tecidos Pancreáticos Humanos
Summary
Modificações pós-translacionais (PTMs) alteram estruturas e funções proteicas. Métodos para o enriquecimento simultâneo de vários tipos de PTM podem maximizar a cobertura nas análises. Apresentamos um protocolo utilizando cromatografia de afinidade metálica ti(IV) automobilizada duplamente, seguida de espectrometria de massa para o enriquecimento simultâneo e análise da proteína N-glicosilação e fosforilação em tecidos pancreáticos.
Abstract
A espectrometria em massa pode fornecer uma cobertura profunda de modificações pós-translacionais (PTMs), embora o enriquecimento dessas modificações de matrizes biológicas complexas seja frequentemente necessário devido à sua baixa estequiometria em comparação com analitos não modificados. A maioria dos fluxos de trabalho de enriquecimento de PTMs em peptídeos em fluxos de trabalho de proteômica inferior, onde as proteínas são digeridas enzimáticamente antes da análise dos peptídeos resultantes, apenas enriquecem um tipo de modificação. É todo o complemento dos PTMs, porém, que leva a funções biológicas, e o enriquecimento de um único tipo de PTM pode perder tal crosstalk de PTMs. O crosstalk PTM tem sido observado entre a glicosilação proteica e a fosforilação, os dois PTMs mais comuns em proteínas humanas e também os dois PTMs mais estudados usando fluxos de trabalho de espectrometria de massa. Usando a estratégia simultânea de enriquecimento aqui descrita, ambos os PTMs são enriquecidos a partir de tecido pancreático humano pós-morte, uma complexa matriz biológica. A cromatografia de afinidade metálica duplamente funcional Ti(IV) é usada para separar várias formas de glicosilação e fosforilação simultaneamente em múltiplas frações em um método conveniente baseado em ponta de giro, permitindo análises a jusante de interações potenciais de crosstalk ptm. Esse fluxo de trabalho de enriquecimento para glico e fosfopeptídeos pode ser aplicado a vários tipos de amostra para alcançar perfis profundos de múltiplos PTMs e identificar moléculas-alvo potenciais para estudos futuros.
Introduction
As modificações pós-translacionais de proteínas (PTMs) desempenham um papel importante na modulação das estruturas proteicas e, consequentemente, suas funções e processos biológicos a jusante. A diversidade do proteome humano aumenta exponencialmente devido à variabilidade combinatória proporcionada por vários PTMs. Diferentes variantes de proteínas de suas sequências canônicas como previsto pelo genoma são conhecidas como proteoformas, e muitas proteoformas surgem dos PTMs1. Estudar a diversidade proteoforme em saúde e doença tornou-se uma área de pesquisa de grande interesse nos últimos anos 2,3.
O estudo de proteoformes e, mais especificamente, PTMs com grande profundidade tornou-se mais fácil através do desenvolvimento de métodos de proteômica baseados em espectrometria de massa (MS). Utilizando ES, os analitos são ionizados, fragmentados e identificados com base no m/z dos fragmentos. Métodos de enriquecimento são muitas vezes necessários devido à baixa abundância relativa de PTMs em comparação com formas não modificadas de proteínas. Embora a análise de proteínas intactas e seus PTMs, chamados de análises de cima para baixo, tenham se tornado mais rotineiras, a digestão enzimática das proteínas e a análise de seus peptídeos componentes em análises de baixo para cima ainda é o caminho mais utilizado para a análise de PTM. Os dois PTMs mais estudados, e os dois PTMs mais comuns in vivo, são glicosilação e fosforilação4. Esses dois PTMs desempenham papéis importantes na sinalização e reconhecimento celular e, portanto, são modificações importantes para caracterizar na pesquisa de doenças.
As propriedades químicas de vários PTMs muitas vezes fornecem rotas para o enriquecimento desses PTMs nos níveis de proteína e peptídeo antes da análise. A glicosylação é um PTM hidrofílico devido à abundância de grupos hidroxis em cada monossacarídeo. Esta propriedade pode ser usada para enriquecer glicopeptídeos na cromatografia de interação hidrofílica (HILIC), que pode separar glicopeptídeos mais hidrofílicos dos peptídeos hidrofóbicos não modificados5. A fosforilação adiciona a moiety fosfato, que é negativamente carregada, exceto no pH ácido. Devido a esta carga, várias cálas metálicas, incluindo titânio, podem ser usadas para atrair e ligar fosforpeptídeos enquanto espécies não fosfoiladas são lavadas. Este é o princípio da cromatografia de afinidade metálica imobilizada (IMAC). Outras discussões sobre essas e outras estratégias de enriquecimento para glicosilação e fosforilação podem ser encontradas em revisões recentes 6,7.
Quantidades comparativamente grandes de material de peptídeo inicial (0,5 mg ou mais) são frequentemente necessárias para protocolos de enriquecimento devido à baixa estequiometria de PTMs em peptídeos. Em cenários onde essa quantidade de amostra pode não ser facilmente obtida, como biópsia do núcleo tumoral ou análises de fluidos cefalorraquidianos, é benéfico usar fluxos de trabalho fáceis que resultam em informações biomoleculares máximas. Estratégias recentes desenvolvidas pelo nosso laboratório e outras têm destacado a análise simultânea e paralela da glicosilação e fosforilação utilizando o mesmo fluxo de trabalho de enriquecimento ptm 8,9,10,11,12. Embora as propriedades químicas desses dois PTMs possam diferir, esses PTMs podem ser analisados em múltiplas etapas devido às técnicas de separação inovadoras e materiais utilizados. Por exemplo, a cromatografia de interação eletrostática-hidrofílica (ERLIC) sobrepõe separações baseadas em interações hidrofílicas entre analitos e a fase móvel com interações de carga entre analitos e o material de fase estacionária 13,14,15,16. No pH ácido, a atração de peptídeos fosforilalatados para a fase estacionária pode melhorar sua retenção e separação de peptídeos não modificados. O material constituído por Ti(IV) imobilizado em microesferas hidrofílicas pode ser usado para elução baseada em HILIC e IMAC para separar fosforpeptídeos e glicopeptídeos neutros, ácidos e sistoluados-6-fosforilados17,18. Esta estratégia é conhecida como Ti(IV)-IMAC dual-funcional. O uso dessas estratégias para enriquecer vários PTMs em um único fluxo de trabalho pode tornar as análises de interações potenciais de crosstalk de PTM mais acessíveis. Além disso, o valor total da amostra e os requisitos de tempo são inferiores aos métodos convencionais de enriquecimento quando realizados em paralelo (ou seja, HILIC e IMAC em alíquotas de amostra separadas).
Para demonstrar a estratégia ti(IV)-IMAC dual-funcional para análise simultânea de glicosilação e fosforilação proteica, aplicamos-na para analisar tecidos pancreáticos humanos pós-morte. O pâncreas produz enzimas digestivas e hormônios regulatórios, incluindo insulina e glucagon. A função pancreática é prejudicada em doença pancreática. No diabetes, a regulação do açúcar no sangue é afetada, levando a níveis mais elevados de glicose no sangue. Na pancreatite, a inflamação resulta da auto-digestão do órgão3. Alterações nos perfis de PTM, incluindo glicosilação e fosforilação, podem resultar, como muitas vezes, em outras doenças.
Aqui, descrevemos um protocolo para um método de enriquecimento simultâneo baseado em spin-tip, baseado em uma estratégia ti(IV)-IMAC dual-funcional, para N-glicopeptídeos e fosfopeptídeos derivados de proteínas extraídas do tecido pancreático. O protocolo inclui extração e digestão de proteínas, enriquecimento, coleta de dados em MS e processamento de dados, como pode ser visto na Figura 1. Os dados representativos deste estudo estão disponíveis via Consórcio ProteomeXchange com o identificador PXD033065.
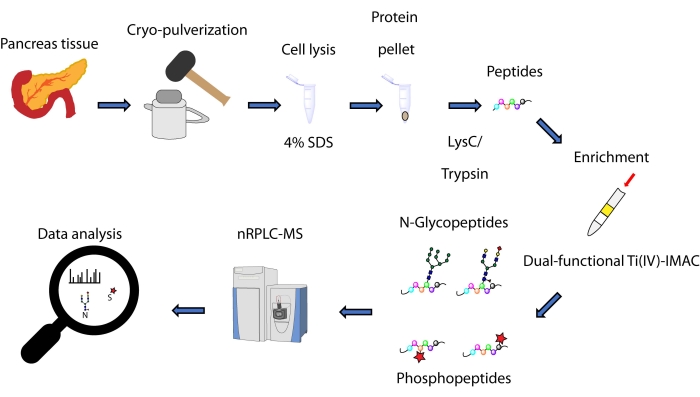
Figura 1: Fluxo de trabalho para análise simultânea de N-glicopeptídeos e fosfopeptídeos de tecidos pancreáticos humanos. Os tecidos são primeiro crio-pulverizados em um pó fino antes da extração de proteínas usando o sulfato de dodecyl de sódio detergente (SDS). As proteínas são então submetidas à digestão enzimática. Os peptídeos resultantes são aliquosantes antes do enriquecimento usando Ti(IV)-IMAC dual-funcional. Os dados brutos são coletados usando espectrometria de massa líquida de fase invertida de nanoescala (nRPLC-MS) e são analisados usando software de pesquisa de banco de dados. Clique aqui para ver uma versão maior desta figura.
Este protocolo visa tornar as análises de PTM mais acessíveis e permitir uma análise mais difundida de vários PTMs no mesmo fluxo de trabalho. Este protocolo pode ser aplicado a outras matrizes biológicas complexas, incluindo células e biofluidos.
Protocol
Representative Results
Discussion
A estratégia Ti(IV)-IMAC dual-funcional é útil para a análise simultânea de N-glicopeptídeos e fosfopeptídeos da mesma amostra em um único fluxo de trabalho de preparação de amostra. Métodos baseados em ERLIC também têm sido mostrados para realizar o enriquecimento simultâneo de PTMs. Ambas as estratégias têm sido utilizadas anteriormente para cobertura profunda em análises de PTM14,18. Ao adaptar o método Ti duplo para diminuir o tempo de incub…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Esta pesquisa foi apoiada em parte pelo financiamento de subvenções do NIH (R01DK071801, RF1AG052324, P01CA250972 e R21AG065728), e da Fundação de Pesquisa de Diabetes Juvenil (1-PNF-2016-250-S-B e SRA-2016-168-S-B). Os dados aqui apresentados também foram obtidos em parte através do apoio de um prêmio NIH/NCATS UL1TR0002373 através do Instituto de Pesquisa Clínica e Translacional da Universidade de Wisconsin. Os instrumentos Orbitrap foram adquiridos através do apoio de uma bolsa de instrumentos compartilhados NIH (NIH-NCRR S10RR029531) e do Escritório do Vice-Chanceler de Pesquisa e Pós-Graduação da Universidade de Wisconsin-Madison. Também gostaríamos de reconhecer o generoso apoio da Organização de Doação de Órgãos e Tecidos da Universidade de Wisconsin, que forneceu pâncreas humano para pesquisa e a ajuda de Dan Tremmel, Dr. Sara D. Sackett e Prof. Jon Odorico para fornecer as amostras ao nosso laboratório. Nossa equipe de pesquisa gostaria de agradecer especial às famílias que doaram tecidos para este estudo. L.L. reconhece a bolsa nih S10OD025084, uma bolsa piloto de câncer de pâncreas do Centro de Câncer carbone da Universidade de Wisconsin (233-AAI9632), bem como um Professor de Conquista Distinto de Vilas e o Professor de Cadeira Distinto Charles Melbourne Johnson com financiamento fornecido pela Wisconsin Alumni Research Foundation e Pela University of Wisconsin-Madison School of Pharmacy.
Materials
| Acetic Acid, Glacial (Certified ACS) | Fisher Scientific | A38S-500 | |
| Acetone (Certified ACS) | Fisher Scientific | A18-1 | |
| Acetonitrile, Optima LC/MS Grade | Fisher Scientific | A955-4 | |
| Ammonium Acetate (Crystalline/Certified ACS) | Fisher Scientific | A637-500 | |
| Ammonium Hydroxide (Certified ACS Plus) | Fisher Scientific | A669-212 | |
| Byonic software | Protein Metrics | n/a | Commercial software used for glycoproteomic analysis (https://proteinmetrics.com/byos/) |
| C18 BEH material | Waters | 186002353 | Material removed from column and used to pack nano capillaries (pulledto integrate tip used directly in line with instrument inlet) |
| CAE-Ti-IMAC, 100% | J&K Scientific | 2749380-1G | Material used for dual-functional Ti(IV)-IMAC; can also be used for conventional IMAC/conventional phosphopeptide enrichment |
| Cellcrusher kit | Cellcrusher | n/a | Used for grinding tissue samples into powder before extraction |
| Eppendorf 5424R Microcentrifuge | Fisher Scientific | 05-401-205 | For temperature-controlled centrifugation |
| cOmplete protease inhibitor cocktail tablets | Sigma | 11697498001 | |
| DTT, Molecular Grade (DL-Dithiothreitol) | Promega | V3151 | Protein reducing agent |
| Ethanol, 200 proof (100%), USP | Fisher | 22-032-601 | |
| Fisherbrand Analog Vortex Mixer | Fisher Scientific | 02-215-414 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (1.5 mL) | Fisher Scientific | 02-681-320 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (2 mL) | Fisher Scientific | 02-681-321 | |
| Fisherbrand Model 120 Sonic Dismembrator | Fisher Scientific | FB120110 | For sample lysis using ultrasonication |
| Formic Acid, 99.0+%, Optima LC/MS Grade | Fisher Scientific | A117-50 | |
| Fused silica capillary (75 μm inner diameter, 360 μm outer diameter) | Polymicro Technologies LLC | 100 m TSP075375 | For in-house pulled and packed columns with integrated emitter |
| Hydrofluoric acid (48 wt. % in H2O) | Sigma-Aldrich | 339261-100ML | Used for opening emitter of pulled capillary column |
| Iodoacetamide, BioUltra | Sigma | I1149-5G | Protein reducing reagent |
| MaxQuant software | n/a | n/a | Free software used for phosphoproteomic analysis (https://www.maxquant.org/) |
| Multi-therm Shaker with heating and cooling | Benchmark Scientific | H5000-HC | Heating block |
| Oasis HLB 1 cc Vac Cartridge, 10 mg Sorbent per Cartridge, 30 µm, 100/pk | Waters | 186000383 | Larger-scale cartridge desalting for tryptic digests (loading capacity approximately up to 1 mg each) |
| OMIX C18 pipette tips, 100 µL tip, 10 – 100 μL elution volume, 1 x 96 tips | Agilent | A57003100 | Smaller-scale packed pipette tip for desalting for enrichment elutions |
| P-2000 Micropipette Puller | Sutter Instrument Co. | P-2000/F | For pulling nano-capillary columns for LC-MS |
| PhosSTOP phosphatase inhibitor tablets | Sigma | 4906845001 | |
| Pierce BCA Protein Assay Kit | Thermo Fisher Scientific | 23225 | |
| Pierce Quantitative Colorimetric Peptide Assay | Thermo Fisher Scientific | 23275 | |
| PolySAX LP (12 μm, pore size 300 Å) | PolyLC | BMSX1203 | Material for strong anion-exchange chromatography used for ERLIC/conventional glycopeptide enrichment |
| Potassium Phosphate Monobasic (Crystalline/Certified ACS) | Fisher Scientific | P285-500 | |
| Pressure injection cell with integrated magnetic stirplate | Next Advance | PC77-MAG | For packing nano-capillary columns with stationary phase up to 2500 psi limit |
| Proteome Discoverer software | Thermo Fisher Scientific | n/a | Commercial software for proteomics anaysis (with integrated database searching software nodes) and data visualization (https://www.thermofisher.com/us/en/home/industrial/mass-spectrometry/liquid-chromatography-mass-spectrometry-lc-ms/lc-ms-software/multi-omics-data-analysis/proteome-discoverer-software.html) |
| SpeedVac SC110 Vacuum Concentrator Model SC110-120 | Savant | n/a | Centrifugal vacuum concentrator for drying samples (under heat) |
| SDS Solution, 10% Sodium Dodecyl Sulfate Solution, Molecular Biology/Electrophoresis | Fisher Scientific | BP2436200 | |
| Sequencing Grade Modified Trypsin | Promega | V5111 | |
| Sodium Chloride (Crystalline/Certified ACS) | Fisher Scientific | S271-500 | |
| TopTip, Empty, 10-200 µL, Pack of 96 | Glygen Corporation | TT2EMT.96 | Empty pipette tip with micron-sized hole used that can be used to pack chromatographic materials for enrichments, bundled with tube adapters |
| Triethylammonium bicarbonate buffer (TEAB, 1 M, pH 8.5 (volatile)) | Sigma | 90360-100ML | |
| Trifluoroacetic acid, Reagent Grade, 99% | Fisher Scientific | 60-017-61 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | Fisher Scientific | BP152-500 | |
| Trypsin/Lys-C Mix, Mass Spec Grade | Promega | V5071 | |
| Urea (Certified ACS) | Fisher Scientific | U15-500 | |
| Water, Optima LC/MS Grade | Fisher Scientific | W64 |
References
- Smith, L. M., Kelleher, N. L. Proteoform: a single term describing protein complexity. Nature Methods. 10 (3), 186-187 (2013).
- Pan, S., Brentnall, T. A., Chen, R. Glycoproteins and glycoproteomics in pancreatic cancer. World Journal of Gastroenterology. 22 (42), 9288-9299 (2016).
- Tabang, D. N., Ford, M., Li, L. Recent advances in mass spectrometry-based glycomic and glycoproteomic studies of pancreatic diseases. Frontiers in Chemistry. 9, 707387 (2021).
- Khoury, G. A., Baliban, R. C., Floudas, C. A. Proteome-wide post-translational modification statistics: frequency analysis and curation of the swiss-prot database. Scientific Reports. 1, 90 (2011).
- Alpert, A. J. Hydrophilic-interaction chromatography for the separation of peptides, nucleic acids and other polar compounds. Journal of Chromatography A. 499, 177-196 (1990).
- Riley, N. M., Bertozzi, C. R., Pitteri, S. J. A pragmatic guide to enrichment strategies for mass spectrometry-based glycoproteomics. Molecular & Cellular Proteomics. 20, 100029 (2020).
- Low, T. Y., et al. Widening the bottleneck of phosphoproteomics: Evolving strategies for phosphopeptide enrichment. Mass Spectrometry Reviews. 40 (4), 309-333 (2021).
- Cho, K. C., Chen, L., Hu, Y., Schnaubelt, M., Zhang, H. Developing workflow for simultaneous analyses of phosphopeptides and glycopeptides. ACS Chemical Biology. 14 (1), 58-66 (2019).
- Zhou, Y., et al. An integrated workflow for global, glyco-, and phospho-proteomic analysis of tumor tissues. Analytical Chemistry. 92 (2), 1842-1849 (2020).
- Tang, R., et al. Facile preparation of bifunctional adsorbents for efficiently enriching N-glycopeptides and phosphopeptides. Analytica Chimica Acta. 1144, 111-120 (2021).
- Wang, Z., Wang, J., Sun, N., Deng, C. A promising nanoprobe based on hydrophilic interaction liquid chromatography and immobilized metal affinity chromatography for capture of glycopeptides and phosphopeptides. Analytica Chimica Acta. 1067, 1-10 (2019).
- Glover, M. S., et al. Characterization of intact sialylated glycopeptides and phosphorylated glycopeptides from IMAC enriched samples by EThcD fragmentation: Toward combining phosphoproteomics and glycoproteomics. International Journal of Mass Spectrometry. 427, 35-42 (2018).
- Alpert, A. J. Electrostatic repulsion hydrophilic interaction chromatography for isocratic separation of charged solutes and selective isolation of phosphopeptides. Analytical Chemistry. 80 (1), 62-76 (2008).
- Cui, Y., et al. Counterion optimization dramatically improves selectivity for phosphopeptides and glycopeptides in electrostatic repulsion-hydrophilic interaction chromatography. Analytical Chemistry. 93 (22), 7908-7916 (2021).
- Cui, Y., et al. Finding the sweet spot in ERLIC mobile phase for simultaneous enrichment of N-Glyco and phosphopeptides. Journal of the American Society for Mass Spectrometry. 30 (12), 2491-2501 (2019).
- Tabang, D. N., et al. Analysis of pancreatic extracellular matrix protein post-translational modifications via electrostatic repulsion-hydrophilic interaction chromatography coupled with mass spectrometry. Molecular Omics. 17 (5), 652-664 (2021).
- Huang, J., et al. Dual-functional Titanium(IV) immobilized metal affinity chromatography approach for enabling large-scale profiling of protein Mannose-6-Phosphate glycosylation and revealing its predominant substrates. Analytical Chemistry. 91 (18), 11589-11597 (2019).
- Huang, J., et al. Dual-functional Ti(IV)-IMAC material enables simultaneous enrichment and separation of diverse glycopeptides and phosphopeptides. Analytical Chemistry. 93 (24), 8568-8576 (2021).
- Jami-Alahmadi, Y., Pandey, V., Mayank, A. K., Wohlschlegel, J. A. A robust method for packing high resolution C18 RP-nano-HPLC columns. Journal of Visualized Experiments: JoVE. (171), e62380 (2021).
- Bern, M., Kil, Y. J., Becker, C. Byonic: advanced peptide and protein identification software. Current Protocols in Bioinformatics. , (2012).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).
- Zhou, Y., et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets. Nature Communications. 10 (1), 1523 (2019).
- Zacharias, L. G., et al. HILIC and ERLIC enrichment of glycopeptides derived from breast and brain cancer cells. Journal of Proteome Research. 15 (10), 3624-3634 (2016).
- Yang, W., et al. Comparison of enrichment methods for intact N- and O-linked glycopeptides using strong anion exchange and hydrophilic interaction liquid chromatography. Analytical Chemistry. 89 (21), 11193-11197 (2017).
- Toghi Eshghi, S., Shah, P., Yang, W., Li, X., Zhang, H. GPQuest: A spectral library matching algorithm for site-specific assignment of tandem mass spectra to intact N-glycopeptides. Analytical Chemistry. 87 (10), 5181-5188 (2015).
- Liu, M. -. Q., et al. pGlyco 2.0 enables precision N-glycoproteomics with comprehensive quality control and one-step mass spectrometry for intact glycopeptide identification. Nature Communications. 8 (1), 438 (2017).
- Lu, L., Riley, N. M., Shortreed, M. R., Bertozzi, C. R., Smith, L. M. O-Pair Search with MetaMorpheus for O-glycopeptide characterization. Nature Methods. 17 (11), 1133-1138 (2020).
- Caval, T., Heck, A. J. R., Reiding, K. R. Meta-heterogeneity: Evaluating and describing the diversity in glycosylation between sites on the same glycoprotein. Molecular & Cellular Proteomics. 20, 100010 (2021).
- Lee, J. S., Smith, E., Shilatifard, A. The language of histone crosstalk. Cell. 142 (5), 682-685 (2010).
- Leutert, M., Entwisle, S. W., Villén, J. Decoding post-translational modification crosstalk with proteomics. Molecular & Cellular Proteomics. 20, 100129 (2021).
- Hart, G. W., Slawson, C., Ramirez-Correa, G., Lagerlof, O. Cross talk between O-GlcNAcylation and phosphorylation: Roles in signaling, transcription, and chronic disease. Annual Review of Biochemistry. 80 (1), 825-858 (2011).

