Una estrategia de enriquecimiento de spin-tip para el análisis simultáneo de N-glicopéptidos y fosfopéptidos de tejidos pancreáticos humanos
Summary
Las modificaciones post-traduccionales (PTM) cambian las estructuras y funciones de las proteínas. Los métodos para el enriquecimiento simultáneo de múltiples tipos de PTM pueden maximizar la cobertura en los análisis. Presentamos un protocolo que utiliza cromatografía de afinidad metálica inmovilizada ti(IV) de doble funcionalidad seguida de espectrometría de masas para el enriquecimiento y análisis simultáneo de la proteína N-glicosilación y fosforilación en tejidos pancreáticos.
Abstract
La espectrometría de masas puede proporcionar una cobertura profunda de las modificaciones post-traduccionales (PTM), aunque el enriquecimiento de estas modificaciones a partir de matrices biológicas complejas es a menudo necesario debido a su baja estequiometría en comparación con los analitos no modificados. La mayoría de los flujos de trabajo de enriquecimiento de PTM en péptidos en flujos de trabajo de proteómica de abajo hacia arriba, donde las proteínas se digieren enzimáticamente antes de que se analicen los péptidos resultantes, solo enriquecen un tipo de modificación. Sin embargo, es todo el complemento de PTM lo que conduce a las funciones biológicas, y el enriquecimiento de un solo tipo de PTM puede pasar por alto dicha diafonía de PTM. Se ha observado diafonía de PTM entre la glicosilación de proteínas y la fosforilación, los dos PTM más comunes en proteínas humanas y también los dos PTM más estudiados utilizando flujos de trabajo de espectrometría de masas. Utilizando la estrategia de enriquecimiento simultáneo descrita en este documento, ambos PTM se enriquecen a partir de tejido pancreático humano post mortem, una matriz biológica compleja. La cromatografía de afinidad metálica inmovilizada ti(IV) de doble funcionalidad se utiliza para separar varias formas de glicosilación y fosforilación simultáneamente en múltiples fracciones en un método conveniente basado en punta de espín, lo que permite análisis aguas abajo de posibles interacciones de diafonía PTM. Este flujo de trabajo de enriquecimiento para glicopéptidos y fosfopéptidos se puede aplicar a varios tipos de muestras para lograr un perfil profundo de múltiples PTM e identificar posibles moléculas objetivo para futuros estudios.
Introduction
Las modificaciones post-traduccionales de proteínas (PTM) desempeñan un papel importante en la modulación de las estructuras proteicas y, en consecuencia, en sus funciones y procesos biológicos posteriores. La diversidad del proteoma humano aumenta exponencialmente debido a la variabilidad combinatoria que ofrecen varios PTM. Diferentes variantes de proteínas de sus secuencias canónicas predichas por el genoma se conocen como proteoformas, y muchos proteoformas surgen de PTM1. El estudio de la diversidad proteoforma en salud y enfermedad se ha convertido en un área de investigación de gran interés en los últimos años 2,3.
El estudio de proteoformas y más específicamente PTM con gran profundidad se ha vuelto más fácil a través del desarrollo de métodos proteómicos basados en espectrometría de masas (EM). Usando EM, los analitos se ionizan, fragmentan e identifican en función de los m /z de los fragmentos. Los métodos de enriquecimiento a menudo son necesarios debido a la baja abundancia relativa de PTM en comparación con las formas no modificadas de proteínas. Aunque el análisis de proteínas intactas y sus PTM, llamados análisis de arriba hacia abajo, se han vuelto más rutinarios, la digestión enzimática de las proteínas y el análisis de sus péptidos componentes en los análisis de abajo hacia arriba sigue siendo la ruta más utilizada para el análisis de PTM. Los dos PTM más ampliamente estudiados, y los dos PTM más comunes in vivo, son la glicosilación y la fosforilación4. Estos dos PTM desempeñan un papel importante en la señalización y el reconocimiento celular y, por lo tanto, son modificaciones importantes para caracterizar en la investigación de enfermedades.
Las propiedades químicas de varios PTM a menudo proporcionan rutas hacia el enriquecimiento de estos PTM a nivel de proteínas y péptidos antes del análisis. La glicosilación es un PTM hidrófilo debido a la abundancia de grupos hidroxilo en cada monosacárido. Esta propiedad se puede utilizar para enriquecer glicopéptidos en cromatografía de interacción hidrófila (HILIC), que puede separar más glicopéptidos hidrófilos de los péptidos hidrofóbicos no modificados5. La fosforilación agrega la fracción de fosfato, que está cargada negativamente excepto a pH ácido. Debido a esta carga, se pueden usar varios cationes metálicos, incluido el titanio, para atraer y unir fosfopéptidos mientras que las especies no fosforiladas se eliminan. Este es el principio de la cromatografía de afinidad de metales inmovilizados (IMAC). Se pueden encontrar más discusiones sobre estas y otras estrategias de enriquecimiento para la glicosilación y la fosforilación en revisiones recientes 6,7.
A menudo se necesitan cantidades comparativamente grandes de material peptídico de partida (0,5 mg o más) para los protocolos de enriquecimiento debido a la baja estequiometría de los PTM en los péptidos. En escenarios en los que esta cantidad de muestra puede no obtenerse fácilmente, como la biopsia del núcleo tumoral o los análisis del líquido cefalorraquídeo, es beneficioso utilizar flujos de trabajo fáciles que den como resultado la máxima información biomolecular. Las estrategias recientes desarrolladas por nuestro laboratorio y otros han destacado el análisis simultáneo y paralelo de la glicosilación y la fosforilación utilizando el mismo flujo de trabajo de enriquecimiento de PTM 8,9,10,11,12. Aunque las propiedades químicas de estos dos PTM pueden diferir, estos PTM pueden analizarse en múltiples pasos debido a las innovadoras técnicas de separación y materiales utilizados. Por ejemplo, la cromatografía de interacción repulsión-hidrofílica electrostática (ERLIC) superpone separaciones basadas en interacciones hidrófilas entre analitos y la fase móvil con interacciones carga-carga entre analitos y el material de fase estacionaria 13,14,15,16. A pH ácido, la atracción de péptidos fosforilados a la fase estacionaria puede mejorar su retención y separación de péptidos no modificados. El material que consiste en Ti(IV) inmovilizado en microesferas hidrofílicas se puede utilizar para la elución basada en HILIC e IMAC para separar fosfopéptidos y glicopéptidos neutros, ácidos y manosa-6-fosforilados17,18. Esta estrategia se conoce como Ti(IV)-IMAC de doble funcionalidad. El uso de estas estrategias para enriquecer varios PTM en un solo flujo de trabajo puede hacer que los análisis de las posibles interacciones de diafonía de PTM sean más accesibles. Además, la cantidad total de muestra y los requisitos de tiempo son menores que los métodos de enriquecimiento convencionales cuando se realizan en paralelo (es decir, HILIC e IMAC en alícuotas de muestra separadas).
Para demostrar la estrategia dual-funcional Ti(IV)-IMAC para el análisis simultáneo de la glicosilación y fosforilación de proteínas, la hemos aplicado para analizar tejidos pancreáticos humanos post-mortem. El páncreas produce tanto enzimas digestivas como hormonas reguladoras, incluyendo insulina y glucagón. La función pancreática se ve afectada en la enfermedad pancreática. En la diabetes, la regulación del azúcar en la sangre se ve afectada, lo que lleva a niveles más altos de glucosa en la sangre. En la pancreatitis, la inflamación resulta de la autodigestión del órgano3. Los cambios en los perfiles de PTM, incluida la glicosilación y la fosforilación, pueden resultar, como suele ser el caso, en otras enfermedades.
Aquí, describimos un protocolo para un método de enriquecimiento simultáneo basado en punta de espín, basado en una estrategia Ti(IV)-IMAC de doble funcionalidad, para N-glicopéptidos y fosfopéptidos derivados de proteínas extraídas del tejido pancreático. El protocolo incluye extracción y digestión de proteínas, enriquecimiento, recopilación de datos de EM y procesamiento de datos, como se puede ver en la Figura 1. Los datos representativos de este estudio están disponibles a través de ProteomeXchange Consortium con el identificador PXD033065.
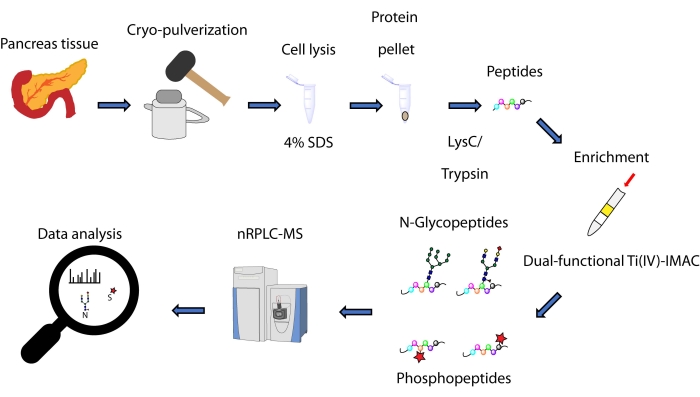
Figura 1: Flujo de trabajo para el análisis simultáneo de N-glicopéptidos y fosfopéptidos de tejidos pancreáticos humanos. Los tejidos primero se criopulforizan en un polvo fino antes de la extracción de proteínas utilizando el detergente dodecil sulfato de sodio (SDS). Las proteínas se someten a digestión enzimática. Los péptidos resultantes se alicitan antes del enriquecimiento utilizando Ti(IV)-IMAC de doble funcionalidad. Los datos brutos se recopilan utilizando cromatografía líquida de fase inversa a nanoescala-espectrometría de masas (nRPLC-MS) y se analizan utilizando un software de búsqueda de bases de datos. Haga clic aquí para ver una versión más grande de esta figura.
Este protocolo está destinado a hacer que los análisis ptm sean más accesibles y permitir un análisis más generalizado de múltiples PTM en el mismo flujo de trabajo. Este protocolo se puede aplicar a otras matrices biológicas complejas, incluidas las células y los biofluidos.
Protocol
Representative Results
Discussion
La estrategia Ti(IV)-IMAC de doble funcionalidad es útil para el análisis simultáneo de N-glicopéptidos y fosfopéptidos de la misma muestra en un solo flujo de trabajo de preparación de muestras. También se ha demostrado que los métodos basados en ERLIC realizan un enriquecimiento simultáneo de PTM. Ambas estrategias se han utilizado previamente para la cobertura profunda en los análisis de PTM14,18. Al adaptar el método Ti dual para disminuir el tiemp…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Esta investigación fue apoyada en parte por fondos de subvención de los NIH (R01DK071801, RF1AG052324, P01CA250972 y R21AG065728) y juvenile Diabetes Research Foundation (1-PNF-2016-250-S-B y SRA-2016-168-S-B). Los datos presentados aquí también se obtuvieron en parte a través del apoyo de un premio NIH / NCATS UL1TR002373 a través del Instituto de Investigación Clínica y Traslacional de la Universidad de Wisconsin. Los instrumentos Orbitrap se compraron a través del apoyo de una subvención de instrumentos compartidos de los NIH (NIH-NCRR S10RR029531) y la Oficina del Vicerrector de Investigación y Educación de Posgrado de la Universidad de Wisconsin-Madison. También nos gustaría agradecer el generoso apoyo de la Organización de Donación de Órganos y Tejidos de la Universidad de Wisconsin que proporcionó páncreas humano para la investigación y la ayuda de Dan Tremmel, la Dra. Sara D. Sackett y el Prof. Jon Odorico por proporcionar las muestras a nuestro laboratorio. Nuestro equipo de investigación desea dar un agradecimiento especial a las familias que donaron tejidos para este estudio. L.L. reconoce la subvención de los NIH S10OD025084, una subvención piloto de cáncer de páncreas del Centro oncológico Carbone de la Universidad de Wisconsin (233-AAI9632), así como una Cátedra Vilas distinguished Achievement y la Cátedra Charles Melbourne Johnson Distinguished Chair con fondos proporcionados por la Wisconsin Alumni Research Foundation y la Facultad de Farmacia de la Universidad de Wisconsin-Madison.
Materials
| Acetic Acid, Glacial (Certified ACS) | Fisher Scientific | A38S-500 | |
| Acetone (Certified ACS) | Fisher Scientific | A18-1 | |
| Acetonitrile, Optima LC/MS Grade | Fisher Scientific | A955-4 | |
| Ammonium Acetate (Crystalline/Certified ACS) | Fisher Scientific | A637-500 | |
| Ammonium Hydroxide (Certified ACS Plus) | Fisher Scientific | A669-212 | |
| Byonic software | Protein Metrics | n/a | Commercial software used for glycoproteomic analysis (https://proteinmetrics.com/byos/) |
| C18 BEH material | Waters | 186002353 | Material removed from column and used to pack nano capillaries (pulledto integrate tip used directly in line with instrument inlet) |
| CAE-Ti-IMAC, 100% | J&K Scientific | 2749380-1G | Material used for dual-functional Ti(IV)-IMAC; can also be used for conventional IMAC/conventional phosphopeptide enrichment |
| Cellcrusher kit | Cellcrusher | n/a | Used for grinding tissue samples into powder before extraction |
| Eppendorf 5424R Microcentrifuge | Fisher Scientific | 05-401-205 | For temperature-controlled centrifugation |
| cOmplete protease inhibitor cocktail tablets | Sigma | 11697498001 | |
| DTT, Molecular Grade (DL-Dithiothreitol) | Promega | V3151 | Protein reducing agent |
| Ethanol, 200 proof (100%), USP | Fisher | 22-032-601 | |
| Fisherbrand Analog Vortex Mixer | Fisher Scientific | 02-215-414 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (1.5 mL) | Fisher Scientific | 02-681-320 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (2 mL) | Fisher Scientific | 02-681-321 | |
| Fisherbrand Model 120 Sonic Dismembrator | Fisher Scientific | FB120110 | For sample lysis using ultrasonication |
| Formic Acid, 99.0+%, Optima LC/MS Grade | Fisher Scientific | A117-50 | |
| Fused silica capillary (75 μm inner diameter, 360 μm outer diameter) | Polymicro Technologies LLC | 100 m TSP075375 | For in-house pulled and packed columns with integrated emitter |
| Hydrofluoric acid (48 wt. % in H2O) | Sigma-Aldrich | 339261-100ML | Used for opening emitter of pulled capillary column |
| Iodoacetamide, BioUltra | Sigma | I1149-5G | Protein reducing reagent |
| MaxQuant software | n/a | n/a | Free software used for phosphoproteomic analysis (https://www.maxquant.org/) |
| Multi-therm Shaker with heating and cooling | Benchmark Scientific | H5000-HC | Heating block |
| Oasis HLB 1 cc Vac Cartridge, 10 mg Sorbent per Cartridge, 30 µm, 100/pk | Waters | 186000383 | Larger-scale cartridge desalting for tryptic digests (loading capacity approximately up to 1 mg each) |
| OMIX C18 pipette tips, 100 µL tip, 10 – 100 μL elution volume, 1 x 96 tips | Agilent | A57003100 | Smaller-scale packed pipette tip for desalting for enrichment elutions |
| P-2000 Micropipette Puller | Sutter Instrument Co. | P-2000/F | For pulling nano-capillary columns for LC-MS |
| PhosSTOP phosphatase inhibitor tablets | Sigma | 4906845001 | |
| Pierce BCA Protein Assay Kit | Thermo Fisher Scientific | 23225 | |
| Pierce Quantitative Colorimetric Peptide Assay | Thermo Fisher Scientific | 23275 | |
| PolySAX LP (12 μm, pore size 300 Å) | PolyLC | BMSX1203 | Material for strong anion-exchange chromatography used for ERLIC/conventional glycopeptide enrichment |
| Potassium Phosphate Monobasic (Crystalline/Certified ACS) | Fisher Scientific | P285-500 | |
| Pressure injection cell with integrated magnetic stirplate | Next Advance | PC77-MAG | For packing nano-capillary columns with stationary phase up to 2500 psi limit |
| Proteome Discoverer software | Thermo Fisher Scientific | n/a | Commercial software for proteomics anaysis (with integrated database searching software nodes) and data visualization (https://www.thermofisher.com/us/en/home/industrial/mass-spectrometry/liquid-chromatography-mass-spectrometry-lc-ms/lc-ms-software/multi-omics-data-analysis/proteome-discoverer-software.html) |
| SpeedVac SC110 Vacuum Concentrator Model SC110-120 | Savant | n/a | Centrifugal vacuum concentrator for drying samples (under heat) |
| SDS Solution, 10% Sodium Dodecyl Sulfate Solution, Molecular Biology/Electrophoresis | Fisher Scientific | BP2436200 | |
| Sequencing Grade Modified Trypsin | Promega | V5111 | |
| Sodium Chloride (Crystalline/Certified ACS) | Fisher Scientific | S271-500 | |
| TopTip, Empty, 10-200 µL, Pack of 96 | Glygen Corporation | TT2EMT.96 | Empty pipette tip with micron-sized hole used that can be used to pack chromatographic materials for enrichments, bundled with tube adapters |
| Triethylammonium bicarbonate buffer (TEAB, 1 M, pH 8.5 (volatile)) | Sigma | 90360-100ML | |
| Trifluoroacetic acid, Reagent Grade, 99% | Fisher Scientific | 60-017-61 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | Fisher Scientific | BP152-500 | |
| Trypsin/Lys-C Mix, Mass Spec Grade | Promega | V5071 | |
| Urea (Certified ACS) | Fisher Scientific | U15-500 | |
| Water, Optima LC/MS Grade | Fisher Scientific | W64 |
References
- Smith, L. M., Kelleher, N. L. Proteoform: a single term describing protein complexity. Nature Methods. 10 (3), 186-187 (2013).
- Pan, S., Brentnall, T. A., Chen, R. Glycoproteins and glycoproteomics in pancreatic cancer. World Journal of Gastroenterology. 22 (42), 9288-9299 (2016).
- Tabang, D. N., Ford, M., Li, L. Recent advances in mass spectrometry-based glycomic and glycoproteomic studies of pancreatic diseases. Frontiers in Chemistry. 9, 707387 (2021).
- Khoury, G. A., Baliban, R. C., Floudas, C. A. Proteome-wide post-translational modification statistics: frequency analysis and curation of the swiss-prot database. Scientific Reports. 1, 90 (2011).
- Alpert, A. J. Hydrophilic-interaction chromatography for the separation of peptides, nucleic acids and other polar compounds. Journal of Chromatography A. 499, 177-196 (1990).
- Riley, N. M., Bertozzi, C. R., Pitteri, S. J. A pragmatic guide to enrichment strategies for mass spectrometry-based glycoproteomics. Molecular & Cellular Proteomics. 20, 100029 (2020).
- Low, T. Y., et al. Widening the bottleneck of phosphoproteomics: Evolving strategies for phosphopeptide enrichment. Mass Spectrometry Reviews. 40 (4), 309-333 (2021).
- Cho, K. C., Chen, L., Hu, Y., Schnaubelt, M., Zhang, H. Developing workflow for simultaneous analyses of phosphopeptides and glycopeptides. ACS Chemical Biology. 14 (1), 58-66 (2019).
- Zhou, Y., et al. An integrated workflow for global, glyco-, and phospho-proteomic analysis of tumor tissues. Analytical Chemistry. 92 (2), 1842-1849 (2020).
- Tang, R., et al. Facile preparation of bifunctional adsorbents for efficiently enriching N-glycopeptides and phosphopeptides. Analytica Chimica Acta. 1144, 111-120 (2021).
- Wang, Z., Wang, J., Sun, N., Deng, C. A promising nanoprobe based on hydrophilic interaction liquid chromatography and immobilized metal affinity chromatography for capture of glycopeptides and phosphopeptides. Analytica Chimica Acta. 1067, 1-10 (2019).
- Glover, M. S., et al. Characterization of intact sialylated glycopeptides and phosphorylated glycopeptides from IMAC enriched samples by EThcD fragmentation: Toward combining phosphoproteomics and glycoproteomics. International Journal of Mass Spectrometry. 427, 35-42 (2018).
- Alpert, A. J. Electrostatic repulsion hydrophilic interaction chromatography for isocratic separation of charged solutes and selective isolation of phosphopeptides. Analytical Chemistry. 80 (1), 62-76 (2008).
- Cui, Y., et al. Counterion optimization dramatically improves selectivity for phosphopeptides and glycopeptides in electrostatic repulsion-hydrophilic interaction chromatography. Analytical Chemistry. 93 (22), 7908-7916 (2021).
- Cui, Y., et al. Finding the sweet spot in ERLIC mobile phase for simultaneous enrichment of N-Glyco and phosphopeptides. Journal of the American Society for Mass Spectrometry. 30 (12), 2491-2501 (2019).
- Tabang, D. N., et al. Analysis of pancreatic extracellular matrix protein post-translational modifications via electrostatic repulsion-hydrophilic interaction chromatography coupled with mass spectrometry. Molecular Omics. 17 (5), 652-664 (2021).
- Huang, J., et al. Dual-functional Titanium(IV) immobilized metal affinity chromatography approach for enabling large-scale profiling of protein Mannose-6-Phosphate glycosylation and revealing its predominant substrates. Analytical Chemistry. 91 (18), 11589-11597 (2019).
- Huang, J., et al. Dual-functional Ti(IV)-IMAC material enables simultaneous enrichment and separation of diverse glycopeptides and phosphopeptides. Analytical Chemistry. 93 (24), 8568-8576 (2021).
- Jami-Alahmadi, Y., Pandey, V., Mayank, A. K., Wohlschlegel, J. A. A robust method for packing high resolution C18 RP-nano-HPLC columns. Journal of Visualized Experiments: JoVE. (171), e62380 (2021).
- Bern, M., Kil, Y. J., Becker, C. Byonic: advanced peptide and protein identification software. Current Protocols in Bioinformatics. , (2012).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).
- Zhou, Y., et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets. Nature Communications. 10 (1), 1523 (2019).
- Zacharias, L. G., et al. HILIC and ERLIC enrichment of glycopeptides derived from breast and brain cancer cells. Journal of Proteome Research. 15 (10), 3624-3634 (2016).
- Yang, W., et al. Comparison of enrichment methods for intact N- and O-linked glycopeptides using strong anion exchange and hydrophilic interaction liquid chromatography. Analytical Chemistry. 89 (21), 11193-11197 (2017).
- Toghi Eshghi, S., Shah, P., Yang, W., Li, X., Zhang, H. GPQuest: A spectral library matching algorithm for site-specific assignment of tandem mass spectra to intact N-glycopeptides. Analytical Chemistry. 87 (10), 5181-5188 (2015).
- Liu, M. -. Q., et al. pGlyco 2.0 enables precision N-glycoproteomics with comprehensive quality control and one-step mass spectrometry for intact glycopeptide identification. Nature Communications. 8 (1), 438 (2017).
- Lu, L., Riley, N. M., Shortreed, M. R., Bertozzi, C. R., Smith, L. M. O-Pair Search with MetaMorpheus for O-glycopeptide characterization. Nature Methods. 17 (11), 1133-1138 (2020).
- Caval, T., Heck, A. J. R., Reiding, K. R. Meta-heterogeneity: Evaluating and describing the diversity in glycosylation between sites on the same glycoprotein. Molecular & Cellular Proteomics. 20, 100010 (2021).
- Lee, J. S., Smith, E., Shilatifard, A. The language of histone crosstalk. Cell. 142 (5), 682-685 (2010).
- Leutert, M., Entwisle, S. W., Villén, J. Decoding post-translational modification crosstalk with proteomics. Molecular & Cellular Proteomics. 20, 100129 (2021).
- Hart, G. W., Slawson, C., Ramirez-Correa, G., Lagerlof, O. Cross talk between O-GlcNAcylation and phosphorylation: Roles in signaling, transcription, and chronic disease. Annual Review of Biochemistry. 80 (1), 825-858 (2011).

