Une stratégie d’enrichissement spin-tip pour l’analyse simultanée des N-glycopeptides et des phosphopeptides des tissus pancréatiques humains
Summary
Les modifications post-traductionnelles (PTM) modifient les structures et les fonctions des protéines. Les méthodes d’enrichissement simultané de plusieurs types de PTM peuvent maximiser la couverture dans les analyses. Nous présentons un protocole utilisant la chromatographie d’affinité métallique immobilisée Ti(IV) double fonctionnelle suivie d’une spectrométrie de masse pour l’enrichissement et l’analyse simultanés de la protéine N-glycosylation et de la phosphorylation dans les tissus pancréatiques.
Abstract
La spectrométrie de masse peut fournir une couverture profonde des modifications post-traductionnelles (PTM), bien que l’enrichissement de ces modifications à partir de matrices biologiques complexes soit souvent nécessaire en raison de leur faible stœchiométrie par rapport aux analytes non modifiés. La plupart des flux de travail d’enrichissement des PTM sur les peptides dans les flux de travail protéomique ascendants, où les protéines sont digérées enzymatiquement avant que les peptides résultants ne soient analysés, n’enrichissent qu’un seul type de modification. C’est l’ensemble du complément des PTM, cependant, qui conduit à des fonctions biologiques, et l’enrichissement d’un seul type de PTM peut manquer une telle diaphonie des PTM. La diaphonie PTM a été observée entre la glycosylation des protéines et la phosphorylation, les deux PTM les plus courants dans les protéines humaines et aussi les deux PTM les plus étudiés en utilisant des flux de travail de spectrométrie de masse. En utilisant la stratégie d’enrichissement simultané décrite ici, les deux PTM sont enrichis à partir de tissu pancréatique humain post-mortem, une matrice biologique complexe. La chromatographie d’affinité métallique immobilisée Ti(IV) à double fonction est utilisée pour séparer simultanément diverses formes de glycosylation et de phosphorylation en plusieurs fractions dans une méthode pratique basée sur la pointe de spin, permettant des analyses en aval des interactions potentielles de diaphonie PTM. Ce flux de travail d’enrichissement pour les glyco- et phosphopeptides peut être appliqué à différents types d’échantillons afin d’obtenir un profilage en profondeur de plusieurs PTM et d’identifier des molécules cibles potentielles pour de futures études.
Introduction
Les modifications post-traductionnelles des protéines (PTM) jouent un rôle majeur dans la modulation des structures protéiques et, par conséquent, de leurs fonctions et des processus biologiques en aval. La diversité du protéome humain augmente de façon exponentielle en raison de la variabilité combinatoire offerte par divers PTM. Différentes variantes de protéines de leurs séquences canoniques prédites par le génome sont connues sous le nom de protéoformes, et de nombreux protéoformes proviennent de PTM1. L’étude de la diversité protéiforme en matière de santé et de maladie est devenue un domaine de recherche d’un grand intérêt ces dernières années 2,3.
L’étude des protéoformes et plus particulièrement des PTM à grande profondeur est devenue plus facile grâce au développement de méthodes protéomiques basées sur la spectrométrie de masse (SEP). En utilisant la SEP, les analytes sont ionisés, fragmentés et identifiés en fonction du m/z des fragments. Les méthodes d’enrichissement sont souvent nécessaires en raison de la faible abondance relative des PTM par rapport aux formes non modifiées de protéines. Bien que l’analyse des protéines intactes et de leurs PTM, appelées analyses descendantes, soit devenue plus courante, la digestion enzymatique des protéines et l’analyse de leurs peptides constitutifs dans les analyses ascendantes restent la voie la plus largement utilisée pour l’analyse PTM. Les deux PTM les plus étudiés, et les deux PTM les plus courants in vivo, sont la glycosylation et la phosphorylation4. Ces deux PTM jouent un rôle majeur dans la signalisation et la reconnaissance cellulaires et sont donc des modifications importantes à caractériser dans la recherche sur les maladies.
Les propriétés chimiques de divers PTM fournissent souvent des voies vers l’enrichissement de ces PTM aux niveaux de protéines et de peptides avant l’analyse. La glycosylation est un PTM hydrophile en raison de l’abondance des groupes hydroxyles sur chaque monosaccharide. Cette propriété peut être utilisée pour enrichir les glycopeptides en chromatographie d’interaction hydrophile (HILIC), qui peut séparer davantage de glycopeptides hydrophiles des peptides hydrophobes non modifiés5. La phosphorylation ajoute la fraction phosphate, qui est chargée négativement sauf à pH acide. En raison de cette charge, divers cations métalliques, y compris le titane, peuvent être utilisés pour attirer et lier les phosphopeptides tandis que les espèces non phosphorylées sont emportées. C’est le principe de la chromatographie d’affinité des métaux immobilisés (IMAC). D’autres discussions sur ces stratégies et d’autres stratégies d’enrichissement pour la glycosylation et la phosphorylation peuvent être trouvées dans des revues récentes 6,7.
Des quantités relativement importantes de matériel peptidique de départ (0,5 mg ou plus) sont souvent nécessaires pour les protocoles d’enrichissement en raison de la faible stœchiométrie des PTM sur les peptides. Dans les scénarios où cette quantité d’échantillon peut ne pas être facilement obtenue, comme la biopsie du noyau tumoral ou les analyses du liquide céphalo-rachidien, il est avantageux d’utiliser des flux de travail faciles qui aboutissent à un maximum d’informations biomoléculaires. Les stratégies récentes développées par notre laboratoire et d’autres ont mis en évidence l’analyse simultanée et parallèle de la glycosylation et de la phosphorylation en utilisant le même flux de travail d’enrichissement PTM 8,9,10,11,12. Bien que les propriétés chimiques de ces deux PTM puissent différer, ces PTM peuvent être analysés en plusieurs étapes en raison des techniques de séparation innovantes et des matériaux utilisés. Par exemple, la chromatographie d’interaction électrostatique répulsion-hydrophile (ERLIC) superpose des séparations basées sur les interactions hydrophiles entre les analytes et la phase mobile avec des interactions charge-charge entre les analytes et le matériau de phase stationnaire 13,14,15,16. À pH acide, l’attraction des peptides phosphorylés vers la phase stationnaire peut améliorer leur rétention et leur séparation des peptides non modifiés. Le matériau constitué de Ti(IV) immobilisé sur des microsphères hydrophiles peut être utilisé pour l’élution HILIC et IMAC afin de séparer les phosphopeptides et les glycopeptides neutres, acides et mannose-6-phosphorylés17,18. Cette stratégie est connue sous le nom de Ti(IV)-IMAC à double fonction. L’utilisation de ces stratégies pour enrichir plusieurs PTM dans un seul flux de travail peut rendre les analyses des interactions de diaphonie PTM potentielles plus accessibles. De plus, la quantité totale d’échantillon et les exigences en temps sont inférieures aux méthodes d’enrichissement conventionnelles lorsqu’elles sont effectuées en parallèle (c.-à-d. HILIC et IMAC sur des aliquotes d’échantillon distinctes).
Pour démontrer la stratégie Ti(IV)-IMAC à double fonction pour l’analyse simultanée de la glycosylation et de la phosphorylation des protéines, nous l’avons appliquée pour analyser les tissus pancréatiques humains post-mortem. Le pancréas produit à la fois des enzymes digestives et des hormones régulatrices, y compris l’insuline et le glucagon. La fonction pancréatique est altérée dans la maladie pancréatique. Dans le diabète, la régulation de la glycémie est affectée, conduisant à des niveaux plus élevés de glucose dans le sang. Dans la pancréatite, l’inflammation résulte de l’auto-digestion de l’organe3. Des changements dans les profils PTM, y compris la glycosylation et la phosphorylation, peuvent entraîner, comme c’est souvent le cas, d’autres maladies.
Nous décrivons ici un protocole pour une méthode d’enrichissement simultané basée sur la pointe de spin, basée sur une stratégie Ti(IV)-IMAC à double fonction, pour les N-glycopeptides et les phosphopeptides dérivés de protéines extraites du tissu pancréatique. Le protocole comprend l’extraction et la digestion des protéines, l’enrichissement, la collecte de données sur la SEP et le traitement des données, comme le montre la figure 1. Les données représentatives de cette étude sont disponibles via ProteomeXchange Consortium avec l’identifiant PXD033065.
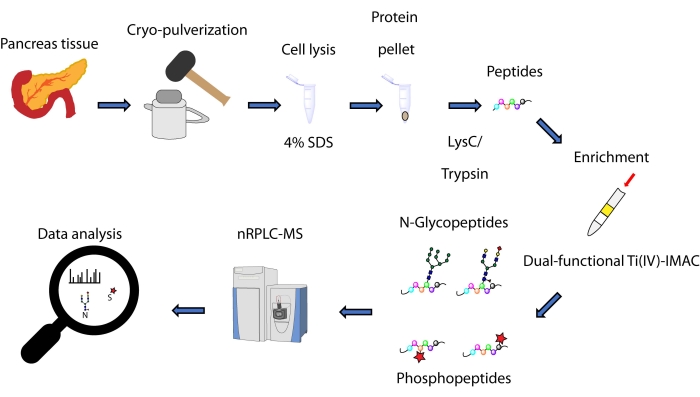
Figure 1 : Flux de travail pour l’analyse simultanée des N-glycopeptides et des phosphopeptides des tissus pancréatiques humains. Les tissus sont d’abord cryo-pulvérisés en une fine poudre avant l’extraction des protéines à l’aide du détergent dodécylsulfate de sodium (SDS). Les protéines sont ensuite soumises à une digestion enzymatique. Les peptides résultants sont alicités avant l’enrichissement à l’aide de Ti(IV)-IMAC à double fonction. Les données brutes sont collectées à l’aide de la chromatographie liquide en phase inversée à l’échelle nanométrique-spectrométrie de masse (nRPLC-MS) et sont analysées à l’aide d’un logiciel de recherche dans une base de données. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Ce protocole est destiné à rendre les analyses PTM plus accessibles et à permettre une analyse plus étendue de plusieurs PTM dans le même flux de travail. Ce protocole peut être appliqué à d’autres matrices biologiques complexes, y compris les cellules et les biofluides.
Protocol
Representative Results
Discussion
La stratégie Ti(IV)-IMAC à double fonction est utile pour l’analyse simultanée des N-glycopeptides et des phosphopeptides du même échantillon dans un seul flux de travail de préparation d’échantillon. Il a également été démontré que les méthodes basées sur ERLIC permettent d’enrichir simultanément les PTM. Les deux stratégies ont déjà été utilisées pour une couverture approfondie dans les analyses PTM14,18. En adaptant la méthode dual …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Cette recherche a été financée en partie par des subventions des NIH (R01DK071801, RF1AG052324, P01CA250972 et R21AG065728) et de la Juvenile Diabetes Research Foundation (1-PNF-2016-250-S-B et SRA-2016-168-S-B). Les données présentées ici ont également été obtenues en partie grâce au soutien d’un prix UL1TR002373 des NIH / NCATS par l’intermédiaire de l’Institut de recherche clinique et translationnelle de l’Université du Wisconsin. Les instruments Orbitrap ont été achetés grâce au soutien d’une subvention d’instruments partagés des NIH (NIH-NCRR S10RR029531) et du Bureau du vice-chancelier pour la recherche et l’enseignement supérieur de l’Université du Wisconsin-Madison. Nous tenons également à remercier le généreux soutien de l’Organisation de don d’organes et de tissus de l’Université du Wisconsin qui a fourni du pancréas humain pour la recherche et l’aide de Dan Tremmel, du Dr Sara D. Sackett et du professeur Jon Odorico pour fournir les échantillons à notre laboratoire. Notre équipe de recherche tient à remercier tout particulièrement les familles qui ont fait don de tissus pour cette étude. L.L. reconnaît la subvention S10OD025084 des NIH, une subvention pilote sur le cancer du pancréas du Carbone Cancer Center de l’Université du Wisconsin (233-AAI9632), ainsi qu’une chaire Vilas Distinguished Achievement et la chaire Charles Melbourne Johnson Distinguished Chair avec un financement fourni par la Wisconsin Alumni Research Foundation et l’Université du Wisconsin-Madison School of Pharmacy.
Materials
| Acetic Acid, Glacial (Certified ACS) | Fisher Scientific | A38S-500 | |
| Acetone (Certified ACS) | Fisher Scientific | A18-1 | |
| Acetonitrile, Optima LC/MS Grade | Fisher Scientific | A955-4 | |
| Ammonium Acetate (Crystalline/Certified ACS) | Fisher Scientific | A637-500 | |
| Ammonium Hydroxide (Certified ACS Plus) | Fisher Scientific | A669-212 | |
| Byonic software | Protein Metrics | n/a | Commercial software used for glycoproteomic analysis (https://proteinmetrics.com/byos/) |
| C18 BEH material | Waters | 186002353 | Material removed from column and used to pack nano capillaries (pulledto integrate tip used directly in line with instrument inlet) |
| CAE-Ti-IMAC, 100% | J&K Scientific | 2749380-1G | Material used for dual-functional Ti(IV)-IMAC; can also be used for conventional IMAC/conventional phosphopeptide enrichment |
| Cellcrusher kit | Cellcrusher | n/a | Used for grinding tissue samples into powder before extraction |
| Eppendorf 5424R Microcentrifuge | Fisher Scientific | 05-401-205 | For temperature-controlled centrifugation |
| cOmplete protease inhibitor cocktail tablets | Sigma | 11697498001 | |
| DTT, Molecular Grade (DL-Dithiothreitol) | Promega | V3151 | Protein reducing agent |
| Ethanol, 200 proof (100%), USP | Fisher | 22-032-601 | |
| Fisherbrand Analog Vortex Mixer | Fisher Scientific | 02-215-414 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (1.5 mL) | Fisher Scientific | 02-681-320 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (2 mL) | Fisher Scientific | 02-681-321 | |
| Fisherbrand Model 120 Sonic Dismembrator | Fisher Scientific | FB120110 | For sample lysis using ultrasonication |
| Formic Acid, 99.0+%, Optima LC/MS Grade | Fisher Scientific | A117-50 | |
| Fused silica capillary (75 μm inner diameter, 360 μm outer diameter) | Polymicro Technologies LLC | 100 m TSP075375 | For in-house pulled and packed columns with integrated emitter |
| Hydrofluoric acid (48 wt. % in H2O) | Sigma-Aldrich | 339261-100ML | Used for opening emitter of pulled capillary column |
| Iodoacetamide, BioUltra | Sigma | I1149-5G | Protein reducing reagent |
| MaxQuant software | n/a | n/a | Free software used for phosphoproteomic analysis (https://www.maxquant.org/) |
| Multi-therm Shaker with heating and cooling | Benchmark Scientific | H5000-HC | Heating block |
| Oasis HLB 1 cc Vac Cartridge, 10 mg Sorbent per Cartridge, 30 µm, 100/pk | Waters | 186000383 | Larger-scale cartridge desalting for tryptic digests (loading capacity approximately up to 1 mg each) |
| OMIX C18 pipette tips, 100 µL tip, 10 – 100 μL elution volume, 1 x 96 tips | Agilent | A57003100 | Smaller-scale packed pipette tip for desalting for enrichment elutions |
| P-2000 Micropipette Puller | Sutter Instrument Co. | P-2000/F | For pulling nano-capillary columns for LC-MS |
| PhosSTOP phosphatase inhibitor tablets | Sigma | 4906845001 | |
| Pierce BCA Protein Assay Kit | Thermo Fisher Scientific | 23225 | |
| Pierce Quantitative Colorimetric Peptide Assay | Thermo Fisher Scientific | 23275 | |
| PolySAX LP (12 μm, pore size 300 Å) | PolyLC | BMSX1203 | Material for strong anion-exchange chromatography used for ERLIC/conventional glycopeptide enrichment |
| Potassium Phosphate Monobasic (Crystalline/Certified ACS) | Fisher Scientific | P285-500 | |
| Pressure injection cell with integrated magnetic stirplate | Next Advance | PC77-MAG | For packing nano-capillary columns with stationary phase up to 2500 psi limit |
| Proteome Discoverer software | Thermo Fisher Scientific | n/a | Commercial software for proteomics anaysis (with integrated database searching software nodes) and data visualization (https://www.thermofisher.com/us/en/home/industrial/mass-spectrometry/liquid-chromatography-mass-spectrometry-lc-ms/lc-ms-software/multi-omics-data-analysis/proteome-discoverer-software.html) |
| SpeedVac SC110 Vacuum Concentrator Model SC110-120 | Savant | n/a | Centrifugal vacuum concentrator for drying samples (under heat) |
| SDS Solution, 10% Sodium Dodecyl Sulfate Solution, Molecular Biology/Electrophoresis | Fisher Scientific | BP2436200 | |
| Sequencing Grade Modified Trypsin | Promega | V5111 | |
| Sodium Chloride (Crystalline/Certified ACS) | Fisher Scientific | S271-500 | |
| TopTip, Empty, 10-200 µL, Pack of 96 | Glygen Corporation | TT2EMT.96 | Empty pipette tip with micron-sized hole used that can be used to pack chromatographic materials for enrichments, bundled with tube adapters |
| Triethylammonium bicarbonate buffer (TEAB, 1 M, pH 8.5 (volatile)) | Sigma | 90360-100ML | |
| Trifluoroacetic acid, Reagent Grade, 99% | Fisher Scientific | 60-017-61 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | Fisher Scientific | BP152-500 | |
| Trypsin/Lys-C Mix, Mass Spec Grade | Promega | V5071 | |
| Urea (Certified ACS) | Fisher Scientific | U15-500 | |
| Water, Optima LC/MS Grade | Fisher Scientific | W64 |
References
- Smith, L. M., Kelleher, N. L. Proteoform: a single term describing protein complexity. Nature Methods. 10 (3), 186-187 (2013).
- Pan, S., Brentnall, T. A., Chen, R. Glycoproteins and glycoproteomics in pancreatic cancer. World Journal of Gastroenterology. 22 (42), 9288-9299 (2016).
- Tabang, D. N., Ford, M., Li, L. Recent advances in mass spectrometry-based glycomic and glycoproteomic studies of pancreatic diseases. Frontiers in Chemistry. 9, 707387 (2021).
- Khoury, G. A., Baliban, R. C., Floudas, C. A. Proteome-wide post-translational modification statistics: frequency analysis and curation of the swiss-prot database. Scientific Reports. 1, 90 (2011).
- Alpert, A. J. Hydrophilic-interaction chromatography for the separation of peptides, nucleic acids and other polar compounds. Journal of Chromatography A. 499, 177-196 (1990).
- Riley, N. M., Bertozzi, C. R., Pitteri, S. J. A pragmatic guide to enrichment strategies for mass spectrometry-based glycoproteomics. Molecular & Cellular Proteomics. 20, 100029 (2020).
- Low, T. Y., et al. Widening the bottleneck of phosphoproteomics: Evolving strategies for phosphopeptide enrichment. Mass Spectrometry Reviews. 40 (4), 309-333 (2021).
- Cho, K. C., Chen, L., Hu, Y., Schnaubelt, M., Zhang, H. Developing workflow for simultaneous analyses of phosphopeptides and glycopeptides. ACS Chemical Biology. 14 (1), 58-66 (2019).
- Zhou, Y., et al. An integrated workflow for global, glyco-, and phospho-proteomic analysis of tumor tissues. Analytical Chemistry. 92 (2), 1842-1849 (2020).
- Tang, R., et al. Facile preparation of bifunctional adsorbents for efficiently enriching N-glycopeptides and phosphopeptides. Analytica Chimica Acta. 1144, 111-120 (2021).
- Wang, Z., Wang, J., Sun, N., Deng, C. A promising nanoprobe based on hydrophilic interaction liquid chromatography and immobilized metal affinity chromatography for capture of glycopeptides and phosphopeptides. Analytica Chimica Acta. 1067, 1-10 (2019).
- Glover, M. S., et al. Characterization of intact sialylated glycopeptides and phosphorylated glycopeptides from IMAC enriched samples by EThcD fragmentation: Toward combining phosphoproteomics and glycoproteomics. International Journal of Mass Spectrometry. 427, 35-42 (2018).
- Alpert, A. J. Electrostatic repulsion hydrophilic interaction chromatography for isocratic separation of charged solutes and selective isolation of phosphopeptides. Analytical Chemistry. 80 (1), 62-76 (2008).
- Cui, Y., et al. Counterion optimization dramatically improves selectivity for phosphopeptides and glycopeptides in electrostatic repulsion-hydrophilic interaction chromatography. Analytical Chemistry. 93 (22), 7908-7916 (2021).
- Cui, Y., et al. Finding the sweet spot in ERLIC mobile phase for simultaneous enrichment of N-Glyco and phosphopeptides. Journal of the American Society for Mass Spectrometry. 30 (12), 2491-2501 (2019).
- Tabang, D. N., et al. Analysis of pancreatic extracellular matrix protein post-translational modifications via electrostatic repulsion-hydrophilic interaction chromatography coupled with mass spectrometry. Molecular Omics. 17 (5), 652-664 (2021).
- Huang, J., et al. Dual-functional Titanium(IV) immobilized metal affinity chromatography approach for enabling large-scale profiling of protein Mannose-6-Phosphate glycosylation and revealing its predominant substrates. Analytical Chemistry. 91 (18), 11589-11597 (2019).
- Huang, J., et al. Dual-functional Ti(IV)-IMAC material enables simultaneous enrichment and separation of diverse glycopeptides and phosphopeptides. Analytical Chemistry. 93 (24), 8568-8576 (2021).
- Jami-Alahmadi, Y., Pandey, V., Mayank, A. K., Wohlschlegel, J. A. A robust method for packing high resolution C18 RP-nano-HPLC columns. Journal of Visualized Experiments: JoVE. (171), e62380 (2021).
- Bern, M., Kil, Y. J., Becker, C. Byonic: advanced peptide and protein identification software. Current Protocols in Bioinformatics. , (2012).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).
- Zhou, Y., et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets. Nature Communications. 10 (1), 1523 (2019).
- Zacharias, L. G., et al. HILIC and ERLIC enrichment of glycopeptides derived from breast and brain cancer cells. Journal of Proteome Research. 15 (10), 3624-3634 (2016).
- Yang, W., et al. Comparison of enrichment methods for intact N- and O-linked glycopeptides using strong anion exchange and hydrophilic interaction liquid chromatography. Analytical Chemistry. 89 (21), 11193-11197 (2017).
- Toghi Eshghi, S., Shah, P., Yang, W., Li, X., Zhang, H. GPQuest: A spectral library matching algorithm for site-specific assignment of tandem mass spectra to intact N-glycopeptides. Analytical Chemistry. 87 (10), 5181-5188 (2015).
- Liu, M. -. Q., et al. pGlyco 2.0 enables precision N-glycoproteomics with comprehensive quality control and one-step mass spectrometry for intact glycopeptide identification. Nature Communications. 8 (1), 438 (2017).
- Lu, L., Riley, N. M., Shortreed, M. R., Bertozzi, C. R., Smith, L. M. O-Pair Search with MetaMorpheus for O-glycopeptide characterization. Nature Methods. 17 (11), 1133-1138 (2020).
- Caval, T., Heck, A. J. R., Reiding, K. R. Meta-heterogeneity: Evaluating and describing the diversity in glycosylation between sites on the same glycoprotein. Molecular & Cellular Proteomics. 20, 100010 (2021).
- Lee, J. S., Smith, E., Shilatifard, A. The language of histone crosstalk. Cell. 142 (5), 682-685 (2010).
- Leutert, M., Entwisle, S. W., Villén, J. Decoding post-translational modification crosstalk with proteomics. Molecular & Cellular Proteomics. 20, 100129 (2021).
- Hart, G. W., Slawson, C., Ramirez-Correa, G., Lagerlof, O. Cross talk between O-GlcNAcylation and phosphorylation: Roles in signaling, transcription, and chronic disease. Annual Review of Biochemistry. 80 (1), 825-858 (2011).

