Una strategia di arricchimento spin-tip per l'analisi simultanea di N-glicopeptidi e fosfopeptidi da tessuti pancreatici umani
Summary
Le modifiche post-traduzionali (PTM) modificano le strutture e le funzioni delle proteine. I metodi per l’arricchimento simultaneo di più tipi di PTM possono massimizzare la copertura nelle analisi. Presentiamo un protocollo che utilizza cromatografia di affinità metallica immobilizzata Ti(IV) a doppia funzionalità seguita da spettrometria di massa per l’arricchimento e l’analisi simultanei della proteina N-glicosilazione e fosforilazione nei tessuti pancreatici.
Abstract
La spettrometria di massa può fornire una copertura approfondita delle modifiche post-traduzionali (PTM), sebbene l’arricchimento di queste modifiche da matrici biologiche complesse sia spesso necessario a causa della loro bassa stechiometria rispetto agli analiti non modificati. La maggior parte dei flussi di lavoro di arricchimento dei PTM sui peptidi nei flussi di lavoro di proteomica bottom-up, in cui le proteine vengono digerite enzimaticamente prima che i peptidi risultanti vengano analizzati, arricchiscono solo un tipo di modifica. È l’intero complemento di PTM, tuttavia, che porta a funzioni biologiche, e l’arricchimento di un singolo tipo di PTM può perdere tale diafonia di PTM. PTM crosstalk è stato osservato tra glicosilazione proteica e fosforilazione, i due PTM più comuni nelle proteine umane e anche i due PTM più studiati utilizzando flussi di lavoro di spettrometria di massa. Utilizzando la strategia di arricchimento simultaneo qui descritta, entrambi i PTM sono arricchiti da tessuto pancreatico umano post-mortem, una matrice biologica complessa. La cromatografia a simmetria metallica immobilizzata Ti(IV) a doppia funzionalità viene utilizzata per separare contemporaneamente varie forme di glicosilazione e fosforilazione in frazioni multiple in un comodo metodo basato sulla punta di spin, consentendo analisi a valle delle potenziali interazioni crosstalk PTM. Questo flusso di lavoro di arricchimento per glico- e fosfopeptidi può essere applicato a vari tipi di campioni per ottenere una profilazione profonda di più PTM e identificare potenziali molecole bersaglio per studi futuri.
Introduction
Le modificazioni post-traduzionali delle proteine (PTM) svolgono un ruolo importante nella modulazione delle strutture proteiche e di conseguenza delle loro funzioni e dei processi biologici a valle. La diversità del proteoma umano aumenta esponenzialmente a causa della variabilità combinatoria offerta da vari PTM. Diverse varianti di proteine dalle loro sequenze canoniche come previsto dal genoma sono conosciute come proteoforme e molte proteoforme derivano da PTM1. Lo studio della diversità proteoforme nella salute e nella malattia è diventato un’area di ricerca di grande interesse negli ultimi anni 2,3.
Lo studio delle proteoforme e più specificamente dei PTM con grande profondità è diventato più facile attraverso lo sviluppo di metodi di proteomica basati sulla spettrometria di massa (MS). Utilizzando la SM, gli analiti sono ionizzati, frammentati e identificati in base alla m/z dei frammenti. I metodi di arricchimento sono spesso necessari a causa della bassa abbondanza relativa di PTM rispetto alle forme non modificate di proteine. Sebbene l’analisi delle proteine intatte e dei loro PTM, chiamate analisi top-down, siano diventate più di routine, la digestione enzimatica delle proteine e l’analisi dei loro peptidi componenti nelle analisi bottom-up è ancora la via più utilizzata per l’analisi PTM. I due PTM più studiati, e i due PTM più comuni in vivo, sono la glicosilazione e la fosforilazione4. Questi due PTM svolgono un ruolo importante nella segnalazione e nel riconoscimento cellulare e quindi sono importanti modifiche da caratterizzare nella ricerca sulle malattie.
Le proprietà chimiche di vari PTM spesso forniscono percorsi verso l’arricchimento di questi PTM a livello di proteine e peptidi prima dell’analisi. La glicosilazione è un PTM idrofilo dovuto all’abbondanza di gruppi ossidrilici su ciascun monosaccaride. Questa proprietà può essere utilizzata per arricchire i glicopeptidi nella cromatografia di interazione idrofila (HILIC), che può separare più glicopeptidi idrofili dai peptidi idrofobici non modificati5. La fosforilazione aggiunge la porzione di fosfato, che è caricata negativamente tranne che a pH acido. A causa di questa carica, vari cationi metallici, incluso il titanio, possono essere utilizzati per attirare e legare i fosfopeptidi mentre le specie non fosforilate vengono lavate via. Questo è il principio della cromatografia di affinità del metallo immobilizzato (IMAC). Ulteriori discussioni su queste e altre strategie di arricchimento per la glicosilazione e la fosforilazione possono essere trovate in recenti recensioni 6,7.
Quantità relativamente grandi di materiale peptidico di partenza (0,5 mg o più) sono spesso necessarie per i protocolli di arricchimento a causa della bassa stechiometria dei PTM sui peptidi. In scenari in cui questa quantità di campione potrebbe non essere facilmente ottenibile, come la biopsia del nucleo tumorale o le analisi del liquido cerebrospinale, è utile utilizzare flussi di lavoro facili che si traducono nella massima informazione biomolecolare. Recenti strategie sviluppate dal nostro laboratorio e da altri hanno evidenziato l’analisi simultanea e parallela della glicosilazione e della fosforilazione utilizzando lo stesso flusso di lavoro di arricchimento PTM 8,9,10,11,12. Sebbene le proprietà chimiche di questi due PTM possano differire, questi PTM possono essere analizzati in più fasi a causa delle innovative tecniche di separazione e dei materiali utilizzati. Ad esempio, la cromatografia di interazione elettrostatica repulsione-idrofila (ERLIC) sovrappone separazioni basate su interazioni idrofile tra analiti e fase mobile con interazioni carica-carica tra analiti e materiale di fase stazionaria 13,14,15,16. A pH acido, l’attrazione dei peptidi fosforilati nella fase stazionaria può migliorare la loro ritenzione e separazione dai peptidi non modificati. Il materiale costituito da Ti(IV) immobilizzato su microsfere idrofile può essere utilizzato per l’eluizione a base di HILIC e IMAC per separare i fosfopeptidi e i glicopeptidi neutri, acidi e mannosio-6-fosforilati17,18. Questa strategia è nota come Ti(IV)-IMAC a doppia funzionalità. L’utilizzo di queste strategie per arricchire più PTM in un unico flusso di lavoro può rendere più accessibili le analisi delle potenziali interazioni crosstalk PTM. Inoltre, la quantità totale del campione e i requisiti di tempo sono inferiori ai metodi di arricchimento convenzionali se eseguiti in parallelo (ad esempio, HILIC e IMAC su aliquote di campione separate).
Per dimostrare la strategia ti(IV)-IMAC dual-funzionale per l’analisi simultanea della glicosilazione proteica e della fosforilazione, l’abbiamo applicata per analizzare i tessuti pancreatici umani post-mortem. Il pancreas produce sia enzimi digestivi che ormoni regolatori, tra cui insulina e glucagone. La funzione pancreatica è compromessa nella malattia pancreatica. Nel diabete, la regolazione della glicemia è influenzata, portando a livelli più elevati di glucosio nel sangue. Nella pancreatite, l’infiammazione deriva dall’autodigestione dell’organo3. I cambiamenti nei profili PTM, tra cui la glicosilazione e la fosforilazione, possono provocare, come spesso accade, in altre malattie.
Qui, descriviamo un protocollo per un metodo di arricchimento simultaneo basato su spin-tip, basato su una strategia Ti(IV)-IMAC a doppia funzionalità, per N-glicopeptidi e fosfopeptidi derivati da proteine estratte dal tessuto pancreatico. Il protocollo include l’estrazione e la digestione delle proteine, l’arricchimento, la raccolta dei dati MS e l’elaborazione dei dati, come si può vedere nella Figura 1. I dati rappresentativi di questo studio sono disponibili tramite ProteomeXchange Consortium con identificatore PXD033065.
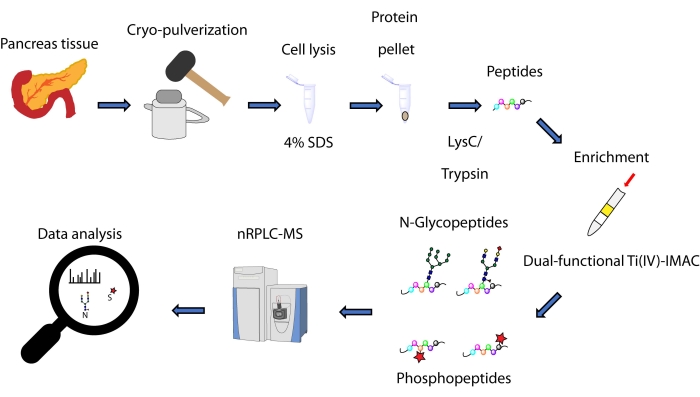
Figura 1: Flusso di lavoro per l’analisi simultanea di N-glicopeptidi e fosfopeptidi da tessuti pancreatici umani. I tessuti vengono prima crio-polverizzati in una polvere fine prima dell’estrazione delle proteine utilizzando il detergente sodio dodecil solfato (SDS). Le proteine vengono quindi sottoposte a digestione enzimatica. I peptidi risultanti vengono aliquotati prima dell’arricchimento utilizzando Ti(IV)-IMAC dual-funzionale. I dati grezzi vengono raccolti utilizzando la cromatografia liquida a fase inversa su scala nanometrica-spettrometria di massa (nRPLC-MS) e vengono analizzati utilizzando un software di ricerca di database. Fare clic qui per visualizzare una versione più grande di questa figura.
Questo protocollo ha lo scopo di rendere le analisi PTM più accessibili e di consentire un’analisi più diffusa di più PTM nello stesso flusso di lavoro. Questo protocollo può essere applicato ad altre matrici biologiche complesse, tra cui cellule e biofluidi.
Protocol
Representative Results
Discussion
La strategia Ti(IV)-IMAC dual-funzionale è utile per l’analisi simultanea di N-glicopeptidi e fosfopeptidi dallo stesso campione in un unico flusso di lavoro di preparazione del campione. I metodi basati su ERLIC hanno anche dimostrato di eseguire l’arricchimento simultaneo dei PTM. Entrambe le strategie sono state utilizzate in precedenza per una copertura profonda nelle analisi PTM14,18. Adattando il metodo dual Ti per ridurre il tempo di incubazione del campi…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Questa ricerca è stata supportata in parte da sovvenzioni del NIH (R01DK071801, RF1AG052324, P01CA250972 e R21AG065728) e della Juvenile Diabetes Research Foundation (1-PNF-2016-250-S-B e SRA-2016-168-S-B). I dati qui presentati sono stati anche in parte ottenuti grazie al supporto di un premio NIH/NCATS UL1TR002373 attraverso l’Istituto per la ricerca clinica e traslazionale dell’Università del Wisconsin. Gli strumenti Orbitrap sono stati acquistati attraverso il supporto di una sovvenzione per strumenti condivisi NIH (NIH-NCRR S10RR029531) e dell’Ufficio del Vice Cancelliere per la ricerca e l’istruzione universitaria presso l’Università del Wisconsin-Madison. Vorremmo anche riconoscere il generoso sostegno dell’Organizzazione per la donazione di organi e tessuti dell’Università del Wisconsin che ha fornito pancreas umano per la ricerca e l’aiuto di Dan Tremmel, Dr. Sara D. Sackett e Prof. Jon Odorico per aver fornito i campioni al nostro laboratorio. Il nostro team di ricerca desidera ringraziare in modo speciale le famiglie che hanno donato tessuti per questo studio. L.L. riconosce la sovvenzione NIH S10OD025084, una sovvenzione pilota per il cancro al pancreas del Carbone Cancer Center dell’Università del Wisconsin (233-AAI9632), nonché una Vilas Distinguished Achievement Professorship e la Charles Melbourne Johnson Distinguished Chair Professorship con finanziamenti forniti dalla Wisconsin Alumni Research Foundation e dalla University of Wisconsin-Madison School of Pharmacy.
Materials
| Acetic Acid, Glacial (Certified ACS) | Fisher Scientific | A38S-500 | |
| Acetone (Certified ACS) | Fisher Scientific | A18-1 | |
| Acetonitrile, Optima LC/MS Grade | Fisher Scientific | A955-4 | |
| Ammonium Acetate (Crystalline/Certified ACS) | Fisher Scientific | A637-500 | |
| Ammonium Hydroxide (Certified ACS Plus) | Fisher Scientific | A669-212 | |
| Byonic software | Protein Metrics | n/a | Commercial software used for glycoproteomic analysis (https://proteinmetrics.com/byos/) |
| C18 BEH material | Waters | 186002353 | Material removed from column and used to pack nano capillaries (pulledto integrate tip used directly in line with instrument inlet) |
| CAE-Ti-IMAC, 100% | J&K Scientific | 2749380-1G | Material used for dual-functional Ti(IV)-IMAC; can also be used for conventional IMAC/conventional phosphopeptide enrichment |
| Cellcrusher kit | Cellcrusher | n/a | Used for grinding tissue samples into powder before extraction |
| Eppendorf 5424R Microcentrifuge | Fisher Scientific | 05-401-205 | For temperature-controlled centrifugation |
| cOmplete protease inhibitor cocktail tablets | Sigma | 11697498001 | |
| DTT, Molecular Grade (DL-Dithiothreitol) | Promega | V3151 | Protein reducing agent |
| Ethanol, 200 proof (100%), USP | Fisher | 22-032-601 | |
| Fisherbrand Analog Vortex Mixer | Fisher Scientific | 02-215-414 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (1.5 mL) | Fisher Scientific | 02-681-320 | |
| Fisherbrand Low-Retention Microcentrifuge Tubes (2 mL) | Fisher Scientific | 02-681-321 | |
| Fisherbrand Model 120 Sonic Dismembrator | Fisher Scientific | FB120110 | For sample lysis using ultrasonication |
| Formic Acid, 99.0+%, Optima LC/MS Grade | Fisher Scientific | A117-50 | |
| Fused silica capillary (75 μm inner diameter, 360 μm outer diameter) | Polymicro Technologies LLC | 100 m TSP075375 | For in-house pulled and packed columns with integrated emitter |
| Hydrofluoric acid (48 wt. % in H2O) | Sigma-Aldrich | 339261-100ML | Used for opening emitter of pulled capillary column |
| Iodoacetamide, BioUltra | Sigma | I1149-5G | Protein reducing reagent |
| MaxQuant software | n/a | n/a | Free software used for phosphoproteomic analysis (https://www.maxquant.org/) |
| Multi-therm Shaker with heating and cooling | Benchmark Scientific | H5000-HC | Heating block |
| Oasis HLB 1 cc Vac Cartridge, 10 mg Sorbent per Cartridge, 30 µm, 100/pk | Waters | 186000383 | Larger-scale cartridge desalting for tryptic digests (loading capacity approximately up to 1 mg each) |
| OMIX C18 pipette tips, 100 µL tip, 10 – 100 μL elution volume, 1 x 96 tips | Agilent | A57003100 | Smaller-scale packed pipette tip for desalting for enrichment elutions |
| P-2000 Micropipette Puller | Sutter Instrument Co. | P-2000/F | For pulling nano-capillary columns for LC-MS |
| PhosSTOP phosphatase inhibitor tablets | Sigma | 4906845001 | |
| Pierce BCA Protein Assay Kit | Thermo Fisher Scientific | 23225 | |
| Pierce Quantitative Colorimetric Peptide Assay | Thermo Fisher Scientific | 23275 | |
| PolySAX LP (12 μm, pore size 300 Å) | PolyLC | BMSX1203 | Material for strong anion-exchange chromatography used for ERLIC/conventional glycopeptide enrichment |
| Potassium Phosphate Monobasic (Crystalline/Certified ACS) | Fisher Scientific | P285-500 | |
| Pressure injection cell with integrated magnetic stirplate | Next Advance | PC77-MAG | For packing nano-capillary columns with stationary phase up to 2500 psi limit |
| Proteome Discoverer software | Thermo Fisher Scientific | n/a | Commercial software for proteomics anaysis (with integrated database searching software nodes) and data visualization (https://www.thermofisher.com/us/en/home/industrial/mass-spectrometry/liquid-chromatography-mass-spectrometry-lc-ms/lc-ms-software/multi-omics-data-analysis/proteome-discoverer-software.html) |
| SpeedVac SC110 Vacuum Concentrator Model SC110-120 | Savant | n/a | Centrifugal vacuum concentrator for drying samples (under heat) |
| SDS Solution, 10% Sodium Dodecyl Sulfate Solution, Molecular Biology/Electrophoresis | Fisher Scientific | BP2436200 | |
| Sequencing Grade Modified Trypsin | Promega | V5111 | |
| Sodium Chloride (Crystalline/Certified ACS) | Fisher Scientific | S271-500 | |
| TopTip, Empty, 10-200 µL, Pack of 96 | Glygen Corporation | TT2EMT.96 | Empty pipette tip with micron-sized hole used that can be used to pack chromatographic materials for enrichments, bundled with tube adapters |
| Triethylammonium bicarbonate buffer (TEAB, 1 M, pH 8.5 (volatile)) | Sigma | 90360-100ML | |
| Trifluoroacetic acid, Reagent Grade, 99% | Fisher Scientific | 60-017-61 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | Fisher Scientific | BP152-500 | |
| Trypsin/Lys-C Mix, Mass Spec Grade | Promega | V5071 | |
| Urea (Certified ACS) | Fisher Scientific | U15-500 | |
| Water, Optima LC/MS Grade | Fisher Scientific | W64 |
References
- Smith, L. M., Kelleher, N. L. Proteoform: a single term describing protein complexity. Nature Methods. 10 (3), 186-187 (2013).
- Pan, S., Brentnall, T. A., Chen, R. Glycoproteins and glycoproteomics in pancreatic cancer. World Journal of Gastroenterology. 22 (42), 9288-9299 (2016).
- Tabang, D. N., Ford, M., Li, L. Recent advances in mass spectrometry-based glycomic and glycoproteomic studies of pancreatic diseases. Frontiers in Chemistry. 9, 707387 (2021).
- Khoury, G. A., Baliban, R. C., Floudas, C. A. Proteome-wide post-translational modification statistics: frequency analysis and curation of the swiss-prot database. Scientific Reports. 1, 90 (2011).
- Alpert, A. J. Hydrophilic-interaction chromatography for the separation of peptides, nucleic acids and other polar compounds. Journal of Chromatography A. 499, 177-196 (1990).
- Riley, N. M., Bertozzi, C. R., Pitteri, S. J. A pragmatic guide to enrichment strategies for mass spectrometry-based glycoproteomics. Molecular & Cellular Proteomics. 20, 100029 (2020).
- Low, T. Y., et al. Widening the bottleneck of phosphoproteomics: Evolving strategies for phosphopeptide enrichment. Mass Spectrometry Reviews. 40 (4), 309-333 (2021).
- Cho, K. C., Chen, L., Hu, Y., Schnaubelt, M., Zhang, H. Developing workflow for simultaneous analyses of phosphopeptides and glycopeptides. ACS Chemical Biology. 14 (1), 58-66 (2019).
- Zhou, Y., et al. An integrated workflow for global, glyco-, and phospho-proteomic analysis of tumor tissues. Analytical Chemistry. 92 (2), 1842-1849 (2020).
- Tang, R., et al. Facile preparation of bifunctional adsorbents for efficiently enriching N-glycopeptides and phosphopeptides. Analytica Chimica Acta. 1144, 111-120 (2021).
- Wang, Z., Wang, J., Sun, N., Deng, C. A promising nanoprobe based on hydrophilic interaction liquid chromatography and immobilized metal affinity chromatography for capture of glycopeptides and phosphopeptides. Analytica Chimica Acta. 1067, 1-10 (2019).
- Glover, M. S., et al. Characterization of intact sialylated glycopeptides and phosphorylated glycopeptides from IMAC enriched samples by EThcD fragmentation: Toward combining phosphoproteomics and glycoproteomics. International Journal of Mass Spectrometry. 427, 35-42 (2018).
- Alpert, A. J. Electrostatic repulsion hydrophilic interaction chromatography for isocratic separation of charged solutes and selective isolation of phosphopeptides. Analytical Chemistry. 80 (1), 62-76 (2008).
- Cui, Y., et al. Counterion optimization dramatically improves selectivity for phosphopeptides and glycopeptides in electrostatic repulsion-hydrophilic interaction chromatography. Analytical Chemistry. 93 (22), 7908-7916 (2021).
- Cui, Y., et al. Finding the sweet spot in ERLIC mobile phase for simultaneous enrichment of N-Glyco and phosphopeptides. Journal of the American Society for Mass Spectrometry. 30 (12), 2491-2501 (2019).
- Tabang, D. N., et al. Analysis of pancreatic extracellular matrix protein post-translational modifications via electrostatic repulsion-hydrophilic interaction chromatography coupled with mass spectrometry. Molecular Omics. 17 (5), 652-664 (2021).
- Huang, J., et al. Dual-functional Titanium(IV) immobilized metal affinity chromatography approach for enabling large-scale profiling of protein Mannose-6-Phosphate glycosylation and revealing its predominant substrates. Analytical Chemistry. 91 (18), 11589-11597 (2019).
- Huang, J., et al. Dual-functional Ti(IV)-IMAC material enables simultaneous enrichment and separation of diverse glycopeptides and phosphopeptides. Analytical Chemistry. 93 (24), 8568-8576 (2021).
- Jami-Alahmadi, Y., Pandey, V., Mayank, A. K., Wohlschlegel, J. A. A robust method for packing high resolution C18 RP-nano-HPLC columns. Journal of Visualized Experiments: JoVE. (171), e62380 (2021).
- Bern, M., Kil, Y. J., Becker, C. Byonic: advanced peptide and protein identification software. Current Protocols in Bioinformatics. , (2012).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Perez-Riverol, Y., et al. The PRIDE database and related tools and resources in 2019: improving support for quantification data. Nucleic Acids Research. 47, 442-450 (2019).
- Zhou, Y., et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets. Nature Communications. 10 (1), 1523 (2019).
- Zacharias, L. G., et al. HILIC and ERLIC enrichment of glycopeptides derived from breast and brain cancer cells. Journal of Proteome Research. 15 (10), 3624-3634 (2016).
- Yang, W., et al. Comparison of enrichment methods for intact N- and O-linked glycopeptides using strong anion exchange and hydrophilic interaction liquid chromatography. Analytical Chemistry. 89 (21), 11193-11197 (2017).
- Toghi Eshghi, S., Shah, P., Yang, W., Li, X., Zhang, H. GPQuest: A spectral library matching algorithm for site-specific assignment of tandem mass spectra to intact N-glycopeptides. Analytical Chemistry. 87 (10), 5181-5188 (2015).
- Liu, M. -. Q., et al. pGlyco 2.0 enables precision N-glycoproteomics with comprehensive quality control and one-step mass spectrometry for intact glycopeptide identification. Nature Communications. 8 (1), 438 (2017).
- Lu, L., Riley, N. M., Shortreed, M. R., Bertozzi, C. R., Smith, L. M. O-Pair Search with MetaMorpheus for O-glycopeptide characterization. Nature Methods. 17 (11), 1133-1138 (2020).
- Caval, T., Heck, A. J. R., Reiding, K. R. Meta-heterogeneity: Evaluating and describing the diversity in glycosylation between sites on the same glycoprotein. Molecular & Cellular Proteomics. 20, 100010 (2021).
- Lee, J. S., Smith, E., Shilatifard, A. The language of histone crosstalk. Cell. 142 (5), 682-685 (2010).
- Leutert, M., Entwisle, S. W., Villén, J. Decoding post-translational modification crosstalk with proteomics. Molecular & Cellular Proteomics. 20, 100129 (2021).
- Hart, G. W., Slawson, C., Ramirez-Correa, G., Lagerlof, O. Cross talk between O-GlcNAcylation and phosphorylation: Roles in signaling, transcription, and chronic disease. Annual Review of Biochemistry. 80 (1), 825-858 (2011).

