Un protocole complet pour la segmentation manuelle des structures du lobe temporal médial
Summary
The present work provides a comprehensive set of guidelines for manually tracing the medial temporal lobe (MTL) structures. This protocol can be applied to research involving structural and/or combined structural-functional magnetic resonance imaging (MRI) investigations of the MTL, in both healthy and clinical groups.
Abstract
Le présent document décrit un protocole complet pour le traçage manuel de l'ensemble des régions du cerveau comprenant le lobe temporal médian (MTL): amygdale, l'hippocampe et les régions associées parahippocampal (de périrhinal, entorhinal, et parahippocampal bon). Contrairement à la plupart des autres protocoles de recherches disponibles, en se concentrant généralement sur certaines zones de MTL (par exemple, l'amygdale et / ou hippocampe), la perspective d'intégration adopté par les présentes lignes directrices de traçage permet la localisation précise de tous les sous-régions de MTL. En intégrant l'information à partir d'une variété de sources, y compris les protocoles de traçabilité existantes ciblant séparément les différentes structures du LTM, les rapports histologiques, et atlas du cerveau, et avec le complément du matériel visuel d'illustration, le présent Protocole fournit un guide précis, intuitif et pratique pour comprendre la MTL anatomie. La nécessité de telles lignes directrices de recherchesest également souligné par illustrant les différences possibles entre les protocoles automatiques et manuelles segmentation. Cette connaissance peut être appliquée vers la recherche impliquant non seulement les enquêtes d'IRM structurelles mais aussi colocalisation et le signal IRMf extraction structurelle-fonctionnelle de ROI anatomiquement définies, dans les groupes en bonne santé et cliniques de même.
Introduction
Le lobe temporal médial (MTL), une zone supposée du plus haut niveau d'intégration de l'information sensorielle 1, a été un sujet fréquent d'analyses ciblées. Par exemple, l'hippocampe et les régions parahippocampal associés ont été étudiés dans la recherche de la mémoire 2-5. En outre, le rôle de l'amygdale a été souvent souligné dans la recherche d'examiner le traitement des émotions et interactions émotion-cognition 6-11. Récemment, des régions MTL ont également retenu l'attention dans le domaine émergent de la personnalité neurosciences, qui relie la structure et la fonction de celles-ci et d'autres régions du cerveau à des variations individuelles dans les traits de personnalité 12. L'évaluation de l'anatomie et la fonction des structures de MTL peut être important en facilitant le diagnostic des maladies dégénératives où des anomalies structurelles et fonctionnelles spécifiques peuvent se produire dans différentes structures du LTM. Par exemple, dans la maladie d'Alzheimer (AD), une importantetrophée du cortex entorhinal et de l'hippocampe peut être observé 13,14, et l'atrophie de l'hippocampe peut prédire la transition de la déficience cognitive légère à 15. Algorithmes de segmentation automatique sont récemment devenus populaires pour segmenter les structures corticales et sous-corticales, mais comme avec n'importe quel outil, ces programmes rencontrent inévitablement des erreurs dans certains cas. Dans de tels cas, un chercheur doit être équipé à la fois les connaissances et les lignes directrices pour reconnaître les frontières anatomiques des structures de MTL. La tendance dans la littérature existante a été de cibler les sous-régions de MTL individuels 16-21, avec de nombreux protocoles tendant à se concentrer sur l'hippocampe 16-19.
Contrairement à la plupart des lignes directrices publiées disponibles pour MTL traçage, le présent protocole fournit un ensemble complet de lignes directrices qui permettent la localisation précise de tous les sous-régions de MTL. Lignes directrices de traçage pour les structures de MTL suivantes sont décrites: l'amygdale (AMY), l'hippocampe (HC), le cortex périrhinal (RPC), le cortex entorhinal (CER), et le cortex para-hippocampique (SSP). L'AMY et le HC sont tracées d'abord, et sont suivis par le gyrus para-hippocampique (PHG) des structures. Notez que le HC terme générique est utilisé ici pour se référer à la formation HC, qui englobe le HC bon, le subiculum, et le segment postérieur de l'uncus 22-24. Notez également que le PHG peut être divisé en deux segments, la partie antérieure et la partie postérieure. Dans la partie antérieure de la PHG, il peut être divisé dans le PHG antéro-latéral et médial, dont les zones corticale correspondre à la République populaire de Chine et l'ERC, respectivement. Le SSP, la zone corticale de la partie postérieure de la PHG, correspond au cortex parahippocampique appropriée. Pour des raisons de simplicité, nous utiliserons les termes RPC et ERC de se référer à la partie antérieure PHG médial et latéral, et soins de santé primaires pour désigner le PHG postérieure. Le segmentation pour chaque structure commence avec une localisation approximative des frontières antérieures et postérieures, ainsi que d'autres sites pertinents, qui est ensuite suivi par le tracé effectivement réalisés tranche par tranche dans le plan frontal, dans un anterior-posterior/rostro-caudal direction. Dans tous les cas, les sections sagittales et axiales sont surveillés de près pour aider la localisation des frontières et des points de repère anatomiques.
La nécessité de ces directives traçage est également illustré dans les figures présentant les différences possibles entre la sortie de protocoles automatiques et manuelles segmentation. L'avantage d'un protocole qui décrit l'ensemble des structures du LTM dans le format visuel actuel est que les variations de l'anatomie (par exemple, le sillon collatéral [CS] de profondeur) qui peuvent affecter les définitions de frontières peuvent être décrites dans le contexte de l'anatomie environnante (par exemple, , la République populaire de Chine et ERC médial et bordures latérales varient dans un endroit en fonction de la profondeur de la CS 25 </sup>). Cela pourrait ne pas être clair ou compréhensible pour un traceur inexpérimenté ou un traceur expérimenté qui ne retrace structures simples ou séparés, et à notre connaissance, il n'existe pas une telle directive visuellement complet.
Le présent protocole est une présentation explicite des lignes directrices servant à MTL traçage dans une enquête précédente déterminer les contributions différentielles des sous-régions de MTL à l'effet de mémoire amélioration de l'émotion 26, adapté à la hausse des images de résolution du cerveau permis par l'évolution récente de résonance magnétique structurelle (MR) d'imagerie . Le tracé est illustré sur les scans obtenus à partir d'un volontaire sain (femme, 24 ans), en utilisant un scanner 3T MR. Images anatomiques ont été acquis 3D MPRAGE (TR = 1800 ms; TE = 2,26 ms; FOV = 256 x 256 mm, la taille de voxel = 1 x 0,5 x 0,5 mm) avec un angle parallèle de l'acquisition de AC-PC. Si des données d'image est acquise avec un angle d'acquisition différentes, comme l'orientation oblique, les données doivent être regdébarrassé de parallèle ou perpendiculaire à l'orientation AC-PC, tels que les descriptions de repère anatomiques traduisent de manière appropriée. Les images ont ensuite été convertis au format nifti et entrée dans un logiciel de segmentation 27 pour le traçage manuel. Les données de numérisation utilisés dans le protocole actuel ont été recueillies dans le cadre d'une étude qui a été approuvé par l'Institutional Review Board, et le bénévole consenti par écrit.
En tirant des informations de différents protocoles de traçabilité distinctes pour ces structures 18-22,28-31, ainsi que des analyses et des atlas anatomiques 23,32,33, le présent protocole présente un ensemble complet de lignes directrices qui traitent des incohérences dans la littérature existante. Complété par les matériaux visuels d'accompagnement, ce travail devrait favoriser meilleure compréhension des structures du LTM, et attiser l'intérêt de la recherche future dans l'adoption de la segmentation manuelle, soit en tant que principale méthode de MTL traçage ou une supplémentationméthode de ry de segmentation automatique. En fournissant un guide précis, intuitif et pratique pour comprendre l'anatomie de MTL, ce protocole aidera les chercheurs à identifier l'emplacement de toutes les sous-régions de MTL, par rapport à leurs structures voisines, même si seulement quelques structures de MTL sont spécifiquement ciblées pour les analyses. Cela permettra non seulement d'augmenter la précision de la localisation mais aussi d'aider les traceurs prendre des décisions éclairées en cas de variation morphologique, ce qui est très probable dans la MTL. Ces lignes directrices peuvent être appliquées à la recherche sur les enquêtes d'IRM structurelles et / ou fonctionnelles de la MTL, y compris les analyses volumétriques et la détection des anomalies du cerveau, ainsi que des procédures de localisation pour fonctionnel, anatomique et analyses tractographic, dans les groupes en bonne santé. Le présent protocole pourrait également être utilisé pour informer la segmentation des structures de MTL pour les patients (par exemple, les patients présentant une atrophie), si les principaux repères anatomiques sont relativement préservés. Tracing objet cliniqueLes données de S peut prendre du temps et des efforts supplémentaires, en fonction de la gravité de l'atrophie et / ou des modifications anatomiques.
Il est important de considérer la distinction entre circonvolutions corticales lors de la définition et ROI. Anatomiquement, gyrus ici désigne à la fois la matière blanche et la matière grise, tandis que le cortex se réfère à matière grise seulement. Selon l'utilisation prévue du retour sur investissement, segmentations peuvent inclure la substance blanche ou exclure.
Nous vous recommandons le dépistage à effectuer séquentiellement, structure par structure, un hémisphère à la fois. Certains logiciels permettent 34 pour tracer les frontières décrites sur une tranche à être collé sur les tranches suivantes, une fonctionnalité qui accélère le processus. Il est toujours recommandé de faire référence à l'hémisphère opposée au besoin, afin de vérifier la cohérence entre les deux côtés (par exemple, dans la détection de repères anatomiques). Alternativement, le suivi en parallèle des mêmes structures dans les deux hémisphères peuvent également être effectuées. Peu importe si le tracé est parallèle ou séquentielle, une fois que le processus est terminé, les traceurs doivent double-vérifier le résultat final et apporter les ajustements nécessaires, le référencement deux hémisphères et plusieurs vues d'avion. Selon l'expérience du traceur et la résolution des données d'imagerie, la segmentation manuelle de la MTL pour les données saines en question peut prendre du 8-10 h ou plus, dans le cas d'un traceur novice, à 3-4 h, dans le cas d'un expérimenté.
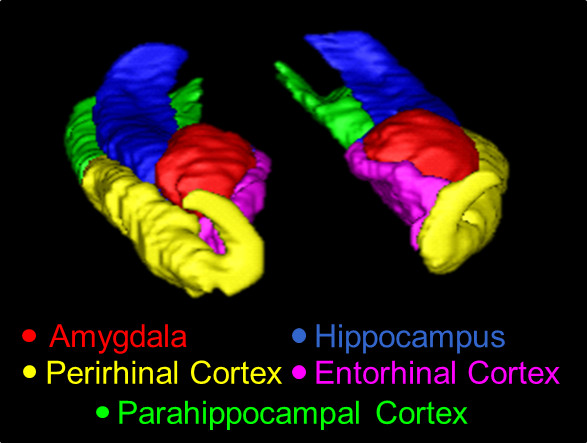
Aperçu Figure 1. 3D de la MTL, tracée en utilisant le présent protocole. Structures indiqués ici sont le AMY (rouge), le HC (bleu), la République populaire de Chine (jaune), l'ERC (rose), et la SSP (vert) .
Protocol
Representative Results
Discussion
Traditionnellement, la segmentation manuelle a été considéré comme l'étalon-or par de nombreux chercheurs. Néanmoins, une délimitation précise des structures individuelles a été compliquée par la morphologie très variable des structures du LTM, et par les faibles contrastes habituellement d'IRM de ces structures contre les zones de tissus et non-neuronales neurones environnants. Historiquement, il ya eu des descriptions contradictoires dans la littérature pour certaines structures de MTL. Dans certa…
Declarações
The authors have nothing to disclose.
Acknowledgements
This research was supported by funds to FD. MM was supported by an IGERT Fellowship under National Science Foundation Grant No. 0903622. The authors wish to thank the Dolcos Lab members for assistance with data collection and preparation.
Materials
| ITK-SNAP | ITK-SNAP Team at University of Pennsylvania and University of Utah | ITK-SNAP v2.2 | |
| FSL | Functional Magnetic Resonance Imaging of the Brain (FMRIB) Analysis Group | FSL v4.1 | |
| 3T Siemens Trio MR Scanner | Siemens | 3T Trio |
Referências
- Amaral, D. G. Introduction: what is where in the medial temporal lobe. Hippocampus. 9, 1-6 (1999).
- Squire, L. R., Zola-Morgan, S. The medial temporal lobe memory system. Science. 253 (5026), 1380-1386 (1991).
- Eichenbaum, H., Otto, T., Cohen, N. J. The hippocampus: what does it do. Behavioral & Neural Biology. 57 (1), 2-36 (1992).
- Henke, K., Buck, A., Weber, B., Wieser, H. G. Human hippocampus establishes associations in memory. Hippocampus. 7 (3), 249-256 (1997).
- Tulving, E., Markowitsch, H. J. Episodic and declarative memory: role of the hippocampus. Hippocampus. 8 (3), 198-204 (1998).
- Dolcos, F., Iordan, A. D., Dolcos, S. Neural correlates of emotion–cognition interactions: a review of evidence from brain imaging investigations. Journal of Cognitive Psychology. 23 (6), 669-694 (2011).
- Davidson, R. J., Irwin, W. The functional neuroanatomy of emotion and affective style. Trends in Cognitive Sciences. 3 (1), 11-21 (1999).
- Lindquist, K. A., Wager, T. D., Kober, H., Bliss-Moreau, E., Barrett, L. F. The brain basis of emotion: a meta-analytic review. The Behavioral and Brain Sciences. 35 (3), 121-143 (2012).
- Phan, K. L., Wager, T., Taylor, S. F., Liberzon, I. Functional neuroanatomy of emotion: a meta-analysis of emotion activation studies in PET and fMRI. Neuroimage. 16 (2), 331-348 (2002).
- Wager, T. D., Phan, K. L., Liberzon, I., Taylor, S. F. Valence, gender, and lateralization of functional brain anatomy in emotion: a meta-analysis of findings from neuroimaging. Neuroimage. 19, 513-531 (2003).
- Zald, D. H. The human amygdala and the emotional evaluation of sensory stimuli. Brain Research Reviews. 41, 88-123 (2003).
- DeYoung, C. G., Hirsh, J. B., Shane, M. S., Papademetris, X., Rajeevan, N., Gray, J. R. Testing predictions from personality neuroscience: brain structure and the big five. Psychological Science. 21 (6), 820-828 (2010).
- Visser, P. J., Verhey, F. R., Hofman, P. A., Scheltens, P., Jolles, J. Medial temporal lobe atrophy predicts Alzheimer’s disease in patients with minor cognitive impairment. Journal of Neurology, Neurosurgery, & Psychiatry. 72 (4), 491-497 (2002).
- Ezekiel, F., et al. Comparisons between global and focal brain atrophy rates in normal aging and Alzheimer disease. Alzheimer Disease & Associated Disorders. 18 (4), 196-201 (2004).
- de Leon, M. J., et al. Imaging and CSF studies in the preclinical diagnosis of Alzheimer’s disease. Annals of the New York Academy of Sciences. 1097, 114-145 (2007).
- Boccardi, M., et al. Survey of protocols for the manual segmentation of the hippocampus: preparatory steps towards a joint EADC-ADNI harmonized protocol. Journal of Alzheimer’s Disease. 26, 61-75 (2011).
- Konrad, C., Ukas, T., Nebel, C., Arolt, V., Toga, A. W., Narr, K. L. Defining the human hippocampus in cerebral magnetic resonance images-an overview of current segmentation protocols. Neuroimage. 47 (4), 1185-1195 (2009).
- Hasboun, D., et al. MR determination of hippocampal volume: comparison of three methods. American Journal of Neuroradiology. 17 (6), 1091-1098 (1996).
- Pantel, J., et al. A new method for the in vivo volumetric measurement of the human hippocampus with high neuroanatomical accuracy. Hippocampus. 10, 752-758 (2000).
- Entis, J. J., Doerga, P., Barrett, L. F., Dickerson, B. C. A reliable protocol for the manual segmentation of the human amygdala and its subregions using ultra-high resolution MRI. Neuroimage. 60 (2), 1226-1235 (2012).
- Goncharova, I. I., Dickerson, B. C., Stoub, T. R., deToledo-Morrell, L. MRI of human entorhinal cortex: a reliable protocol for volumetric measurement. Neurobiology of Aging. 22, 737-745 (2001).
- Watson, C., et al. Anatomic basis of amygdaloid and hippocampal volume measurement by magnetic resonance imaging. Neurology. 42 (9), 1743-1750 (1992).
- Duvernoy, H. The human hippocampus: functional anatomy, vascularization, and serial sections with MRI. Third Edition. , (2005).
- Amaral, D. G., Witter, M. P. The three-dimensional organization of the hippocampal formation: a review of anatomical data. Neurociência. 31 (3), 571-591 (1989).
- Insausti, R., et al. MR volumetric analysis of the human entorhinal, perirhinal, and temporopolar cortices. American Journal of Neuroradiology. 19 (4), 659-671 (1998).
- Dolcos, F., LaBar, K. S., Cabeza, R. Interaction between the amygdala and the medial temporal lobe memory system predicts better memory for emotional events. Neuron. 42 (5), 855-863 (2004).
- Yushkevich, P. A., et al. User-guided 3D active contour segmentation of anatomical structures: significantly improved efficiency and reliability. Neuroimage. 31 (3), 1116-1128 (2006).
- Bonilha, L., Kobayashi, E., Cendes, F., Li, M. L. Protocol for volumetric segmentation of medial temporal structures using high-resolution 3-D magnetic resonance imaging. Human Brain Mapping. 22 (2), 145-154 (2004).
- Bronen, R. A., Cheung, G. Relationship of hippocampus and amygdala to coronal MRI landmarks. Magnetic Resonance Imaging. 9 (3), 449-457 (1991).
- Pruessner, J. C., et al. Volumetry of temporopolar, perirhinal, entorhinal and parahippocampal cortex from high-resolution MR images: considering the variability of the collateral sulcus. Cerebral Cortex. 12 (12), 1342-1353 (2002).
- Pruessner, J. C., et al. Volumetry of hippocampus and amygdala with high-resolution MRI and three-dimensional analysis software: minimizing the discrepancies between laboratories. Cerebral Cortex. 10 (4), 433-442 (2000).
- Duvernoy, H. . The human brain: surface, three-dimensional sectional anatomy with MRI, and blood supply. Second Edition. , (1999).
- Amaral, D. G., Lavenex, P., et al. . in The hippocampus book. Hippocampal neuroanatomy. , (2006).
- Blaizot, X., et al. The human parahippocampal region: I. temporal pole cytoarchitectonic and MRI correlation. Cerebral Cortex. 20 (9), 2198-2212 (2010).
- Ding, S. -. L., Van Hoesen, G. W. Borders, extent, and topography of human perirhinal cortex as revealed using multiple modern neuroanatomical and pathological markers. Human Brain Mapping. 31 (9), 1359-1379 (2010).
- Ding, S. -. L., Van Hoesen, G. W., Cassell, M. D., Poremba, A. Parcellation of human temporal polar cortex: a combined analysis of multiple cytoarchitectonic, chemoarchitectonic, and pathological markers. The Journal of Comparative Neurology. 514 (6), 595-623 (2009).
- Frankó, E., Insausti, A. M., Artacho-Pérula, E., Insausti, R., Chavoix, C. Identification of the human medial temporal lobe regions on magnetic resonance images. Human Brain Mapping. 35 (1), 248-256 (2014).
- Lehmann, M., et al. Atrophy patterns in Alzheimer’s disease and semantic dementia: a comparison of FreeSurfer and manual volumetric measurements. Neuroimage. 49 (3), 2264-2274 (2010).
- Winterburn, J. L., et al. A novel in vivo atlas of human hippocampal subfields using high-resolution 3T magnetic resonance imaging. Neuroimage. 74, 254-265 (2013).
- Malykhin, N. V., Bouchard, T. P., Ogilvie, C. J., Coupland, N. J., Seres, P., Camicioli, R. Three-dimensional volumetric analysis and reconstruction of amygdala and hippocampal head, body and tail. Psychiatry research. Neuroimaging. 155 (2), 155-165 (2007).
- Patenaude, B., Smith, S. M., Kennedy, D. N., Jenkinson, M. A Bayesian model of shape and appearance for subcortical brain segmentation. Neuroimage. 56 (3), 907-922 (2011).
- Smith, S. M., et al. Advances in functional and structural MR image analysis and implementation as FSL. Neuroimage. 23, (2004).
- Woolrich, M. W., et al. Bayesian analysis of neuroimaging data in FSL. Neuroimage. 45, (2009).
- Smith, S. M. Fast robust automated brain extraction. Human Brain Mapping. 17 (3), 143-155 (2002).
- Morey, R. A., et al. A comparison of automated segmentation and manual tracing for quantifying hippocampal and amygdala volumes. Neuroimage. 45 (3), 855-866 (2009).
- Baldassano, C., Beck, D. M., Fei-Fei, L. Differential connectivity within the parahippocampal place area. Neuroimage. 75, 228-237 (2013).
- Shrout, P. E., Fleiss, J. L. Intraclass correlations: uses in assessing rater reliability. Psychological Bulletin. 86 (2), 420-428 (1979).
- Bland, J. M., Altman, D. G. A note on the use of the intraclass correlation coefficient in the evaluation of agreement between two methods of measurement. Computers in Biology and Medicine. 20, 337-340 (1990).
- Yushkevich, P. A., et al. A high-resolution computational atlas of the human hippocampus from postmortem magnetic resonance imaging at 9.4 T. Neuroimage. 44 (2), 385-398 (2009).

