Un protocolo integral para la segmentación manual de las estructuras mediales Temporal Lobe
Summary
The present work provides a comprehensive set of guidelines for manually tracing the medial temporal lobe (MTL) structures. This protocol can be applied to research involving structural and/or combined structural-functional magnetic resonance imaging (MRI) investigations of the MTL, in both healthy and clinical groups.
Abstract
El presente documento describe un protocolo integral de trazado manual del conjunto de las regiones del cerebro que comprende el lóbulo temporal medial (MTL): la amígdala, el hipocampo y las regiones del hipocampo asociadas (perirrinal, entorrinal y del hipocampo propiamente dicho). A diferencia de la mayoría de los protocolos de seguimiento disponibles, por lo general se centra en ciertas áreas MTL (por ejemplo, la amígdala y / o hipocampo), la perspectiva integradora adoptada por las presentes directrices de rastreo permite la localización clara de todas las subregiones MTL. Mediante la integración de la información de una variedad de fuentes, incluyendo los protocolos de rastreo existentes orientación separado diversas estructuras MTL, informes histológicos y atlas del cerebro, y con el complemento de los materiales visuales ilustrativos, el presente protocolo proporciona una guía precisa, intuitiva y conveniente para la comprensión de la anatomía MTL. La necesidad de tales directrices rastreoTambién se pone de relieve mediante la ilustración de las posibles diferencias entre los protocolos de segmentación automática y manual. Este conocimiento puede ser aplicada a la investigación que involucra no sólo a las investigaciones de resonancia magnética estructural sino también colocalización y la señal fMRI extracción estructural-funcional de regiones de interés definidas anatómicamente, en grupos sanos y clínicos por igual.
Introduction
El lóbulo temporal medial (MTL), una zona putativa del más alto nivel de integración de la información sensorial 1, ha sido un tema frecuente de análisis específicos. Por ejemplo, el hipocampo y las áreas del hipocampo asociados se han estudiado ampliamente en la investigación de la memoria 2-5. Además, el papel de la amígdala se ha destacado con frecuencia en investigaciones que examinen procesamiento de las emociones y las interacciones emoción-cognición 6-11. Recientemente, varias regiones MTL también han sido objeto de atención en el emergente campo de la neurociencia de la personalidad, que une la estructura y función de estas y otras regiones del cerebro a la variación individual en rasgos de personalidad 12. La evaluación de la anatomía y función de las estructuras MTL puede ser importante para facilitar el diagnóstico de las enfermedades degenerativas que se pueden producir anomalías estructurales y funcionales específicos en diferentes estructuras MTL. Por ejemplo, en la enfermedad de Alzheimer (EA), una significativatrofeo de la corteza entorrinal y el hipocampo se puede observar 13,14, y la atrofia del hipocampo puede predecir la transición de deterioro cognitivo leve a AD 15. Algoritmos de segmentación automáticos han vuelto populares para la segmentación de las estructuras corticales y subcorticales, pero como con cualquier herramienta, estos programas encontrarán inevitablemente errores en algunos casos. En tales casos, un investigador debe estar equipado con los conocimientos y directrices para reconocer los límites anatómicos de las estructuras MTL. La tendencia en la literatura existente es dirigirse a las subregiones MTL individuales 16-21, con muchos protocolos que tienden a centrarse en el hipocampo 16-19.
A diferencia de la mayoría de las guías publicadas disponibles para el rastreo MTL, el presente protocolo proporciona un amplio conjunto de directrices que permitan la localización clara de todas las subregiones MTL. Se describen las pautas de rastreo para las siguientes estructuras MTL: la amígdala (AMI), el hipocampo (HC), la corteza perirrinal (PRC), la corteza entorrinal (ERC), y la corteza del hipocampo (APS). El AMY y la HC se trazan primero, y luego son seguidos por la circunvolución del hipocampo (PHG) estructuras. Tenga en cuenta que el HC término genérico se utiliza aquí para referirse a la formación de HC, que abarca la HC adecuada, el subículo, y el segmento posterior de la Uncus 22-24. Además, observe que la PHG se puede dividir en dos segmentos, la porción anterior y la porción posterior. Dentro de la porción anterior de la PHG, puede ser dividida en el PHG anterior lateral y medial, cuyas áreas corticales corresponden a la República Popular China y de la ERC, respectivamente. El APS, el área cortical de la parte posterior de la PHG, corresponde a la corteza del hipocampo adecuada. Por razones de simplicidad, usaremos los términos PRC y ERC para referirse al PHG anterior lateral y medial, y la atención primaria de salud para referirse a la PHG posterior. El segmentation para cada estructura comienza con una localización aproximada de las fronteras anteriores y posteriores, así como otros puntos de interés relevantes, que es seguido por el trazado real realizado rebanada por rebanada en el plano coronal, en un anterior-posterior/rostro-caudal dirección. En todos los casos, las secciones sagital y axial se siguen de cerca para ayudar a la localización de los límites anatómicos y monumentos.
La necesidad de tales directrices de rastreo también se ilustra en las figuras que muestran las posibles diferencias entre la salida de los protocolos de segmentación automática y manual. La ventaja de un protocolo que describe todas las estructuras MTL en el formato visual actual es que las variaciones en la anatomía (por ejemplo, el surco colateral profundidad [CA]) que puede afectar a las definiciones de frontera puede ser descrito en el contexto de la anatomía circundante (por ejemplo, , la República Popular China y ERC bordes medial y lateral varían en ubicación dependiendo de la profundidad de la CS 25 </sarriba>). Esto podría no ser clara o comprensible para un trazador inexperto o un trazador con experiencia que sólo rastrea las estructuras individuales o separados, y hasta donde sabemos, no existe tal directiva visualmente incompleta.
El presente protocolo es una presentación explícita de los criterios que utiliza para el rastreo de MTL en una investigación previa identificación de las contribuciones diferenciales de las subregiones MTL en el sentido de aumentar la memoria de la emoción 26, adaptado a las imágenes de resolución más altas del cerebro permitidos por la evolución reciente de resonancia magnética estructural (RM) . El trazado se ilustra en las imágenes obtenidas a partir de un voluntario sano (mujer, 24 años), utilizando un escáner 3T MR. Imágenes anatómicas fueron adquiridos como 3D MPRAGE (TR = 1800 ms, TE = 2,26 ms; FOV = 256 x 256 mm; tamaño de voxel = 1 x 0,5 x 0,5 mm) con un ángulo paralelo adquisición de AC-PC. Si los datos de imagen se adquiere con un ángulo de adquisición diferente, tales como la orientación oblicua, los datos deben ser regridded a una paralela u orientación perpendicular a AC-PC, de manera que las descripciones históricas anatómicas traducen adecuadamente. Las imágenes fueron convertidas a formato NIfTI y la entrada en el software de segmentación 27 para el rastreo manual. Escaneado de datos utilizados en el protocolo actual se recogieron como parte de un estudio que fue aprobado por la Junta de Revisión Institucional, y el voluntario dado su consentimiento por escrito.
Al llamar la información de varios protocolos de trazado independientes para estas estructuras 18-22,28-31, así como de los análisis anatómicos y atlas 23,32,33, el presente protocolo presenta un amplio conjunto de directrices que abordan las inconsistencias en la literatura existente. Complementado por los materiales visuales que se acompañan, se espera que este trabajo para promover la comprensión más clara de las estructuras MTL, y revuelva el interés de la investigación futura en la adopción de la segmentación manual, ya sea como principal método de rastreo MTL o como suplementaciónmétodo ry para la segmentación automática. Al proporcionar una guía precisa, intuitiva y conveniente para la comprensión de la anatomía MTL, este protocolo ayudará a los investigadores a identificar la ubicación de todas las subregiones MTL, con relación a sus estructuras vecinas, incluso cuando sólo algunas estructuras MTL están destinados específicamente para los análisis. Esto no sólo aumentará la precisión de la localización, pero también ayudará a los trazadores a tomar decisiones informadas en los casos de variación morfológica, que es altamente probable que en el MTL. Estas directrices se pueden aplicar a la investigación con RM investigaciones estructurales y / o funcionales del MTL, incluidos los análisis volumétricos y cerebro detección de anomalías, así como los procedimientos de localización de funcional, anatómica y análisis tractographic, en grupos sanos. El presente Protocolo también podría utilizarse para informar a la segmentación de las estructuras MTL para los pacientes (por ejemplo, pacientes con atrofia), si los principales puntos de referencia anatómicos están relativamente preservadas. Rastreo sujeto clínicadatos de s 'puede llevar tiempo y esfuerzo adicional, dependiendo de la gravedad de la atrofia y / o cambios anatómicos.
Es importante tener en cuenta la distinción entre circunvoluciones y cortezas en la definición de ROI. Anatómicamente, el giro que aquí se refiere tanto a la sustancia blanca y sustancia gris, mientras que la corteza se refiere a la materia gris solamente. Dependiendo del uso previsto de la ROI, segmentaciones pueden incluir la materia blanca o excluye.
Recomendamos que el trazado se realiza secuencialmente, subestructura de subestructura, un hemisferio a la vez. Algunos paquetes de software de 34 permitir el seguimiento de las fronteras señaladas en una rebanada de ser pegado en rodajas posteriores, una función que acelera el proceso. Siempre es conveniente hacer referencia al hemisferio oposición, según sea necesario, con el fin de comprobar la coherencia entre las dos partes (por ejemplo, en la detección de puntos de referencia anatómicos). Como alternativa, el trazado en paralelo de las mismas estructuras dentro de los dos hemisferios también se pueden realizar. Sin importar si el trazado es secuencial o en paralelo, una vez que el proceso se haya completado, los trazadores deben a comprobar el resultado final y hacer los ajustes necesarios, haciendo referencia a los dos hemisferios y múltiples vistas en planta. Dependiendo de la experiencia del trazador y la resolución de los datos de las imágenes, la segmentación manual de la MTL de datos sujetos sanos puede tardar 8-10 horas o más, en el caso de un trazador novato, a 3-4 horas, en el caso de un experimentado.
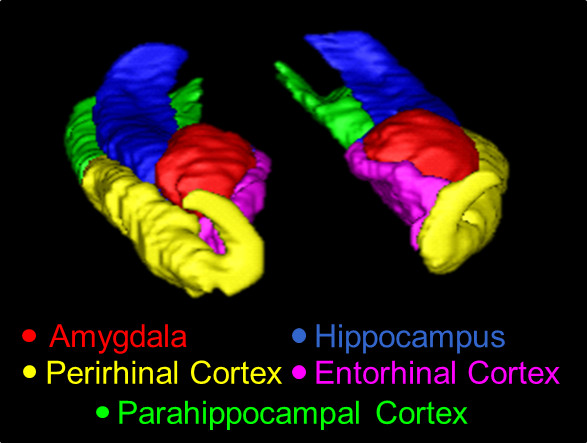
Generalidades Figura 1. 3D de la MTL, trazada utilizando el presente protocolo. Estructuras que aparecen aquí son el AMY (rojo), el HC (azul), la República Popular China (amarillo), la CEI (rosa), y la atención primaria de salud (verde) .
Protocol
Representative Results
Discussion
Tradicionalmente, la segmentación manual se ha considerado el estándar de oro por muchos investigadores. Sin embargo, una delimitación precisa de las estructuras individuales se ha complicado por la morfología muy variable de las estructuras MTL, y por los contrastes de resonancia magnética por lo general débiles de estas estructuras contra las zonas circundantes de tejido y no neurales neural. Históricamente, ha habido descripciones conflictivas en la literatura para algunas estructuras MTL. En algunos casos de …
Declarações
The authors have nothing to disclose.
Acknowledgements
This research was supported by funds to FD. MM was supported by an IGERT Fellowship under National Science Foundation Grant No. 0903622. The authors wish to thank the Dolcos Lab members for assistance with data collection and preparation.
Materials
| ITK-SNAP | ITK-SNAP Team at University of Pennsylvania and University of Utah | ITK-SNAP v2.2 | |
| FSL | Functional Magnetic Resonance Imaging of the Brain (FMRIB) Analysis Group | FSL v4.1 | |
| 3T Siemens Trio MR Scanner | Siemens | 3T Trio |
Referências
- Amaral, D. G. Introduction: what is where in the medial temporal lobe. Hippocampus. 9, 1-6 (1999).
- Squire, L. R., Zola-Morgan, S. The medial temporal lobe memory system. Science. 253 (5026), 1380-1386 (1991).
- Eichenbaum, H., Otto, T., Cohen, N. J. The hippocampus: what does it do. Behavioral & Neural Biology. 57 (1), 2-36 (1992).
- Henke, K., Buck, A., Weber, B., Wieser, H. G. Human hippocampus establishes associations in memory. Hippocampus. 7 (3), 249-256 (1997).
- Tulving, E., Markowitsch, H. J. Episodic and declarative memory: role of the hippocampus. Hippocampus. 8 (3), 198-204 (1998).
- Dolcos, F., Iordan, A. D., Dolcos, S. Neural correlates of emotion–cognition interactions: a review of evidence from brain imaging investigations. Journal of Cognitive Psychology. 23 (6), 669-694 (2011).
- Davidson, R. J., Irwin, W. The functional neuroanatomy of emotion and affective style. Trends in Cognitive Sciences. 3 (1), 11-21 (1999).
- Lindquist, K. A., Wager, T. D., Kober, H., Bliss-Moreau, E., Barrett, L. F. The brain basis of emotion: a meta-analytic review. The Behavioral and Brain Sciences. 35 (3), 121-143 (2012).
- Phan, K. L., Wager, T., Taylor, S. F., Liberzon, I. Functional neuroanatomy of emotion: a meta-analysis of emotion activation studies in PET and fMRI. Neuroimage. 16 (2), 331-348 (2002).
- Wager, T. D., Phan, K. L., Liberzon, I., Taylor, S. F. Valence, gender, and lateralization of functional brain anatomy in emotion: a meta-analysis of findings from neuroimaging. Neuroimage. 19, 513-531 (2003).
- Zald, D. H. The human amygdala and the emotional evaluation of sensory stimuli. Brain Research Reviews. 41, 88-123 (2003).
- DeYoung, C. G., Hirsh, J. B., Shane, M. S., Papademetris, X., Rajeevan, N., Gray, J. R. Testing predictions from personality neuroscience: brain structure and the big five. Psychological Science. 21 (6), 820-828 (2010).
- Visser, P. J., Verhey, F. R., Hofman, P. A., Scheltens, P., Jolles, J. Medial temporal lobe atrophy predicts Alzheimer’s disease in patients with minor cognitive impairment. Journal of Neurology, Neurosurgery, & Psychiatry. 72 (4), 491-497 (2002).
- Ezekiel, F., et al. Comparisons between global and focal brain atrophy rates in normal aging and Alzheimer disease. Alzheimer Disease & Associated Disorders. 18 (4), 196-201 (2004).
- de Leon, M. J., et al. Imaging and CSF studies in the preclinical diagnosis of Alzheimer’s disease. Annals of the New York Academy of Sciences. 1097, 114-145 (2007).
- Boccardi, M., et al. Survey of protocols for the manual segmentation of the hippocampus: preparatory steps towards a joint EADC-ADNI harmonized protocol. Journal of Alzheimer’s Disease. 26, 61-75 (2011).
- Konrad, C., Ukas, T., Nebel, C., Arolt, V., Toga, A. W., Narr, K. L. Defining the human hippocampus in cerebral magnetic resonance images-an overview of current segmentation protocols. Neuroimage. 47 (4), 1185-1195 (2009).
- Hasboun, D., et al. MR determination of hippocampal volume: comparison of three methods. American Journal of Neuroradiology. 17 (6), 1091-1098 (1996).
- Pantel, J., et al. A new method for the in vivo volumetric measurement of the human hippocampus with high neuroanatomical accuracy. Hippocampus. 10, 752-758 (2000).
- Entis, J. J., Doerga, P., Barrett, L. F., Dickerson, B. C. A reliable protocol for the manual segmentation of the human amygdala and its subregions using ultra-high resolution MRI. Neuroimage. 60 (2), 1226-1235 (2012).
- Goncharova, I. I., Dickerson, B. C., Stoub, T. R., deToledo-Morrell, L. MRI of human entorhinal cortex: a reliable protocol for volumetric measurement. Neurobiology of Aging. 22, 737-745 (2001).
- Watson, C., et al. Anatomic basis of amygdaloid and hippocampal volume measurement by magnetic resonance imaging. Neurology. 42 (9), 1743-1750 (1992).
- Duvernoy, H. The human hippocampus: functional anatomy, vascularization, and serial sections with MRI. Third Edition. , (2005).
- Amaral, D. G., Witter, M. P. The three-dimensional organization of the hippocampal formation: a review of anatomical data. Neurociência. 31 (3), 571-591 (1989).
- Insausti, R., et al. MR volumetric analysis of the human entorhinal, perirhinal, and temporopolar cortices. American Journal of Neuroradiology. 19 (4), 659-671 (1998).
- Dolcos, F., LaBar, K. S., Cabeza, R. Interaction between the amygdala and the medial temporal lobe memory system predicts better memory for emotional events. Neuron. 42 (5), 855-863 (2004).
- Yushkevich, P. A., et al. User-guided 3D active contour segmentation of anatomical structures: significantly improved efficiency and reliability. Neuroimage. 31 (3), 1116-1128 (2006).
- Bonilha, L., Kobayashi, E., Cendes, F., Li, M. L. Protocol for volumetric segmentation of medial temporal structures using high-resolution 3-D magnetic resonance imaging. Human Brain Mapping. 22 (2), 145-154 (2004).
- Bronen, R. A., Cheung, G. Relationship of hippocampus and amygdala to coronal MRI landmarks. Magnetic Resonance Imaging. 9 (3), 449-457 (1991).
- Pruessner, J. C., et al. Volumetry of temporopolar, perirhinal, entorhinal and parahippocampal cortex from high-resolution MR images: considering the variability of the collateral sulcus. Cerebral Cortex. 12 (12), 1342-1353 (2002).
- Pruessner, J. C., et al. Volumetry of hippocampus and amygdala with high-resolution MRI and three-dimensional analysis software: minimizing the discrepancies between laboratories. Cerebral Cortex. 10 (4), 433-442 (2000).
- Duvernoy, H. . The human brain: surface, three-dimensional sectional anatomy with MRI, and blood supply. Second Edition. , (1999).
- Amaral, D. G., Lavenex, P., et al. . in The hippocampus book. Hippocampal neuroanatomy. , (2006).
- Blaizot, X., et al. The human parahippocampal region: I. temporal pole cytoarchitectonic and MRI correlation. Cerebral Cortex. 20 (9), 2198-2212 (2010).
- Ding, S. -. L., Van Hoesen, G. W. Borders, extent, and topography of human perirhinal cortex as revealed using multiple modern neuroanatomical and pathological markers. Human Brain Mapping. 31 (9), 1359-1379 (2010).
- Ding, S. -. L., Van Hoesen, G. W., Cassell, M. D., Poremba, A. Parcellation of human temporal polar cortex: a combined analysis of multiple cytoarchitectonic, chemoarchitectonic, and pathological markers. The Journal of Comparative Neurology. 514 (6), 595-623 (2009).
- Frankó, E., Insausti, A. M., Artacho-Pérula, E., Insausti, R., Chavoix, C. Identification of the human medial temporal lobe regions on magnetic resonance images. Human Brain Mapping. 35 (1), 248-256 (2014).
- Lehmann, M., et al. Atrophy patterns in Alzheimer’s disease and semantic dementia: a comparison of FreeSurfer and manual volumetric measurements. Neuroimage. 49 (3), 2264-2274 (2010).
- Winterburn, J. L., et al. A novel in vivo atlas of human hippocampal subfields using high-resolution 3T magnetic resonance imaging. Neuroimage. 74, 254-265 (2013).
- Malykhin, N. V., Bouchard, T. P., Ogilvie, C. J., Coupland, N. J., Seres, P., Camicioli, R. Three-dimensional volumetric analysis and reconstruction of amygdala and hippocampal head, body and tail. Psychiatry research. Neuroimaging. 155 (2), 155-165 (2007).
- Patenaude, B., Smith, S. M., Kennedy, D. N., Jenkinson, M. A Bayesian model of shape and appearance for subcortical brain segmentation. Neuroimage. 56 (3), 907-922 (2011).
- Smith, S. M., et al. Advances in functional and structural MR image analysis and implementation as FSL. Neuroimage. 23, (2004).
- Woolrich, M. W., et al. Bayesian analysis of neuroimaging data in FSL. Neuroimage. 45, (2009).
- Smith, S. M. Fast robust automated brain extraction. Human Brain Mapping. 17 (3), 143-155 (2002).
- Morey, R. A., et al. A comparison of automated segmentation and manual tracing for quantifying hippocampal and amygdala volumes. Neuroimage. 45 (3), 855-866 (2009).
- Baldassano, C., Beck, D. M., Fei-Fei, L. Differential connectivity within the parahippocampal place area. Neuroimage. 75, 228-237 (2013).
- Shrout, P. E., Fleiss, J. L. Intraclass correlations: uses in assessing rater reliability. Psychological Bulletin. 86 (2), 420-428 (1979).
- Bland, J. M., Altman, D. G. A note on the use of the intraclass correlation coefficient in the evaluation of agreement between two methods of measurement. Computers in Biology and Medicine. 20, 337-340 (1990).
- Yushkevich, P. A., et al. A high-resolution computational atlas of the human hippocampus from postmortem magnetic resonance imaging at 9.4 T. Neuroimage. 44 (2), 385-398 (2009).

