Eine umfassende Protokoll für die manuelle Segmentierung der medialen Temporallappen Structures
Summary
The present work provides a comprehensive set of guidelines for manually tracing the medial temporal lobe (MTL) structures. This protocol can be applied to research involving structural and/or combined structural-functional magnetic resonance imaging (MRI) investigations of the MTL, in both healthy and clinical groups.
Abstract
Der vorliegende Beitrag beschreibt ein umfassendes Protokoll für die manuelle Verfolgung der Satz von Hirnregionen, die den medialen Temporallappen (MTL): Amygdala, Hippocampus, und die damit verbundenen parahippocampal Regionen (perirhinal, entorhinalen und parahippocampalen richtige). Anders als die meisten anderen Tracing Protokolle zur Verfügung, in der Regel die Konzentration auf bestimmte Bereiche MTL (zB Amygdala und / oder Hippocampus), ermöglicht die klare Lokalisierung aller MTL Teilbereiche der durch die vorliegenden Leitlinien verabschiedet Verfolgung integrative Perspektive. Durch die Integration von Informationen aus einer Vielzahl von Quellen, einschließlich erhaltene Verfolgung Protokolle separat Targeting verschiedenen MTL-Strukturen, histologische Berichte, Atlanten und Gehirn, und mit dem Komplement der illustrativen Bildmaterial stellt die vorliegende Protokoll eine präzise, intuitive und komfortable Führung für das Verständnis der MTL Anatomie. Die Notwendigkeit einer solchen Verfolgungs Richtlinienist auch durch die Darstellung mögliche Unterschiede zwischen automatischer und manueller Segmentierung Protokolle hervorgehoben. Dieses Wissen kann in Richtung Forschung, die nicht nur strukturelle MRT-Untersuchungen angewendet werden, sondern auch Struktur-Funktions Kolokalisations-und fMRI-Signal-Extraktion aus anatomisch definierten ROIs, in gesunden und klinischen Gruppen gleichermaßen.
Introduction
Der mediale Temporallappen (MTL), eine mutmaßliche Bereich der höchsten Ebene der Integration von Sinnesinformation 1, war ein häufiges Thema der gezielten Analysen. Zum Beispiel Hippocampus und zugehörigen parahippocampal Bereichen wurden ausführlich im Speicher Forschungs 2-5 untersucht. Auch die Rolle der Amygdala wurde häufig in der Forschung untersucht Emotionsverarbeitung und Emotion-Kognition Wechselwirkungen 6-11 betont. Kürzlich haben verschiedene MTL Regionen auch in dem neuen Gebiet der Neurowissenschaften Persönlichkeit, die die Struktur und Funktion von diesen und anderen Hirnregionen verknüpft, um individuelle Unterschiede in Persönlichkeitsmerkmale 12 aufgenommen Aufmerksamkeit. Beurteilung der Anatomie und Funktion der MTL Strukturen können zur Erleichterung der Diagnose von degenerativen Erkrankungen, bei denen spezifische strukturelle und funktionelle Anomalien können in verschiedenen Strukturen auftreten MTL wichtig sein. Zum Beispiel bei Alzheimer-Krankheit (AD), eine bedeutendeTrophäe des entorhinalen Cortex und Hippocampus beobachtet werden 13,14, und Atrophie des Hippocampus kann den Übergang von der leichten kognitiven Beeinträchtigung AD 15 vorherzusagen. Automatische Segmentierungsalgorithmen haben vor kurzem populär für die Segmentierung kortikalen und subkortikalen Strukturen geworden, aber wie bei jedem Werkzeug, diese Programme zwangsläufig Fehler auftreten, in einigen Fällen. In solchen Fällen sollte ein Forscher sowohl mit dem Wissen und Richtlinien, um die anatomischen Grenzen der MTL-Strukturen zu erkennen ausgestattet werden. Die Tendenz in der vorhandenen Literatur war es, einzelne Teilbereiche gezielt MTL 16-21, mit vielen Protokollen eher auf Hippocampus 16-19 konzentrieren.
Anders als die meisten der verfügbaren veröffentlichten Richtlinien für MTL Verfolgung, stellt die vorliegende Protokoll eine umfassende Reihe von Richtlinien, die für eine klare Lokalisierung aller MTL Teilbereiche zu ermöglichen. Tracing Leitlinien für die folgenden MTL-Strukturen werden beschrieben: die Amygdala (AMY), der Hippocampus (HC), die perirhinal Kortex (VR China), dem entorhinalen Kortex (ERC) und die parahippocampal Kortex (PHC). AMY und die HC werden zuerst zurückgeführt, und werden dann durch den Gyrus dentatus (PHG) Strukturen verfolgt. Beachten Sie, dass der Oberbegriff HC wird hier verwendet, um die HC-Bildung, die den HC richtige umfasst, der subiculum, und dem hinteren Segment des uncus 22-24 beziehen. Beachten Sie auch, dass der PHG kann in zwei Segmente, den vorderen Teil und dem hinteren Teil unterteilt werden. Im vorderen Teil des PHG, kann es weiter in die lateralen und medialen Vorder PHG, deren kortikale Bereiche entsprechen der VR China und der ERC bzw. geteilt werden. Die PHC, der kortikale Bereich des hinteren Teils des PHG, entspricht der parahippocampal Kortex richtig. Der Einfachheit halber werden wir mit den Begriffen Volksrepublik China und ERC auf die lateralen und medialen Vorder PHG beziehen und PHC zu der hinteren PHG beziehen. Die segmentation für jede Struktur beginnt mit einer groben Lokalisierung der vorderen und hinteren Ränder, zusammen mit anderen relevanten Landmarken, die dann von der tatsächlichen Spur in der Koronarebene durchgeführt Scheibe-für-Scheibe folgt, in einer anterior-posterior/rostro-caudal Richtung. In allen Fällen werden die sagittalen und axialen Abschnitten eng überwacht werden, um die Lokalisierung der anatomischen Grenzen und Sehenswürdigkeiten unterstützen.
Die Notwendigkeit einer solchen Verfolgungs Richtlinien auch in Zahlen anzeigt mögliche Unterschiede zwischen dem Ausgang der automatischen und manuellen Segmentierung Protokolle dargestellt. Der Vorteil eines Protokolls, das alle der MTL-Strukturen in der visuellen Format beschrieben ist, daß Variationen in der Anatomie (z. B. die Sicherungsfurche [CS]-Anteil), die Grenzdefinitionen beeinflussen kann, kann in Zusammenhang mit der umgebenden Anatomie (beschrieben beispiels , die VR China und ERC medialen und lateralen Grenzen variieren in der Lage, je nach Tiefe der CS 25 </sbis>). Dies könnte nicht klar oder verständlich zu einem unerfahrenen Tracer oder einem erfahrenen Tracer, die nur Spuren ein-oder getrennte Strukturen, und nach unserem Wissen nicht so ein umfassender Leitfaden visuell nicht existieren.
Das vorliegende Protokoll ist eine explizite Darstellung von Leitlinien für die MTL-Tracing in einer früheren Untersuchung identifiziert Differenzbeiträge MTL Teilbereiche auf die Speicherfördernde Wirkung von Emotion 26, um eine höhere Auflösung Gehirn Bilder von den jüngsten Entwicklungen in der strukturellen Magnetresonanz (MR)-Bildgebung erlaubt angepasst verwendet . Die Ablaufverfolgung ist auf Scans von einem gesunden Freiwilligen (weiblich, im Alter von 24) erhalten dargestellt, mit einem 3T MR-Scanner. Mit einem Anschaffungs Winkel parallel zur AC-PC Anatomische Bilder wurden als 3D-MPRAGE (; TE = 2,26 ms;; FOV = 256 x 256 mm Voxelgröße = 1 x 0,5 x 0,5 mm TR = 1800 ms) erworben. Wenn Bilddaten mit einem unterschiedlichen Erfassungswinkel, wie z. B. Schrägorientierung erfasst, sollten die Daten reg seinentledigt zu einem parallel oder senkrecht zur Ausrichtung AC-PC, so dass anatomische Wahrzeichen Beschreibungen übersetzen entsprechend. Die Bilder wurden dann zu NIfTI Format und Eingabe in 27 Segmentierung Software für Handverfolgung übersetzt. Scan-Daten in der aktuellen Protokoll wurde im Rahmen einer Studie, die vom Institutional Review Board genehmigt wurde, und der Freiwilligen mit schriftlicher Einwilligung erhoben.
Indem Informationen aus verschiedenen separaten Verfolgung Protokolle für diese Strukturen 18-22,28-31, als auch von anatomischen Analysen und Atlanten 23,32,33, präsentiert das vorliegende Protokoll eine umfassende Reihe von Richtlinien, die Ungereimtheiten in der vorhandenen Literatur zu adressieren. Durch die begleitenden visuellen Materialien Ergänzt wird diese Arbeit voraussichtlich klareres Verständnis der MTL-Strukturen fördern und wecken Interesse der Zukunftsforschung bei der Annahme manuelle Segmentierung, entweder als primäre Methode der MTL-Tracing oder als Ergänzungry-Methode, um die automatische Segmentierung. Durch eine genaue, intuitive und komfortable Führung für das Verständnis der Anatomie MTL, wird dieses Protokoll helfen Forscher identifizieren die Lage aller Teilbereiche MTL, relativ zu ihren benachbarten Strukturen, auch wenn nur einige MTL-Strukturen sind speziell für gezielte Analysen. Dies wird nicht nur Lokalisierungsgenauigkeit zu erhöhen, sondern wird auch dazu beitragen Tracer fundierte Entscheidungen in Fällen von morphologischen Veränderung, die in der MTL sehr wahrscheinlich ist. Diese Richtlinien können angewendet werden, um Forschung an strukturellen und / oder funktionellen MRT Untersuchungen der MTL, einschließlich volumetrische Analysen und Gehirn Anomalie-Erkennung, sowie Lokalisierung von Verfahren für die funktionelle oder anatomische und tractographic Analysen bei gesunden Gruppen. Dieses Protokoll kann auch verwendet werden, um die Segmentierung der MTL Strukturen für Patienten (z. B. Patienten mit Atrophie) zu informieren, wenn die großen anatomischen Orientierungspunkte werden relativ beibehalten. Tracing klinischen Themas 'Daten können zusätzliche Zeit und Mühe nehmen, abhängig von der Schwere der Atrophie und / oder anatomische Veränderungen.
Es ist wichtig, den Unterschied zwischen Kortex und Gyrus berücksichtigen bei der Festlegung der ROI. Anatomisch, bezieht sich hier sowohl Gyrus weißen Substanz und grauen Substanz, während Kortex bezieht sich auf Materie nur grau. Abhängig von der beabsichtigten Verwendung des ROI kann Segmentierungen weißen Substanz einschließen oder ausschließen.
Wir empfehlen die Rückverfolgung nacheinander durchgeführt werden, Unterbau Unterbau, eine Hemisphäre zu einem Zeitpunkt. Bestimmte Software-Pakete 34 sorgen für eine Verfolgung grenzt auf einer Scheibe skizziert auf nachfolgende Scheiben, ein Feature, das beschleunigt den Prozess eingefügt werden. Es ist immer ratsam, um die gegenüberliegende Hemisphäre Referenz nach Bedarf, um die für die Konsistenz zwischen den beiden Seiten (zB in anatomischen Landmarken Erkennung) zu überprüfen. Alternativ parallel Verfolgung der gleichen Strukturen innerhalb der beiden Halbkugels kann auch durchgeführt werden. Unabhängig davon, ob die Ablaufverfolgung ist sequentiell oder parallel, sobald der Prozess abgeschlossen ist, sollte die Tracer überprüfen Sie die End-Ergebnis und Anpassungen nach Bedarf, verweisen beide Hemisphären und Mehrfachebene Blick. Je nach Erfahrung des Tracers und der Auflösung der Bilddaten, manuelle Segmentierung des MTL für gesunden Probanden Daten können von 8 bis 10 h oder mehr im Falle eines Novizen Tracer zu 3-4 Stunden in die zu nehmen,,,, Bei einem erfahrenen ein.
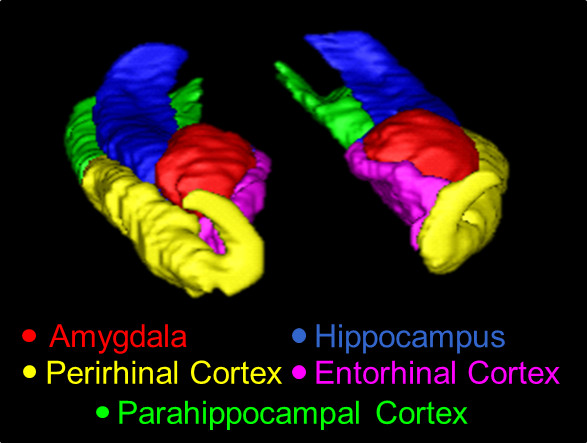
Abbildung 1. Ein 3D-Überblick über die MTL, verfolgt mit dem vorliegenden Protokoll. Strukturen hier dargestellt sind die AMY (rot), der HC (blau), die VR China (gelb), der Europäische Forschungsrat (pink) und der PHC (grün) .
Protocol
Representative Results
Discussion
Traditionell hat manuelle Segmentierung von vielen Forschern wurde als Goldstandard. Es wurde jedoch eine genaue Abgrenzung der einzelnen Strukturen durch die sehr variable Morphologie der MTL Strukturen erschwert und durch die in der Regel schwach MRI Kontraste dieser Strukturen gegen die umgebenden Nervengewebe und nicht-neuronalen Bereichen. In der Vergangenheit haben einige MTL-Strukturen gab es widersprüchliche Beschreibungen in der Literatur. In einigen Fällen von Segmentieren der PRC zum Beispiel der Sicherungs…
Declarações
The authors have nothing to disclose.
Acknowledgements
This research was supported by funds to FD. MM was supported by an IGERT Fellowship under National Science Foundation Grant No. 0903622. The authors wish to thank the Dolcos Lab members for assistance with data collection and preparation.
Materials
| ITK-SNAP | ITK-SNAP Team at University of Pennsylvania and University of Utah | ITK-SNAP v2.2 | |
| FSL | Functional Magnetic Resonance Imaging of the Brain (FMRIB) Analysis Group | FSL v4.1 | |
| 3T Siemens Trio MR Scanner | Siemens | 3T Trio |
Referências
- Amaral, D. G. Introduction: what is where in the medial temporal lobe. Hippocampus. 9, 1-6 (1999).
- Squire, L. R., Zola-Morgan, S. The medial temporal lobe memory system. Science. 253 (5026), 1380-1386 (1991).
- Eichenbaum, H., Otto, T., Cohen, N. J. The hippocampus: what does it do. Behavioral & Neural Biology. 57 (1), 2-36 (1992).
- Henke, K., Buck, A., Weber, B., Wieser, H. G. Human hippocampus establishes associations in memory. Hippocampus. 7 (3), 249-256 (1997).
- Tulving, E., Markowitsch, H. J. Episodic and declarative memory: role of the hippocampus. Hippocampus. 8 (3), 198-204 (1998).
- Dolcos, F., Iordan, A. D., Dolcos, S. Neural correlates of emotion–cognition interactions: a review of evidence from brain imaging investigations. Journal of Cognitive Psychology. 23 (6), 669-694 (2011).
- Davidson, R. J., Irwin, W. The functional neuroanatomy of emotion and affective style. Trends in Cognitive Sciences. 3 (1), 11-21 (1999).
- Lindquist, K. A., Wager, T. D., Kober, H., Bliss-Moreau, E., Barrett, L. F. The brain basis of emotion: a meta-analytic review. The Behavioral and Brain Sciences. 35 (3), 121-143 (2012).
- Phan, K. L., Wager, T., Taylor, S. F., Liberzon, I. Functional neuroanatomy of emotion: a meta-analysis of emotion activation studies in PET and fMRI. Neuroimage. 16 (2), 331-348 (2002).
- Wager, T. D., Phan, K. L., Liberzon, I., Taylor, S. F. Valence, gender, and lateralization of functional brain anatomy in emotion: a meta-analysis of findings from neuroimaging. Neuroimage. 19, 513-531 (2003).
- Zald, D. H. The human amygdala and the emotional evaluation of sensory stimuli. Brain Research Reviews. 41, 88-123 (2003).
- DeYoung, C. G., Hirsh, J. B., Shane, M. S., Papademetris, X., Rajeevan, N., Gray, J. R. Testing predictions from personality neuroscience: brain structure and the big five. Psychological Science. 21 (6), 820-828 (2010).
- Visser, P. J., Verhey, F. R., Hofman, P. A., Scheltens, P., Jolles, J. Medial temporal lobe atrophy predicts Alzheimer’s disease in patients with minor cognitive impairment. Journal of Neurology, Neurosurgery, & Psychiatry. 72 (4), 491-497 (2002).
- Ezekiel, F., et al. Comparisons between global and focal brain atrophy rates in normal aging and Alzheimer disease. Alzheimer Disease & Associated Disorders. 18 (4), 196-201 (2004).
- de Leon, M. J., et al. Imaging and CSF studies in the preclinical diagnosis of Alzheimer’s disease. Annals of the New York Academy of Sciences. 1097, 114-145 (2007).
- Boccardi, M., et al. Survey of protocols for the manual segmentation of the hippocampus: preparatory steps towards a joint EADC-ADNI harmonized protocol. Journal of Alzheimer’s Disease. 26, 61-75 (2011).
- Konrad, C., Ukas, T., Nebel, C., Arolt, V., Toga, A. W., Narr, K. L. Defining the human hippocampus in cerebral magnetic resonance images-an overview of current segmentation protocols. Neuroimage. 47 (4), 1185-1195 (2009).
- Hasboun, D., et al. MR determination of hippocampal volume: comparison of three methods. American Journal of Neuroradiology. 17 (6), 1091-1098 (1996).
- Pantel, J., et al. A new method for the in vivo volumetric measurement of the human hippocampus with high neuroanatomical accuracy. Hippocampus. 10, 752-758 (2000).
- Entis, J. J., Doerga, P., Barrett, L. F., Dickerson, B. C. A reliable protocol for the manual segmentation of the human amygdala and its subregions using ultra-high resolution MRI. Neuroimage. 60 (2), 1226-1235 (2012).
- Goncharova, I. I., Dickerson, B. C., Stoub, T. R., deToledo-Morrell, L. MRI of human entorhinal cortex: a reliable protocol for volumetric measurement. Neurobiology of Aging. 22, 737-745 (2001).
- Watson, C., et al. Anatomic basis of amygdaloid and hippocampal volume measurement by magnetic resonance imaging. Neurology. 42 (9), 1743-1750 (1992).
- Duvernoy, H. The human hippocampus: functional anatomy, vascularization, and serial sections with MRI. Third Edition. , (2005).
- Amaral, D. G., Witter, M. P. The three-dimensional organization of the hippocampal formation: a review of anatomical data. Neurociência. 31 (3), 571-591 (1989).
- Insausti, R., et al. MR volumetric analysis of the human entorhinal, perirhinal, and temporopolar cortices. American Journal of Neuroradiology. 19 (4), 659-671 (1998).
- Dolcos, F., LaBar, K. S., Cabeza, R. Interaction between the amygdala and the medial temporal lobe memory system predicts better memory for emotional events. Neuron. 42 (5), 855-863 (2004).
- Yushkevich, P. A., et al. User-guided 3D active contour segmentation of anatomical structures: significantly improved efficiency and reliability. Neuroimage. 31 (3), 1116-1128 (2006).
- Bonilha, L., Kobayashi, E., Cendes, F., Li, M. L. Protocol for volumetric segmentation of medial temporal structures using high-resolution 3-D magnetic resonance imaging. Human Brain Mapping. 22 (2), 145-154 (2004).
- Bronen, R. A., Cheung, G. Relationship of hippocampus and amygdala to coronal MRI landmarks. Magnetic Resonance Imaging. 9 (3), 449-457 (1991).
- Pruessner, J. C., et al. Volumetry of temporopolar, perirhinal, entorhinal and parahippocampal cortex from high-resolution MR images: considering the variability of the collateral sulcus. Cerebral Cortex. 12 (12), 1342-1353 (2002).
- Pruessner, J. C., et al. Volumetry of hippocampus and amygdala with high-resolution MRI and three-dimensional analysis software: minimizing the discrepancies between laboratories. Cerebral Cortex. 10 (4), 433-442 (2000).
- Duvernoy, H. . The human brain: surface, three-dimensional sectional anatomy with MRI, and blood supply. Second Edition. , (1999).
- Amaral, D. G., Lavenex, P., et al. . in The hippocampus book. Hippocampal neuroanatomy. , (2006).
- Blaizot, X., et al. The human parahippocampal region: I. temporal pole cytoarchitectonic and MRI correlation. Cerebral Cortex. 20 (9), 2198-2212 (2010).
- Ding, S. -. L., Van Hoesen, G. W. Borders, extent, and topography of human perirhinal cortex as revealed using multiple modern neuroanatomical and pathological markers. Human Brain Mapping. 31 (9), 1359-1379 (2010).
- Ding, S. -. L., Van Hoesen, G. W., Cassell, M. D., Poremba, A. Parcellation of human temporal polar cortex: a combined analysis of multiple cytoarchitectonic, chemoarchitectonic, and pathological markers. The Journal of Comparative Neurology. 514 (6), 595-623 (2009).
- Frankó, E., Insausti, A. M., Artacho-Pérula, E., Insausti, R., Chavoix, C. Identification of the human medial temporal lobe regions on magnetic resonance images. Human Brain Mapping. 35 (1), 248-256 (2014).
- Lehmann, M., et al. Atrophy patterns in Alzheimer’s disease and semantic dementia: a comparison of FreeSurfer and manual volumetric measurements. Neuroimage. 49 (3), 2264-2274 (2010).
- Winterburn, J. L., et al. A novel in vivo atlas of human hippocampal subfields using high-resolution 3T magnetic resonance imaging. Neuroimage. 74, 254-265 (2013).
- Malykhin, N. V., Bouchard, T. P., Ogilvie, C. J., Coupland, N. J., Seres, P., Camicioli, R. Three-dimensional volumetric analysis and reconstruction of amygdala and hippocampal head, body and tail. Psychiatry research. Neuroimaging. 155 (2), 155-165 (2007).
- Patenaude, B., Smith, S. M., Kennedy, D. N., Jenkinson, M. A Bayesian model of shape and appearance for subcortical brain segmentation. Neuroimage. 56 (3), 907-922 (2011).
- Smith, S. M., et al. Advances in functional and structural MR image analysis and implementation as FSL. Neuroimage. 23, (2004).
- Woolrich, M. W., et al. Bayesian analysis of neuroimaging data in FSL. Neuroimage. 45, (2009).
- Smith, S. M. Fast robust automated brain extraction. Human Brain Mapping. 17 (3), 143-155 (2002).
- Morey, R. A., et al. A comparison of automated segmentation and manual tracing for quantifying hippocampal and amygdala volumes. Neuroimage. 45 (3), 855-866 (2009).
- Baldassano, C., Beck, D. M., Fei-Fei, L. Differential connectivity within the parahippocampal place area. Neuroimage. 75, 228-237 (2013).
- Shrout, P. E., Fleiss, J. L. Intraclass correlations: uses in assessing rater reliability. Psychological Bulletin. 86 (2), 420-428 (1979).
- Bland, J. M., Altman, D. G. A note on the use of the intraclass correlation coefficient in the evaluation of agreement between two methods of measurement. Computers in Biology and Medicine. 20, 337-340 (1990).
- Yushkevich, P. A., et al. A high-resolution computational atlas of the human hippocampus from postmortem magnetic resonance imaging at 9.4 T. Neuroimage. 44 (2), 385-398 (2009).

