Studio del reticolo endoplasmatico e mitocondri interazioni<em> In Situ</em> Proximity Assay legatura in cellule fissate
Summary
Qui, descriviamo una procedura per visualizzare e quantificare con alta sensibilità le interazioni endogeni tra il reticolo endoplasmatico e mitocondri nelle cellule fissate. Il protocollo è dotato di un ottimizzato de test legatura situ prossimità targeting inositolo 1,4,5-trifosfato recettore / glucosio-regolata proteina 75 / D complesso canale anionico / cyclophilin voltaggio-dipendente all'interfaccia membrana mitocondriale associata.
Abstract
Structural interactions between the endoplasmic reticular (ER) and mitochondrial membranes, in domains known as mitochondria-associated membranes (MAM), are crucial hubs for cellular signaling and cell fate. Particularly, these inter-organelle contact sites allow the transfer of calcium from the ER to mitochondria through the voltage-dependent anion channel (VDAC)/glucose-regulated protein 75 (GRP75)/inositol 1,4,5-triphosphate receptor (IP3R) calcium channeling complex. While this subcellular compartment is under intense investigation in both physiological and pathological conditions, no simple and sensitive method exists to quantify the endogenous amount of ER-mitochondria contact in cells. Similarly, MAMs are highly dynamic structures, and there is no suitable approach to follow modifications of ER-mitochondria interactions without protein overexpression. Here, we report an optimized protocol based on the use of an in situ proximity ligation assay to visualize and quantify endogenous ER-mitochondria interactions in fixed cells by using the close proximity between proteins of the outer mitochondrial membrane (VDAC1) and of the ER membrane (IP3R1) at the MAM interface. Similar in situ proximity ligation experiments can also be performed with the GRP75/IP3R1 and cyclophilin D/IP3R1 pairs of antibodies. This assay provides several advantages over other imaging procedures, as it is highly specific, sensitive, and suitable to multiple-condition testing. Therefore, the use of this in situ proximity ligation assay should be helpful to better understand the physiological regulations of ER-mitochondria interactions, as well as their role in pathological contexts.
Introduction
Mitocondri e reticolo endoplasmatico (ER) non sono organelli indipendenti nella cellula, ma interagiscono strutturalmente e funzionalmente a siti di contatto definite membrane reticolo endoplasmatico mitocondri-associato (MAM). Infatti, DAM corrispondono alle regioni in cui le membrane del ER e mitocondri sono strettamente apposto, permettendo interazioni tra proteine da entrambi i lati. Ciò nonostante, le membrane di questi organelli non si fondono in queste regioni, in modo da mantenere le loro entità separate. I DAM svolgono un ruolo cruciale nel calcio (Ca 2+) e il trasferimento dei fosfolipidi da ER a mitocondri, impattando il metabolismo energetico e la sopravvivenza delle cellule 1-3.
L'associazione tra ER e mitocondri è stato visualizzato nel 1970 con microscopia elettronica. Da allora, la trasmissione microscopia elettronica 4,5, elettroni tomografia 6,7 o immuno-localizzazione di ER e specifici mitocondri fluoroforos / proteine fluorescenti 8 sono stati classicamente utilizzati per studiare le interazioni ER-mitocondri. Un altro strumento utile per l'analisi di MAM si basa sull'uso di frazionamento subcellulare. Esso permette di isolare frazioni MAM mediante ultracentrifugazione differenziale accoppiato ad un gradiente Percoll 9. Tuttavia, il prodotto finale contiene frazioni arricchite MAM, anziché frazioni pure. Complessivamente, queste strategie non sono particolarmente sensibili e / o quantitativo, e non sono facilmente suscettibili di grande screening. In alternativa, utilizzando approcci genetici fluorescenti linker inter-organelli droga-inducibile sono emerse, ma non consentire l'analisi delle interazioni organelli ai livelli di espressione delle proteine endogene 10.
Sulla base di scoperta di Szabadkai del complesso IP3R / GRP75 / VDAC al MAM 11, abbiamo sviluppato un metodo quantitativo per analizzare le interazioni ER-mitocondri. Abbiamo usato il ligati in situ di prossimitàil test di rilevare e quantificare le interazioni tra VDAC1 e IP3R1, due proteine organello-superficiali coinvolti nella -channeling complesso Ca 2+ all'interfaccia MAM in cellule fisse 12. Brevemente, abbiamo sondato VDAC1 alla membrana esterna mitocondriale (topo anti-VDAC1 anticorpo primario) e IP3R1 sulla membrana ER (coniglio anti-IP3R1 anticorpo primario) (figura 1, pannello A). Quindi, secondo il test, abbiamo aggiunto sia anti-topo e IgG anti-coniglio (sonde saggio mouse e vicinanza coniglio legatura), che sono coniugato a estensioni oligonucleotidi complementari. Se le due proteine sono mirati ad una distanza inferiore a 40 nm, gli oligonucleotidi possono ibridare con oligonucleotidi connettore successivamente aggiunti per consentire la formazione di un modello circolare DNA (figura 1, pannello B). Questa molecola di DNA circolare è legatura ed amplificato, creando un prodotto singolo filamento DNA covalentemente attaccata ad una delle sonde di prossimità (figura 1, pannello c) </strong>. Poiché la distanza tra ER e mitocondri all'interfaccia MAM varia da 10 nm a 25 nm 6, vicinanza legatura ed amplificazione può essere fatto, portando a successiva rivelazione causa della ibridazione della Texas oligonucleotidi rossi marcati sonde (Figura 1, pannello d ). Ogni punto rappresenta fluorescente interazioni tra VDAC1 / IP3R1, permettendo così la quantificazione delle interazioni in situ ER-mitocondri nelle singole celle.
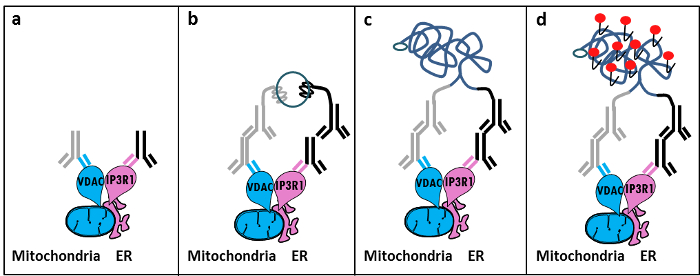
Figura 1: Schema Illustrazione del rilevamento delle interazioni reticolo endoplasmatico-mitocondri in situ prossimità legatura Assay. a) Un anticorpo mouse principale diretto contro VDAC1 e un anticorpo di coniglio diretto contro primaria IP3R1 possono legarsi ai loro epitopi in prossimità all'interfaccia MAM, b) aggiunta di una coppia di sonde legatura di prossimitàdiretto contro mouse e IgG di coniglio. Queste sonde hanno attaccato filamenti di DNA che possono formare modelli per la legatura di oligonucleotidi connettore. c) Il filamento di DNA circolare formata dopo la legatura può essere amplificato e d) visualizzati mediante microscopia a fluorescenza dot utilizzando oligonucleotidi Texas red-marcato. Clicca qui per vedere una versione più grande di questa figura.
Simile de esperimenti di analisi in situ prossimità legatura possono essere eseguite con la coppia GRP75 / IP3R1 di anticorpi, così come cyclophilin D (CypD) / anticorpi IP3R1, se si considera che CypD ha dimostrato di interagire con il complesso IP3R / GRP75 / VDAC all'interfaccia MAM 12-14.
Protocol
Representative Results
Discussion
Collectively, our studies indicate that the in situ proximity ligation assay is truly a relevant strategy to follow and quantify endogenous ER-mitochondria interactions in fixed cells, without the need for using organelle-specific fluorophores or fluorescent proteins. The specific use of VDAC1/IP3R1 antibodies has been adapted to study ER-mitochondria interactions in HuH7 cells. However, alternative isoforms of VDAC and IP3R may be used, depending on the cell type. In this case, antibodies need to be validated b…
Declarações
The authors have nothing to disclose.
Acknowledgements
Ringraziamo tutte le persone nel nostro laboratorio che hanno contribuito a ottimizzare e validare il protocollo. Questo lavoro è stato sostenuto da INSERM e l'agenzia nazionale di ricerca (ANR-09-JCJC-0116 E ANR-11-BSV1-033-02). ET è stato sostenuto durante il suo dottorato di ricerca da un assegno di ricerca dal Ministero francese dell'istruzione superiore e della ricerca.
Materials
| Formaldehyde | Sigma | F-8775 | |
| Glycine | Sigma | G-8898 | |
| Triton | Sigma | T8532 | |
| 35mm Glass bottom culture dishes | MatTeK corporation | P35G-0-14-C | |
| Blocking solution | Sigma | DUO-92004 or DUO-92002 | provided in the Duolink PLA probes, Sigma |
| VDAC1 antibody | Abcam | ab14734 | |
| IP3R1-H80 antibody | Santa Cruz | sc28614 | |
| CypD antibody | Abcam | ab110324 | |
| Grp75 antibody | Santa Cruz | sc13967 | |
| TBS 10X | euromedex | ET220 | Dilute to obtain 1X |
| Tween 100X | euromedex | 2001-B | dilute in TBS to obtain 0,01% |
| PLA Probes Mouse MINUS | Sigma | DUO-92004 | Duolink, Sigma |
| PLA Probes Rabbit PLUS | Sigma | DUO-92002 | Duolink, Sigma |
| Duolink detection reagents red | Sigma | DUO-92008 | Duolink, Sigma |
| Ligation solution | Sigma | DUO-92008 | Part of the Duolink detection reagents red, Sigma |
| Ligase | Sigma | DUO-92008 | Part of the Duolink detection reagents red, Sigma |
| Amplification solution | Sigma | DUO-92008 | Part of the Duolink detection reagents red, Sigma |
| Polymerase | Sigma | DUO-92008 | Part of the Duolink detection reagents red, Sigma |
| Duolink Mounting Medium | Sigma | DUO80102 | Duolink, Sigma |
| Softwares: | |||
| Blob-finder software | BlobFinder is a freely distributed software that can perform calculations on cells from fluorescence microscopy images. This software can be downloaded for free from The Centre for Image Analysis at Uppsala University who have developed the software and the work was supported by the EU FP6 Project ENLIGHT and Olink Bioscience. http://www.cb.uu.se/~amin/BlobFinder/index_files/Page430.htm | ||
| ImageJ software | Can be downloaded for free from: http://rsb.info.nih.gov/ij/download.html |
Referências
- Bravo-Sagua, R., et al. Organelle communication: signaling crossroads between homeostasis and disease. The international journal of biochemistry & cell biology. 50, 55-59 (2014).
- Giorgi, C., et al. Mitochondria-associated membranes: composition, molecular mechanisms, and physiopathological implications. Antioxidants & redox signaling. 22, 995-1019 (2015).
- Phillips, M. J., Voeltz, G. K. Structure and function of ER membrane contact sites with other organelles. Nature reviews. Molecular cell biology. 17, 69-82 (2016).
- Cosson, P., et al. The RTM resistance to potyviruses in Arabidopsis thaliana: natural variation of the RTM genes and evidence for the implication of additional genes. PLoS One. 7, 39169 (2012).
- Mannella, C. A. Structure and dynamics of the mitochondrial inner membrane cristae. Biochim Biophys Acta. 1763, 542-548 (2006).
- Csordas, G., et al. Structural and functional features and significance of the physical linkage between ER and mitochondria. The Journal of cell biology. 174, 915-921 (2006).
- Mannella, C. A., Buttle, K., Rath, B. K., Marko, M. Electron microscopic tomography of rat-liver mitochondria and their interaction with the endoplasmic reticulum. Biofactors. 8, 225-228 (1998).
- Rizzuto, R., et al. Close contacts with the endoplasmic reticulum as determinants of mitochondrial Ca2+ responses. Science. 280, 1763-1766 (1998).
- Wieckowski, M. R., Giorgi, C., Lebiedzinska, M., Duszynski, J., Pinton, P. Isolation of mitochondria-associated membranes and mitochondria from animal tissues and cells. Nat Protoc. 4, 1582-1590 (2009).
- Csordas, G., et al. Imaging interorganelle contacts and local calcium dynamics at the ER-mitochondrial interface. Mol Cell. 39, 121-132 (2010).
- Szabadkai, G., et al. Chaperone-mediated coupling of endoplasmic reticulum and mitochondrial Ca2+ channels. J Cell Biol. 175, 901-911 (2006).
- Tubbs, E., et al. Mitochondria-associated endoplasmic reticulum membrane (MAM) integrity is required for insulin signaling and is implicated in hepatic insulin resistance. Diabetes. 63, 3279-3294 (2014).
- Paillard, M., et al. Depressing Mitochondria-Reticulum Interactions Protects Cardiomyocytes From Lethal Hypoxia-Reoxygenation Injury. Circulation. 128, 1555-1565 (2013).
- Rieusset, J., et al. Disruption of calcium transfer from ER to mitochondria links alterations of mitochondria-associated ER membrane integrity to hepatic insulin resistance. Diabetologia. 59, 614-623 (2016).
- Allalou, A., Wahlby, C. BlobFinder, a tool for fluorescence microscopy image cytometry. Computer methods and programs in biomedicine. 94, 58-65 (2009).
- Theurey, P., et al. Mitochondria-associated endoplasmic reticulum membranes allow adaptation of mitochondrial metabolism to glucose availability in the liver. Journal of molecular cell biology. , (2016).
- de Brito, O. M., Scorrano, L. Mitofusin 2 tethers endoplasmic reticulum to mitochondria. Nature. 456, 605-610 (2008).
- Soderberg, O., et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nature methods. 3, 995-1000 (2006).
- De Pinto, V., Messina, A., Lane, D. J., Lawen, A. Voltage-dependent anion-selective channel (VDAC) in the plasma membrane. FEBS letters. 584, 1793-1799 (2010).
- Kaul, S. C., Taira, K., Pereira-Smith, O. M., Wadhwa, R. Mortalin: present and prospective. Experimental gerontology. 37, 1157-1164 (2002).

