प्रोग्रामेबल इन विट्रो ट्रांसक्रिप्शन और मॉलिक्यूलर कंप्यूटेशन के लिए डीएनए-टेथर्ड आरएनए पॉलीमरेज
Summary
हम इन विट्रो ट्रांसक्रिप्शन प्रतिक्रियाओं को विनियमित करने के लिए एक उपन्यास डीएनए-सीमित T7 आरएनए पॉलीमरेज की इंजीनियरिंग का वर्णन करते हैं। हम प्रोटीन संश्लेषण और लक्षण वर्णन के लिए कदमों पर चर्चा करते हैं, प्रूफ-ऑफ-कॉन्सेप्ट ट्रांसक्रिप्शनल नियमन को मान्य करते हैं, और आणविक कंप्यूटिंग, निदान और आणविक सूचना प्रसंस्करण में इसके अनुप्रयोगों पर चर्चा करते हैं।
Abstract
डीएनए नैनो प्रोग्राम करने योग्य आत्म-नाभिक एसिड की असेंबली को विभिन्न अनुप्रयोगों के लिए उपयोगकर्ता-निर्धारित आकार और गतिशीलता में सक्षम बनाता है। यह काम दर्शाता है कि डीएनए नैनो से अवधारणाओं को फेज-व्युत्पन्न T7 आरएनए पॉलीमरेज (आरएनएपी) की एंजाइमेटिक गतिविधि को प्रोग्राम करने और स्केलेबल सिंथेटिक जीन नियामक नेटवर्क बनाने के लिए इस्तेमाल किया जा सकता है। सबसे पहले, एक ओलिगोन्यूक्लियोटाइड-टेथेर टी 7 आरएनएपी को एन-मरणासन्न स्नैप-टैग आरएनएपी की अभिव्यक्ति और बाद में एक बेंजिल्गुएनिन (बीजी)-संशोधित ओलिगोन्यूक्लियोटाइड के साथ स्नैप-टैग के रासायनिक युग्मन की अभिव्यक्ति के माध्यम से इंजीनियर किया जाता है। इसके बाद, न्यूक्लिक-एसिड स्ट्रैंड विस्थापन का उपयोग मांग पर पॉलीमरेज ट्रांसक्रिप्शन को प्रोग्राम करने के लिए किया जाता है। इसके अलावा, सहायक न्यूक्लिक एसिड असेंबली का उपयोग डीएनए-प्रोग्राम किए गए T7 आरएनएपी के बीच बातचीत को अपने डीएनए टेम्पलेट्स के साथ विनियमित करने के लिए “कृत्रिम प्रतिलेखन कारक” के रूप में किया जा सकता है। यह इन विट्रो ट्रांसक्रिप्शन नियामक तंत्र विभिन्न प्रकार के सर्किट व्यवहार जैसे डिजिटल तर्क, प्रतिक्रिया, कैस्केडिंग और मल्टीप्लेक्सिंग को लागू कर सकता है। इस जीन नियामक वास्तुकला की रचना डिजाइन अमूर्तता, मानकीकरण और स्केलिंग की सुविधा प्रदान करती है। इन सुविधाओं से बायो सेंसिंग, डिजीज डिटेक्शन और डाटा स्टोरेज जैसे एप्लीकेशंस के लिए इन विट्रो जेनेटिक डिवाइसेज की तेजी से प्रोटोटाइप हो सकेगी ।
Introduction
डीएनए कंप्यूटिंग गणना के माध्यम के रूप में डिजाइन ओलिगोन्यूक्लियोटाइड्स के एक सेट का उपयोग करता है। इन ओलिगोन्यूक्लियोटाइड को उपयोगकर्ता-निर्दिष्ट तर्क के अनुसार गतिशील रूप से इकट्ठा करने और विशिष्ट न्यूक्लिक-एसिड इनपुट का जवाब देने के लिए दृश्यों के साथ प्रोग्राम किया जाता है। प्रूफ-ऑफ-कॉन्सेप्ट अध्ययनों में, गणना के उत्पादन में आम तौर पर फ्लोरोसेंटली लेबल वाले ओलिगोन्यूक्लियोटाइड का एक सेट होता है जिसे जेल इलेक्ट्रोफोरेसिस या फ्लोरेसेंस प्लेट रीडर्स के माध्यम से पता लगाया जा सकता है। पिछले 30 वर्षों में, तेजी से जटिल डीएनए कंप्यूटेशनल सर्किटरी का प्रदर्शन किया गया है, जैसे विभिन्न डिजिटल तर्क झरना, रासायनिक प्रतिक्रिया नेटवर्क, और तंत्रिका नेटवर्क1,2,3। इन डीएनए सर्किट की तैयारी के साथ सहायता करने के लिए, गणितीय मॉडल सिंथेटिक जीन सर्किट4, 5की कार्यक्षमता की भविष्यवाणी करने के लिए इस्तेमाल किया गया है, और कम्प्यूटेशनल उपकरण ऑर्थोगोनल डीएनए अनुक्रम डिजाइन6,7,8,9,10 के लिए विकसित किया गया है . सिलिकॉन आधारित कंप्यूटरों की तुलना में, डीएनए कंप्यूटर के फायदों में बायोमॉलिक्यूल्स के साथ सीधे इंटरफेस करने की उनकी क्षमता शामिल है, बिजली की आपूर्ति के अभाव में समाधान में काम करते हैं, साथ ही उनकी समग्र कॉम्पैक्टनेस और स्थिरता भी शामिल है। अगली पीढ़ी के अनुक्रमण के आगमन के साथ, डीएनए कंप्यूटर संश्लेषण की लागत पिछले दो दशकों से मूर के कानून11की तुलना में तेजी से एक दर पर कम हो रही है । ऐसे डीएनए आधारित कंप्यूटरों के अनुप्रयोग अब उभरने लगे हैं, जैसे रोग निदान12,13,आणविक जैव भौतिकी 14 को शक्ति देने के लिए और डेटा भंडारण प्लेटफार्म15के रूप में ।
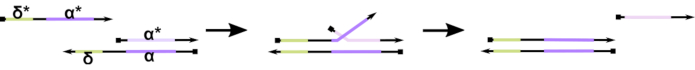
चित्रा 1:टोहोल्ड-मध्यस्थता डीएनए स्ट्रैंड विस्थापन का तंत्र। δ, टोहोल्ड, आंशिक डुप्लेक्स पर एक स्वतंत्र, अनबाउंड अनुक्रम है। जब एक पूरक डोमेन (δ *) एक दूसरे कतरा पर शुरू किया जाता है, मुक्त δ डोमेन संकरण के लिए एक toehold के रूप में कार्य करता है, कतरा के बाकी के लिए अनुमति देता है (ɑ *) धीरे से एक ज़िपिंग के माध्यम से अपने प्रतियोगी विस्थापित/ जैसे-जैसे δ की लंबाई बढ़ती है, आगे की प्रतिक्रिया के लिए एजीजी कम हो जाता है, और विस्थापन अधिक आसानी से होता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
आज तक, अधिकांश डीएनए कंप्यूटर गतिशील डीएनए नैनो के क्षेत्र में एक अच्छी तरह से स्थापित आकृति का उपयोग करते हैं जिसे टोहोल्ड-मध्यस्थता डीएनए स्ट्रैंड विस्थापन (टीएमडीएसडी, चित्रा 1) 16के रूप में जानाजाताहै। इस आकृति में आंशिक रूप से डबल-फंसे डीएनए (डीएसडीएनए) डुप्लेक्स होते हैं जो छोटे “टोहोल्ड” ओवरहांग (यानी, 7-से 10 न्यूक्लियोटिड्स (एनटी)) प्रदर्शित करते हैं। न्यूक्लिक एसिड “इनपुट” किस्में टोहोल्ड के माध्यम से आंशिक डुप्लेक्स के साथ बातचीत कर सकती हैं। यह आंशिक डुप्लेक्स से एक किस्में के विस्थापन की ओर जाता है, और यह मुक्त कतरा फिर डाउनस्ट्रीम आंशिक डुप्लेक्स के लिए इनपुट के रूप में काम कर सकता है। इस प्रकार, टीएमडीएसडी सिग्नल कैस्केडिंग और सूचना प्रसंस्करण को सक्षम बनाता है। सिद्धांत रूप में, ऑर्थोगोनल टीएमडीएसडी रूपांकनों समाधान में स्वतंत्र रूप से काम कर सकते हैं, समानांतर सूचना प्रसंस्करण को सक्षम करते हैं। टीएमडीएसडी प्रतिक्रिया पर कई भिन्नताएं रही हैं, जैसे टोहोल्ड-मध्यस्थता डीएनए स्ट्रैंड एक्सचेंज(टीएमडीएसई) 17,डबल-लॉन्ग डोमेन18,अनुक्रम-बेमेल टोहोल्ड19,और “हैंडहोल्ड”-मध्यस्थता वाले फंसे विस्थापन20के साथ “लीकरहित” टोहोल्ड। ये अभिनव डिजाइन सिद्धांत डीएनए कंप्यूटिंग प्रदर्शन में सुधार के लिए अधिक पतले देखते टीएमडीडी एनर्जेस और गतिशीलता की अनुमति देते हैं।
ट्रांसक्रिप्शनल जीन सर्किट जैसे सिंथेटिक जीन सर्किट भी गणना21, 22,23में सक्षम हैं। इन सर्किटों को प्रोटीन ट्रांसक्रिप्शन कारकों द्वारा विनियमित किया जाता है, जो विशिष्ट नियामक डीएनए तत्वों के लिए बाध्यकारी द्वारा जीन के प्रतिलेखन को सक्रिय या दबाते हैं। डीएनए आधारित सर्किट की तुलना में, ट्रांसक्रिप्शनल सर्किट के कई फायदे हैं। सबसे पहले, एंजाइमेटिक ट्रांसक्रिप्शन में मौजूदा उत्प्रेरक डीएनए सर्किट की तुलना में बहुत अधिक टर्नओवर दर है, इस प्रकार इनपुट की एक प्रति प्रति आउटपुट की अधिक प्रतियां पैदा होती हैं और सिग्नल प्रवर्धन का अधिक कुशल साधन प्रदान करती हैं। इसके अलावा, ट्रांसक्रिप्शनल सर्किट विभिन्न कार्यात्मक अणुओं का उत्पादन कर सकते हैं, जैसे कि एप्टामर्स या मैसेंजर आरएनए (एमआरएनए) चिकित्सीय प्रोटीन के लिए एन्कोडिंग, कंप्यूटेशन आउटपुट के रूप में, जिसका विभिन्न अनुप्रयोगों के लिए शोषण किया जा सकता है। हालांकि, वर्तमान ट्रांसक्रिप्शनल सर्किट की एक प्रमुख सीमा उनकी स्केलेबिलिटी की कमी है। इसका कारण यह है कि आर्थोगोनल प्रोटीन आधारित प्रतिलेखन कारकों का एक बहुत ही सीमित सेट है, और नए प्रोटीन ट्रांसक्रिप्शन कारकों का डी नोवो डिजाइन तकनीकी रूप से चुनौतीपूर्ण और समय लेने वाला रहता है।
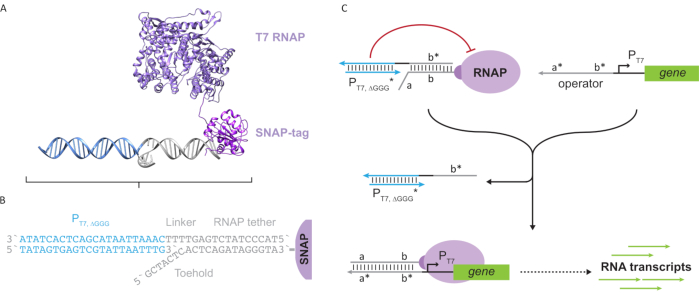
चित्रा 2:अमूर्तता और “तार” और “पिंजरे” बहुलक परिसर के तंत्र । (ए और बी)एक ओलिगोन्यूक्लियोटाइड तार को स्नैप-टैग प्रतिक्रिया के माध्यम से T7 बहुलक के लिए लेबल किया गया है । एक टेथर-पूरक ओवरहांग के साथ “अशुद्ध” T7 प्रमोटर से मिलकर एक पिंजरे यह तार और ब्लॉक प्रतिलेखन गतिविधि को संकरित करने के लिए अनुमति देता है । (ग)जब ऑपरेटर(ए * बी *)मौजूद होता है, तो यह ओलिगोन्यूक्लियोटाइड टेथर(एबी)पर टोहोल्ड से बांधता है और पिंजरे के बी * क्षेत्र को विस्थापित करता है, जिससे प्रतिलेखन होता है । इस आंकड़े को चाउ और शिह27से संशोधित किया गया है । संक्षिप्त: आरएनएपी = आरएनए पॉलीमरेज। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
यह पेपर आणविक कंप्यूटिंग के लिए एक उपन्यास बिल्डिंग ब्लॉक का परिचय देता है जो डीएनए-आधारित सर्किट की स्केलेबिलिटी के साथ ट्रांसक्रिप्शनल सर्किट की कार्यक्षमताओं को जोड़ती है। यह बिल्डिंग ब्लॉक एक T7 RNAP सहसंयोजक रूप से एक फंसे डीएनए तार(चित्रा 2A)के साथ जुड़ा हुआ है । इस डीएनए-सीमित T7 आरएनएपी को संश्लेषित करने के लिए, पॉलीमरेज को एन-टर्मिनल स्नैप-टैग24 से जोड़ा गया था और एस्चेरिचिया कोलाईमें फिर से व्यक्त किया गया था । स्नैप-टैग को बीजी सब्सट्रेट के साथ कार्यात्मक एक ओलिगोन्यूक्लियोटाइड के साथ प्रतिक्रिया व्यक्त की गई थी। ओलिगोन्यूक्लियोटाइड टेथर डीएनए संकरण के माध्यम से पॉलीमरेज के करीब निकटता में आणविक मेहमानों की स्थिति की अनुमति देता है। ऐसा ही एक अतिथि एक प्रतिस्पर्धी ट्रांसक्रिप्शनल अवरोधक था जिसे “पिंजरे” के रूप में जाना जाता है, जिसमें एक “अशुद्ध” टी 7 प्रमोटर डीएनए डुप्लेक्स होता है जिसमें कोई जीन डाउनस्ट्रीम(चित्रा 2B) नहीं होता है। जब अपने ओलिगोन्यूक्लियोटाइड तार के माध्यम से आरएनएपी के लिए बाध्य, पिंजरे आरएनएपी बाध्यकारी के लिए अन्य डीएनए टेम्पलेट्स को मात देकर पॉलीमरेज गतिविधि करते हैं, आरएनएपी को “ऑफ” राज्य(चित्रा 2C)में प्रदान करते हैं।
एक “पर” राज्य के लिए बहुलक को सक्रिय करने के लिए, जीन के T7 प्रमोटर के ऊपर एकल फंसे “ऑपरेटर” डोमेन के साथ T7 डीएनए टेम्पलेट्स डिजाइन किए गए थे । ऑपरेटर डोमेन (यानी, डोमेन ए * बी * फिगर 2सी)को टीएमडीएसडी के माध्यम से आरएनएपी से पिंजरे को विस्थापित करने और आरएनएपी समीपस्थ को जीन के T7 प्रमोटर को स्थान देने के लिए डिज़ाइन किया जा सकता है, इस प्रकार प्रतिलेखन शुरू किया जा सकता है। वैकल्पिक रूप से, डीएनए टेम्पलेट्स भी डिजाइन किए गए थे जहां ऑपरेटर अनुक्रम सहायक न्यूक्लिक-एसिड किस्में के पूरक थे जिन्हें “कृत्रिम प्रतिलेखन कारक” (यानी, चित्र 3 ए में टीएफए और टीएफबी किस्में) के रूप में जाना जाता है। जब दोनों किस्में प्रतिक्रिया में पेश की जाती हैं, तो वे ऑपरेटर साइट पर इकट्ठा होंगे, एक नया छद्म-समीपस्थ डोमेन ए * बी *बना एंगे। यह डोमेन तब ट्रांसक्रिप्शन(चित्रा 3B)शुरू करने के लिए टीएमडीएसडी के माध्यम से पिंजरे को विस्थापित कर सकता है। इन किस्में या तो बहिर्जनात्मक या उत्पादित आपूर्ति की जा सकती है।
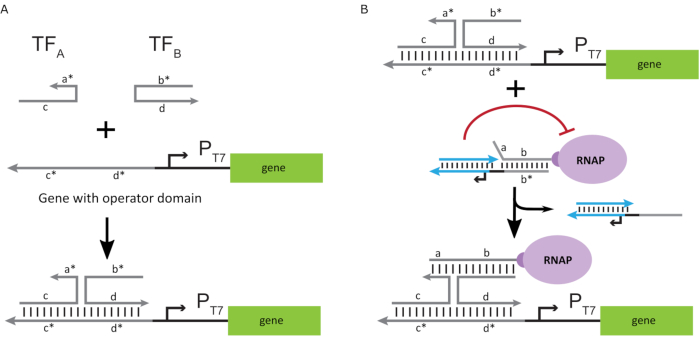
चित्र 3:तीन-घटक स्विच एक्टिवेटर के माध्यम से पॉलीमरेज गतिविधि की चयनात्मक प्रोग्रामिंग। (क)जब प्रतिलेखन कारक (टीएफए और टीएफबी)मौजूद होते हैं, तो वे प्रमोटर के अपस्ट्रीम ऑपरेटर डोमेन को बांधते हैं, जो टोहोल्ड मध्यस्थता डीएनए विस्थापन के माध्यम से पिंजरे को विस्थापित करने में सक्षम एक छद्म एकल-फंसे अनुक्रम(ए * बी *)बनाते हैं । (ख)यह ए * बी * डोमेन ट्रांसक्रिप्शन शुरू करने के लिए टीएमडीएसडी के माध्यम से पिंजरे को विस्थापित कर सकता है । इस आंकड़े को चाउ और शिह27से संशोधित किया गया है । संक्षिप्त रूप: TF = प्रतिलेखन कारक; आरएनएपी = आरएनए पॉलीमरेज; TMDSD = toehold-मध्यस्थता डीएनए कतरा विस्थापन । कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
इन विट्रो ट्रांसक्रिप्शनल विनियमन के लिए न्यूक्लिक एसिड-आधारित ट्रांसक्रिप्शन कारकों का उपयोग डिजिटल तर्क, प्रतिक्रिया और सिग्नल कैस्केडिंग जैसे परिष्कृत सर्किट व्यवहारों के स्केलेबल कार्यान्वयन की अनुमति देता है। उदाहरण के लिए, कोई भी न्यूक्लिक एसिड दृश्यों को डिजाइन करके तर्क गेट कैस्केड का निर्माण कर सकता है जैसे कि अपस्ट्रीम जीन से ट्रांसक्रिप्ट एक डाउनस्ट्रीम जीन को सक्रिय करते हैं। इस प्रस्तावित प्रौद्योगिकी द्वारा सक्षम कैस्केडिंग और मल्टीप्लेक्सिंग का शोषण करने वाला एक अनुप्रयोग पोर्टेबल निदान और आणविक डेटा प्रसंस्करण के लिए अधिक परिष्कृत आणविक कंप्यूटिंग सर्किटरी का विकास है। इसके अलावा, आणविक कंप्यूटिंग और डी नोवो आरएनए संश्लेषण क्षमताओं को एकीकृत करने से नए अनुप्रयोगों को सक्षम किया जा सकता है। उदाहरण के लिए, एक आणविक सर्किट को उपयोगकर्ता-परिभाषित आरएनए के संयोजन का पता लगाने के लिए डिज़ाइन किया जा सकता है और चिकित्सीय आरएनए या एमआरएएनए एन्कोडिंग कार्यात्मक पेप्टाइड्स या प्रोटीन को पॉइंट-ऑफ-केयर चिकित्सा अनुप्रयोगों के लिए आउटपुट कर सकते हैं।
Protocol
Representative Results
Discussion
यह अध्ययन टी 7 आरएनए पॉलीमरेज की गतिविधि को नियंत्रित करने के लिए एक डीएनए नैनो-प्रेरित दृष्टिकोण को दर्शाता है, जो एक बीजी-कार्यात्मक ओलिगोन्यूक्लियोटाइड के साथ एन-टर्मिनल स्नैप-टैग किए गए रिकॉम्बिन?…
Declarações
The authors have nothing to disclose.
Acknowledgements
L.Y.T.C अनुसंधान कोष में नई सीमाओं से उदार समर्थन स्वीकार-अंवेषण (NFRF-E), कनाडा के प्राकृतिक विज्ञान और इंजीनियरिंग अनुसंधान परिषद (NSERC) डिस्कवरी ग्रांट, और डिजाइन पहल है, जो कनाडा के पहले अनुसंधान उत्कृष्टता कोष (CFREF) से धन प्राप्त द्वारा टोरंटो चिकित्सा विश्वविद्यालय ।
Materials
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
Referências
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -. J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981 (2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324 (2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -. B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607 (2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C., Doudna, J. A. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Bioquímica. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -. B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).

