DNA-Tethered RNA Polymerase für programmierbare In-vitro-Transkription und molekulare Berechnung
Summary
Wir beschreiben das Engineering einer neuartigen DNA-gebundenen T7-RNA-Polymerase zur Regulierung von In-vitro-Transkriptionsreaktionen. Wir diskutieren die Schritte für die Proteinsynthese und -charakterisierung, validieren die Proof-of-Concept-Transkriptionsregulation und diskutieren ihre Anwendungen in den Bereichen Molecular Computing, Diagnostik und molekulare Informationsverarbeitung.
Abstract
Die DNA-Nanotechnologie ermöglicht die programmierbare Selbstorganisation von Nukleinsäuren in vom Benutzer vorgegebene Formen und Dynamiken für verschiedene Anwendungen. Diese Arbeit zeigt, dass Konzepte aus der DNA-Nanotechnologie verwendet werden können, um die enzymatische Aktivität der phagenabgeleiteten T7-RNA-Polymerase (RNAP) zu programmieren und skalierbare synthetische Genregulationsnetzwerke aufzubauen. Zunächst wird ein Oligonukleotid-gebundenes T7 RNAP durch Expression eines N-terminalen SNAP-markierten RNAP und anschließende chemische Kopplung des SNAP-Tags mit einem Benzylguanin (BG)-modifizierten Oligonukleotid hergestellt. Als nächstes wird die Nuklein-Säure-Strangverschiebung verwendet, um die Polymerase-Transkription nach Bedarf zu programmieren. Darüber hinaus können Hilfs-Nukleinsäure-Assemblierungen als “künstliche Transkriptionsfaktoren” verwendet werden, um die Wechselwirkungen zwischen dem DNA-programmierten T7-RNAP und seinen DNA-Templates zu regulieren. Dieser Regulatorische Mechanismus für die In-vitro-Transkription kann eine Vielzahl von Schaltkreisverhalten wie digitale Logik, Feedback, Kaskadierung und Multiplexing implementieren. Die Zusammensetzbarkeit dieser genregulatorischen Architektur erleichtert die Designabstraktion, Standardisierung und Skalierung. Diese Funktionen werden das Rapid Prototyping von in vitro genetischen Geräten für Anwendungen wie Biosensorik, Krankheitserkennung und Datenspeicherung ermöglichen.
Introduction
DNA-Computing verwendet eine Reihe von entworfenen Oligonukleotiden als Medium für die Berechnung. Diese Oligonukleotide sind mit Sequenzen programmiert, die sich dynamisch nach einer benutzerdefinierten Logik zusammensetzen und auf spezifische Nukleinsäureeinträge reagieren. In Proof-of-Concept-Studien besteht die Ausgabe der Berechnung typischerweise aus einem Satz fluoreszierend markierter Oligonukleotide, die über Gelelektrophorese oder Fluoreszenzplattenleser nachgewiesen werden können. In den letzten 30 Jahren wurden immer komplexere DNA-Rechenschaltungen demonstriert, wie verschiedene digitale Logikkaskaden, chemische Reaktionsnetzwerke und neuronale Netze1,2,3. Um die Herstellung dieser DNA-Schaltkreise zu unterstützen, wurden mathematische Modelle verwendet, um die Funktionalität der synthetischen Genschaltkreise4,5vorherzusagen, und Berechnungswerkzeuge wurden für das orthogonale DNA-Sequenzdesign entwickelt6,7,8,9,10 . Im Vergleich zu siliziumbasierten Computern gehören zu den Vorteilen von DNA-Computern ihre Fähigkeit, direkt mit Biomolekülen zu verbinden, in Lösung zu arbeiten, wenn keine Stromversorgung vorhanden ist, sowie ihre allgemeine Kompaktheit und Stabilität. Mit dem Aufkommen der Next-Generation-Sequenzierung sind die Kosten für die Synthese von DNA-Computern in den letzten zwei Jahrzehnten schneller gesunken als das Mooresche Gesetz11. Anwendungen solcher DNA-basierten Computer beginnen sich nun zu entwickeln, wie z.B. für die Krankheitsdiagnose12,13, für die Stromversorgung der molekularen Biophysik14und als Datenspeicherplattformen15.
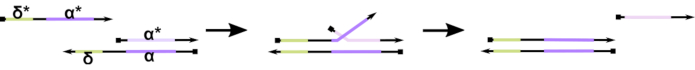
Abbildung 1: Mechanismus der Zehengriff-vermittelten DNA-Strangverschiebung. Der Toehold, δ, ist eine freie, ungebundene Sequenz auf einem Teilduplex. Wenn eine komplementäre Domäne (δ*) auf einem zweiten Strang eingeführt wird, dient die freie δ Domäne als Haltepunkt für die Hybridisierung, so dass der Rest des Strangs (ɑ*) seinen Konkurrenten durch eine reversible Reaktion, die als Strangmigration bezeichnet wird, langsam verdrängen kann. Wenn die Länge δ zunimmt, nimmt das ΔG für die Vorwärtsreaktion ab und die Verschiebung erfolgt leichter. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Bis heute verwendet die Mehrheit der DNA-Computer ein etabliertes Motiv auf dem Gebiet der dynamischen DNA-Nanotechnologie, das als Zehengriff-vermittelte DNA-Strangverschiebung (TMDSD, Abbildung 1)16bekannt ist. Dieses Motiv besteht aus einem teilweise doppelsträngigen DNA-Duplex (dsDNA), der kurze “Zehenhalte”-Überhänge (d.h. 7- bis 10 Nukleotide (nt)) aufweist. Nukleinsäure-“Eingangsstränge” können mit den Teilduplexen durch den Zehengriff interagieren. Dies führt zur Verschiebung eines der Stränge aus dem Teilduplex, und dieser freigesetzte Strang kann dann als Eingang für nachgeschaltete Teilduplexe dienen. Somit ermöglicht TMDSD die Signalkaskadierung und Informationsverarbeitung. Prinzipiell können orthogonale TMDSD-Motive in Lösung eigenständig arbeiten und ermöglichen so eine parallele Informationsverarbeitung. Es gab eine Reihe von Variationen der TMDSD-Reaktion, wie z.B. Toehold-vermittelter DNA-Strangaustausch (TMDSE)17, “lecklose” Zehengriffe mit doppelt langen Domänen18, sequenz-mismatched Zehenhalte19und “Handhold”-vermittelte Strangverschiebung20. Diese innovativen Designprinzipien ermöglichen eine feiner abgestimmte TMDSD-Energetik und -Dynamik zur Verbesserung der DNA-Rechenleistung.
Synthetische Genschaltkreise, wie transkriptionelle Genschaltkreise, sind ebenfalls in der Lage,21,22,23zu berechnen. Diese Schaltkreise werden durch Proteintranskriptionsfaktoren reguliert, die die Transkription eines Gens durch Bindung an bestimmte regulatorische DNA-Elemente aktivieren oder unterdrücken. Im Vergleich zu DNA-basierten Schaltkreisen haben Transkriptionsschaltkreise mehrere Vorteile. Erstens hat die enzymatische Transkription eine viel höhere Fluktuationsrate als bestehende katalytische DNA-Schaltkreise, wodurch mehr Kopien der Ausgabe pro einzelner Kopie der Eingabe erzeugt werden und ein effizienteres Mittel zur Signalverstärkung bereitgestellt wird. Darüber hinaus können Transkriptionsschaltkreise verschiedene funktionelle Moleküle wie Aptamere oder Boten-RNA (mRNA), die für therapeutische Proteine kodieren, als Rechenergebnisse erzeugen, die für verschiedene Anwendungen genutzt werden können. Eine wesentliche Einschränkung der aktuellen Transkriptionsschaltungen ist jedoch ihre mangelnde Skalierbarkeit. Dies liegt daran, dass es nur eine sehr begrenzte Anzahl orthogonaler Protein-basierter Transkriptionsfaktoren gibt und das De-novo-Design neuer Proteintranskriptionsfaktoren technisch anspruchsvoll und zeitaufwendig bleibt.
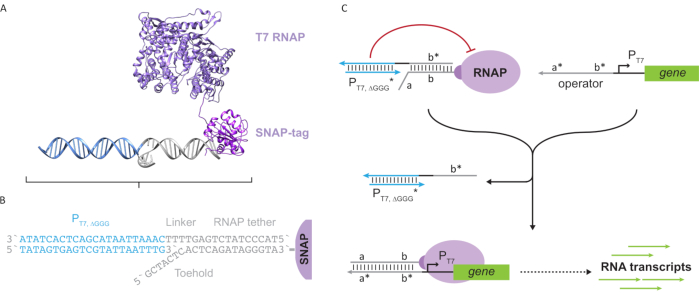
Abbildung 2: Abstraktion und Mechanismus des “Tether”- und “Cage”-Polymerase-Komplexes. (A und B) Ein Oligonukleotid-Tether wird enzymatisch durch die SNAP-Tag-Reaktion zu einer T7-Polymerase markiert. Ein Käfig, der aus einem “künstlichen” T7-Promotor mit einem Tether-Komplement-Überhang besteht, ermöglicht es, mit dem Tether zu hybridisieren und die Transkriptionsaktivität zu blockieren. (C) Wenn der Operator (a*b*) vorhanden ist, bindet er an den Haltepunkt des Oligonukleotid-Tethers (ab) und verschiebt den b*-Bereich des Käfigs, wodurch eine Transkription stattfinden kann. Diese Figur wurde von Chou und Shih27modifiziert. Abkürzungen: RNAP = RNA-Polymerase. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Dieses Papier stellt einen neuartigen Baustein für molekulares Rechnen vor, der die Funktionalitäten von Transkriptionsschaltkreisen mit der Skalierbarkeit von DNA-basierten Schaltkreisen kombiniert. Dieser Baustein ist ein T7 RNAP kovalent gebunden mit einem einzelsträngigen DNA-Tether (Abbildung 2A). Um dieses DNA-tethered T7 RNAP zu synthetisieren, wurde die Polymerase zu einem N-terminalen SNAP-Tag24 fusioniert und rekombinant in Escherichia coliexprimiert. Der SNAP-Tag wurde dann mit einem Oligonukleotid umgesetzt, das mit dem BG-Substrat funktionalisiert wurde. Das Oligonukleotid-Tether ermöglicht die Positionierung molekularer Gäste in unmittelbarer Nähe zur Polymerase mittels DNA-Hybridisierung. Einer dieser Gäste war ein kompetitiver Transkriptionsblocker, der als “Käfig” bezeichnet wird und aus einem “künstlichen” T7-Promotor-DNA-Duplex ohne nachgeschaltetes Gen besteht (Abbildung 2B). Wenn der Käfig über seinen Oligonukleotid-Tether an das RNAP gebunden ist, stoppt er die Polymeraseaktivität, indem er andere DNA-Vorlagen für die RNAP-Bindung überkompetiert, wodurch der RNAP in einen “OFF” -Zustand versetzt wird (Abbildung 2C).
Um die Polymerase in einen “ON”-Zustand zu aktivieren, wurden T7-DNA-Templates mit einzelsträngigen “Operator”-Domänen vor dem T7-Promotor des Gens entworfen. Die Operatordomäne (d.h. Domäne a*b* Abbildung 2C)kann so konstruiert werden, dass sie den Käfig über TMDSD aus dem RNAP verdrängt und die RNAP proximal zum T7-Promotor des Gens positioniert, wodurch die Transkription eingeleitet wird. Alternativ wurden auch DNA-Vorlagen entworfen, bei denen die Operatorsequenz komplementär zu Hilfs-Nukleinsäuresträngen war, die als “künstliche Transkriptionsfaktoren” bezeichnet werden (d. h. TFA- und TFB-Stränge in Abbildung 3A). Wenn beide Stränge in die Reaktion eingeführt werden, werden sie sich am Bedienerstandort zusammensetzen und eine neue pseudo-zusammenhängende Domäne a * b *erzeugen. Diese Domäne kann dann den Käfig über TMDSD verschieben, um die Transkription zu initiieren (Abbildung 3B). Diese Stränge können entweder exogen zugeführt oder hergestellt werden.
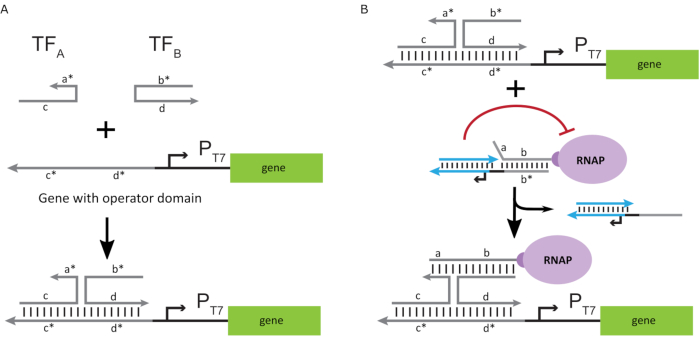
Abbildung 3: Selektive Programmierung der Polymeraseaktivität durch einen Drei-Komponenten-Schalteraktivator. (A) Wenn die Transkriptionsfaktoren (TFA und TFB) vorhanden sind, binden sie an die operatordomäne vor dem Promotor und bilden eine pseudo-einzelsträngige Sequenz (a*b*),die in der Lage ist, den Käfig durch Zehengriff-vermittelte DNA-Verschiebung zu verdrängen. (B) Diese a*b*-Domäne kann den Käfig über TMDSD verdrängen, um die Transkription zu initiieren. Diese Figur wurde von Chou und Shih27modifiziert. Abkürzungen: TF = Transkriptionsfaktor; RNAP = RNA-Polymerase; TMDSD = Toehold-vermittelte DNA-Strangverschiebung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die Verwendung von Nukleinsäure-basierten Transkriptionsfaktoren für die In-vitro-Transkriptionsregulation ermöglicht die skalierbare Implementierung anspruchsvoller Schaltkreisverhaltensweisen wie digitale Logik, Feedback und Signalkaskadierung. Zum Beispiel kann man Logik-Gate-Kaskaden bauen, indem man Nukleinsäuresequenzen so entwirft, dass die Transkripte eines upstream-Gens ein downstreams Gen aktivieren. Eine Anwendung, die die Kaskadierung und das Multiplexing nutzt, die durch diese vorgeschlagene Technologie ermöglicht werden, ist die Entwicklung ausgefeilterer molekularer Computerschaltungen für die tragbare Diagnostik und molekulare Datenverarbeitung. Darüber hinaus kann die Integration der Molekularen Computer- und De-novo-RNA-Synthesefähigkeiten neue Anwendungen ermöglichen. Zum Beispiel kann ein molekularer Schaltkreis entworfen werden, um eine oder eine Kombination von benutzerdefinierten RNAs als therapeutische Input- und Output-RNAs oder mRNAs zu erkennen, die funktionelle Peptide oder Proteine für medizinische Point-of-Care-Anwendungen kodieren.
Protocol
Representative Results
Discussion
Diese Studie zeigt einen von der DNA-Nanotechnologie inspirierten Ansatz zur Kontrolle der Aktivität der T7-RNA-Polymerase durch kovalente Kopplung eines N-terminalen SNAP-markierten rekombinanten T7 RNAP mit einem BG-funktionalisierten Oligonukleotid, das anschließend zur Programmierung von TMDSD-Reaktionen verwendet wurde. Durch das Design wurde der SNAP-Tag am N-Terminus der Polymerase positioniert, da der C-Terminus des Wildtyps T7 RNAP im Proteinstrukturkern vergraben ist und wichtige Kontakte mit der DNA-Vorlage<…
Declarações
The authors have nothing to disclose.
Acknowledgements
L.Y.T.C würdigt die großzügige Unterstützung des New Frontiers in Research Fund-Exploration (NFRF-E), des Discovery Grant des Natural Sciences and Engineering Research Council of Canada (NSERC) und der Medicine by Design Initiative der University of Toronto, die vom Canada First Research Excellence Fund (CFREF) finanziert wird.
Materials
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
Referências
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -. J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981 (2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324 (2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -. B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607 (2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C., Doudna, J. A. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Bioquímica. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -. B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).

