ARN polymérase attachée à l’ADN pour la transcription in vitro programmable et le calcul moléculaire
Summary
Nous décrivons l’ingénierie d’une nouvelle ARN polymérase T7 attachée à l’ADN pour réguler les réactions de transcription in vitro. Nous discutons des étapes de la synthèse et de la caractérisation des protéines, validons la régulation transcriptionnelle de la preuve de concept et discutons de ses applications en informatique moléculaire, en diagnostic et en traitement de l’information moléculaire.
Abstract
La nanotechnologie de l’ADN permet l’auto-assemblage programmable des acides nucléiques en formes et dynamiques prescrites par l’utilisateur pour diverses applications. Ces travaux démontrent que les concepts de la nanotechnologie de l’ADN peuvent être utilisés pour programmer l’activité enzymatique de l’ARN polymérase T7 dérivée des phages (RNAP) et construire des réseaux de régulation de gènes synthétiques évolutifs. Tout d’abord, un RNAP T7 attaché à un oligonucléotide est conçu par l’expression d’un RNAP marqué SNAP N-terminal et le couplage chimique ultérieur du marqueur SNAP avec un oligonucléotide modifié par benzylguanine (BG). Ensuite, le déplacement des brins d’acide nucléique est utilisé pour programmer la transcription de la polymérase à la demande. En outre, les assemblages auxiliaires d’acides nucléiques peuvent être utilisés comme « facteurs de transcription artificiels » pour réguler les interactions entre l’ARNP T7 programmé par l’ADN et ses modèles d’ADN. Ce mécanisme de régulation de la transcription in vitro peut mettre en œuvre une variété de comportements de circuit tels que la logique numérique, la rétroaction, la cascade et le multiplexage. La composabilité de cette architecture de régulation des gènes facilite l’abstraction, la normalisation et la mise à l’échelle de la conception. Ces caractéristiques permettront le prototypage rapide de dispositifs génétiques in vitro pour des applications telles que la biodétection, la détection de maladies et le stockage de données.
Introduction
Le calcul de l’ADN utilise un ensemble d’oligonucléotides conçus comme support de calcul. Ces oligonucléotides sont programmés avec des séquences pour s’assembler dynamiquement selon la logique spécifiée par l’utilisateur et répondre à des entrées d’acide nucléique spécifiques. Dans les études de preuve de concept, le résultat du calcul consiste généralement en un ensemble d’oligonucléotides marqués par fluorescence qui peuvent être détectés par électrophorèse sur gel ou lecteurs de plaques de fluorescence. Au cours des 30 dernières années, des circuits de calcul de l’ADN de plus en plus complexes ont été démontrés, tels que diverses cascades logiques numériques, des réseaux de réactions chimiques et des réseaux de neurones1,2,3. Pour aider à la préparation de ces circuits d’ADN, des modèles mathématiques ont été utilisés pour prédire la fonctionnalité des circuits de gènes synthétiques4,5, et des outils informatiques ont été développés pour la conception de séquences d’ADN orthogonales6,7,8,9,10 . Par rapport aux ordinateurs à base de silicium, les avantages des ordinateurs à ADN comprennent leur capacité à s’interfacer directement avec les biomolécules, à fonctionner en solution en l’absence d’alimentation électrique, ainsi que leur compacité et leur stabilité globales. Avec l’avènement du séquençage de nouvelle génération, le coût de la synthèse des ordinateurs à ADN a diminué au cours des deux dernières décennies à un rythme plus rapide que la loi de Moore11. Des applications de ces ordinateurs basés sur l’ADN commencent maintenant à émerger, telles que pour le diagnostic des maladies12,13, pour alimenter la biophysique moléculaire14, et comme plates-formes de stockage de données15.
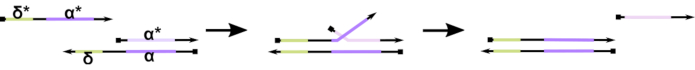
Figure 1: Mécanisme de déplacement des brins d’ADN médiés par les orteils. L’orteil, δ, est une séquence libre et non liée sur un duplex partiel. Lorsqu’un domaine complémentaire (δ*) est introduit sur un deuxième brin, le domaine δ libre sert de base pour l’hybridation, permettant au reste du brin (ɑ*) de déplacer lentement son concurrent par une réaction réversible de zipping/décompression connue sous le nom de migration de brin. À mesure que la longueur de δ augmente, le ΔG de la réaction vers l’avant diminue et le déplacement se produit plus facilement. Veuillez cliquer ici pour voir une version agrandie de cette figure.
À ce jour, la majorité des ordinateurs à ADN utilisent un motif bien établi dans le domaine de la nanotechnologie dynamique de l’ADN connu sous le nom de déplacement de brin d’ADN médié par les orteils (TMDSD, Figure 1)16. Ce motif est constitué d’un duplex d’ADN partiellement double brin (dsDNA) affichant de courts surplombs « toehold » (c’est-à-dire 7 à 10 nucléotides (nt)). Les brins d’entrée d’acide nucléique peuvent interagir avec les duplex partiels à travers l’orteil. Cela conduit au déplacement de l’un des brins du duplex partiel, et ce brin libéré peut alors servir d’entrée pour les duplex partiels en aval. Ainsi, TMDSD permet la cascade du signal et le traitement de l’information. En principe, les motifs tmDSD orthogonaux peuvent fonctionner indépendamment dans la solution, ce qui permet un traitement parallèle de l’information. Il y a eu un certain nombre de variations sur la réaction TMDSD, telles que l’échange de brins d’ADN médié par la toehold (TMDSE)17,les orteils « sans fuite » avec des domaines doubles longs18,les orteils dépareillés par séquence19et le déplacement de brin médié par la « poignée »20. Ces principes de conception innovants permettent une énergie et une dynamique TMDSD plus finement ajustées pour améliorer les performances de calcul de l’ADN.
Les circuits de gènes synthétiques, tels que les circuits de gènes transcriptionnels, sont également capables de calculer21,22,23. Ces circuits sont régulés par des facteurs de transcription des protéines, qui activent ou répriment la transcription d’un gène en se liant à des éléments d’ADN régulateurs spécifiques. Par rapport aux circuits à base d’ADN, les circuits transcriptionnels présentent plusieurs avantages. Premièrement, la transcription enzymatique a un taux de rotation beaucoup plus élevé que les circuits d’ADN catalytiques existants, générant ainsi plus de copies de sortie par copie unique d’entrée et fournissant un moyen plus efficace d’amplification du signal. En outre, les circuits transcriptionnels peuvent produire différentes molécules fonctionnelles, telles que des aptamères ou des ARN messagers (ARNm) codant pour des protéines thérapeutiques, en tant que sorties de calcul, qui peuvent être exploitées pour différentes applications. Cependant, une limitation majeure des circuits transcriptionnels actuels est leur manque d’évolutivité. En effet, il existe un ensemble très limité de facteurs de transcription basés sur des protéines orthogonales, et la conception de novo de nouveaux facteurs de transcription protéique reste techniquement difficile et prend beaucoup de temps.
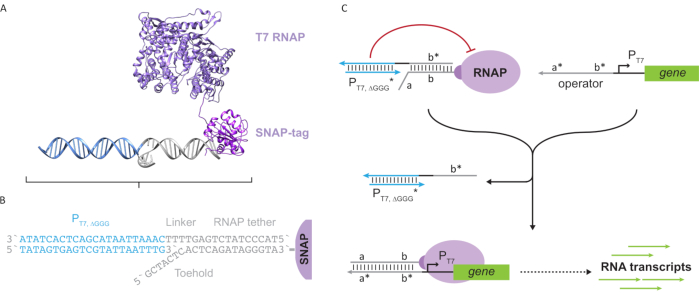
Figure 2: Abstraction et mécanisme du complexe de polymérase « attache » et « cage ». (A et B) Une téther oligonucléotidique est marquée enzymatiquement en une polymérase T7 par la réaction SNAP-tag. Une cage constituée d’un « faux » promoteur T7 avec un porte-à-faux attache-complément lui permet de s’hybrider à l’attache et de bloquer l’activité transcriptionnelle. (C) Lorsque l’opérateur (a*b*) est présent, il se lie à l’orteil sur l’attache oligonucléotidique (ab) et déplace la région b* de la cage, permettant la transcription. Cette figure a été modifiée à partir de Chou et Shih27. Abréviations : RNAP = ARN polymérase. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Cet article présente un nouveau bloc de construction pour l’informatique moléculaire qui combine les fonctionnalités des circuits transcriptionnels avec l’évolutivité des circuits à base d’ADN. Ce bloc de construction est un RNAP T7 attaché de manière covalente avec une attache d’ADN simple brin(Figure 2A). Pour synthétiser ce RNAP T7 attaché à l’ADN, la polymérase a été fusionnée à un marqueur SNAP N-terminal24 et exprimée de manière recombinante dans Escherichia coli. Le SNAP-tag a ensuite réagi avec un oligonucléotide fonctionnalisé avec le substrat BG. L’attache oligonucléotidique permet le positionnement d’invités moléculaires à proximité de la polymérase via l’hybridation de l’ADN. L’un de ces invités était un bloqueur transcriptionnel compétitif appelé « cage », qui consiste en un « faux » duplex d’ADN promoteur T7 sans gène en aval(Figure 2B). Lorsqu’elle est liée à l’ARNP via son attache oligonucléotidique, la cage bloque l’activité de la polymérase en surpassant les autres modèles d’ADN pour la liaison À l’ARNP, rendant l’ARNP dans un état « OFF »(Figure 2C).
Pour activer la polymérase à un état « ON », des modèles d’ADN T7 avec des domaines « opérateurs » monocaténaires en amont du promoteur T7 du gène ont été conçus. Le domaine de l’opérateur (c’est-à-dire le domaine a*b* Figure 2C) peut être conçu pour déplacer la cage de l’ARNP via TMDSD et positionner le RNAP proximal au promoteur T7 du gène, initiant ainsi la transcription. Alternativement, des modèles d’ADN ont également été conçus lorsque la séquence de l’opérateur était complémentaire aux brins auxiliaires d’acide nucléique appelés « facteurs de transcription artificiels » (c.-à-d. les brins TFA et TFB à la figure 3A). Lorsque les deux brins sont introduits dans la réaction, ils s’assemblent sur le site de l’opérateur, créant un nouveau domaine pseudo-contigu a*b*. Ce domaine peut ensuite déplacer la cage via TMDSD pour initier la transcription(Figure 3B). Ces brins peuvent être fournis de manière exogène ou produits.
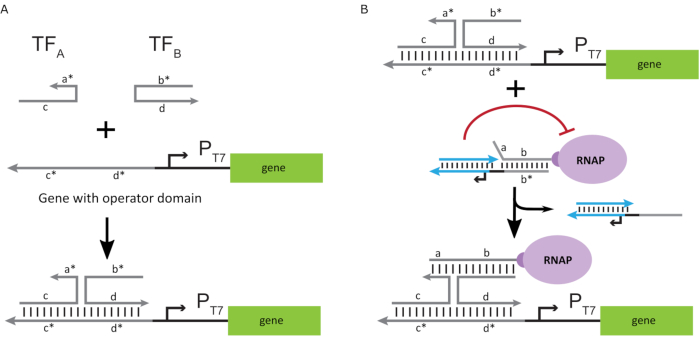
Figure 3: Programmation sélective de l’activité de la polymérase à l’aide d’un activateur de commutation à trois composants. (A) Lorsque les facteurs de transcription (TFA et TFB)sont présents, ils se lient au domaine opérateur en amont du promoteur, formant une pseudo séquence monocaténaire (a*b*)capable de déplacer la cage par déplacement d’ADN médié par l’orteil. (B) Ce domaine a*b* peut déplacer la cage via TMDSD pour initier la transcription. Cette figure a été modifiée à partir de Chou et Shih27. Abréviations : TF = facteur de transcription ; RNAP = ARN polymérase; TMDSD = déplacement de brin d’ADN médié par les orteils. Veuillez cliquer ici pour voir une version agrandie de cette figure.
L’utilisation de facteurs de transcription à base d’acides nucléiques pour la régulation transcriptionnelle in vitro permet la mise en œuvre évolutive de comportements de circuit sophistiqués tels que la logique numérique, la rétroaction et la cascade de signaux. Par exemple, on peut construire des cascades de portes logiques en concevant des séquences d’acides nucléiques telles que les transcriptions d’un gène en amont activent un gène en aval. Une application qui exploite la cascade et le multiplexage rendus possibles par cette technologie proposée est le développement de circuits informatiques moléculaires plus sophistiqués pour le diagnostic portable et le traitement des données moléculaires. En outre, l’intégration des capacités de calcul moléculaire et de synthèse d’ARN de novo peut permettre de nouvelles applications. Par exemple, un circuit moléculaire peut être conçu pour détecter un ou une combinaison d’ARN définis par l’utilisateur en tant qu’ENTRÉES ET SORTIES D’ARN thérapeutiques ou d’ARNm codant pour des peptides ou des protéines fonctionnels pour des applications médicales au point de service.
Protocol
Representative Results
Discussion
Cette étude démontre une approche inspirée de la nanotechnologie de l’ADN pour contrôler l’activité de l’ARN polymérase T7 en couplant de manière covalente un RNAP T7 recombinant marqué N-terminal SNAP avec un oligonucléotide fonctionnalisé BG, qui a ensuite été utilisé pour programmer les réactions TMDSD. De par sa conception, la balise SNAP a été positionnée à la terminaison N de la polymérase, car la terminaison C de l’ARNR T7 de type sauvage est enfouie dans le noyau de la structure protéi…
Declarações
The authors have nothing to disclose.
Acknowledgements
L.Y.T.C reconnaît le généreux soutien du Fonds-exploration Nouvelles frontières en recherche (NFRF-E), de la subvention de découverte du Conseil de recherches en sciences naturelles et en génie du Canada (CRSNG) et de l’initiative Medicine by Design de l’Université de Toronto, qui reçoit un financement du Fonds d’excellence en recherche Canada First (CFREF).
Materials
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
Referências
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -. J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981 (2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324 (2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -. B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607 (2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C., Doudna, J. A. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Bioquímica. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -. B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).

