プログラム可能なインビトロ転写と分子計算のためのDNAテザリングRNAポリメラーゼ
Summary
インビトロ転写反応を調節する新規DNAつながされたT7 RNAポリメラーゼの工学について述べています。タンパク質合成と特性評価のステップについて議論し、概念実証の転写制御を検証し、分子コンピューティング、診断、分子情報処理における応用について議論する。
Abstract
DNAナノテクノロジーは、核酸をプログラム可能な自己集合化をユーザー指定の形状および多様なアプリケーションのためのダイナミクスに可能にします。この研究は、DNAナノテクノロジーの概念を使用して、ファージ由来のT7 RNAポリメラーゼ(RNAP)の酵素活性をプログラムし、スケーラブルな合成遺伝子調節ネットワークを構築できることを実証しています。まず、オリゴヌクレオチドと結合したT7 RNAPは、N末端SNAPタグ付きRNAPの発現と、その後のSNAPタグとベンジルグアニン(BG)修飾オリゴヌクレオチドとの化学的結合を介して設計される。次に、核酸鎖変位を使用して、オンデマンドでポリメラーゼ転写をプログラムします。さらに、補助核酸アセンブリを「人工転写因子」として使用して、DNAプログラムT7 RNAPとDNAテンプレートとの相互作用を調節することができます。このインビトロ転写調節機構は、デジタルロジック、フィードバック、カスケード、多重化などの様々な回路動作を実装できます。この遺伝子調節アーキテクチャの構成性は、設計の抽象化、標準化、およびスケーリングを促進します。これらの機能により、生体センシング、疾患検出、データ保存などのアプリケーションに対するインビトロ遺伝子デバイスの迅速な試作が可能になります。
Introduction
DNAコンピューティングは、計算の媒体として設計されたオリゴヌクレオチドのセットを使用します。これらのオリゴヌクレオチドは、ユーザー指定のロジックに従って動的に組み立て、特定の核酸入力に応答する配列でプログラムされています。概念実証研究では、計算の出力は、典型的には、ゲル電気泳動または蛍光プレートリーダーを介して検出することができる蛍光標識オリゴヌクレオチドのセットで構成されています。過去30年間で、さまざまなデジタル論理カスケード、化学反応ネットワーク、ニューラルネットワーク1、2、3など、ますます複雑化するDNA計算回路が実証されています。これらのDNA回路の作製を支援するために、合成遺伝子回路4、5の機能性を予測するために数理モデルが使用され、直交DNA配列設計6、7、8、9、10の計算ツールが開発されています。.シリコンベースのコンピュータと比較すると、DNAコンピュータの利点は、生体分子と直接インターフェースし、電源がない場合に溶液中で動作する能力、ならびに全体的なコンパクト性と安定性を備えています。次世代シーケンシングの出現により、DNAコンピュータの合成コストは、ムーアの法則11よりも速い速度で過去20年間減少しています。このようなDNAベースのコンピュータの応用は、現在、疾患診断12、13、分子生物物理学14、およびデータ記憶プラットフォーム15として、現れ始めている。
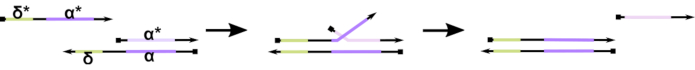
図1:足のホールド媒介DNA鎖変位のメカニズム δの間のつま先は、部分二重での自由な非バインド シーケンスです。第2の鎖に相補的なドメイン(δ*)が導入されると、フリー δドメインはハイブリダイゼーションの足掛かりとして機能し、ストランド移行と呼ばれる圧縮/解凍可逆反応を通じて競合他社をゆっくりと置き換えることを可能にします( 安打 *)δの長さが長くなるにつれて、前進反応のΔGは減少し、変位はより容易に起こる。 この図の大きなバージョンを表示するには、ここをクリックしてください。
現在までに、DNAコンピュータの大部分は、足のホールド媒介DNA鎖変位として知られる動的DNAナノテクノロジーの分野において確立されたモチーフを利用している(TMDSD,図1)16。このモチーフは、短い「足掛け」の突出し(すなわち、7-〜10ヌクレオチド(nt))を示す部分的に二本鎖DNA(dsDNA)二重から成る。核酸「入力」鎖は、足止めを通して部分的な二重鎖と相互作用することができる。これにより、部分的なデュプレックスからのストランドの 1 つの変位が起き、この解放されたストランドは下流の部分的なデュプレックスの入力として機能します。このように、TMDSD は信号のカスケードと情報処理を可能にします。基本的に、直交TMDSDモチーフは、ソリューションで独立して動作することができ、並列情報処理を可能にします。TMDSD反応には、足がかりを媒介するDNA鎖交換(TMDSE)17、二重長ドメイン18との「リークレス」足音、配列不一致の足掛かり19、および「ハンドホールド」媒介鎖変位20など多くのバリエーションがあった。これらの革新的な設計原則により、より細かく調整されたTMDSDのエネルギーとダイナミクスがDNAコンピューティングの性能を向上させることができます。
転写遺伝子回路などの合成遺伝子回路は、計算21、22、23も可能である。これらの回路は、タンパク質転写因子によって調節され、特定の調節DNA要素に結合することによって遺伝子の転写を活性化または再抑制する。DNAベースの回路と比較して、転写回路には複数の利点があります。まず、酵素転写は既存の触媒DNA回路よりもはるかに高い回転率を有し、入力の単一コピー当たりの出力のコピーが多く生成され、より効率的な信号増幅手段を提供する。さらに、転写回路は、治療用タンパク質をコードするアプタマーやメッセンジャーRNA(mRNA)などの異なる機能分子を計算出力として生成することができ、異なる用途に利用することができます。しかし、現在の転写回路の大きな制限は、スケーラビリティの欠如です。これは、直交タンパク質ベースの転写因子のセットが非常に限られており、新しいタンパク質転写因子のデノボ設計は技術的に困難で時間がかかるためです。
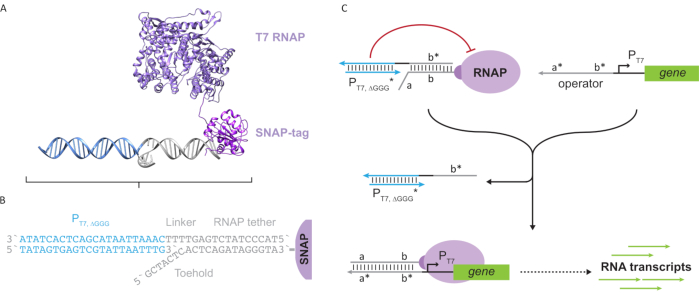
図2:「テザー」および「ケージ」ポリメラーゼ複合体の抽象化およびメカニズム(AおよびB)のオリゴヌクレオチドテザーは、SNAPタグ反応を通じてT7ポリメラーゼに酵素的に標識されている。テザー補体オーバーハングを有する「フェイク」T7プロモーターからなるケージは、テザーとブロック転写活性にハイブリダイズすることを可能にする。(C)オペレータ(a*b*)が存在すると、オリゴヌクレオチドテザー(ab)上のつま先に結合し、ケージのb*領域を置き換え、転写が起こることを可能にする。この図は、チョウとシー27から変更されています。略語: RNAP = RNA ポリメラーゼこの図の大きなバージョンを表示するには、ここをクリックしてください。
この論文では、転写回路の機能性とDNAベース回路の拡張性を組み合わせた分子計算のための新しいビルディングブロックを紹介する。このビルディングブロックは、T7 RNAP共有結合体である一本鎖DNAテザー(図2A)このDNAテザリングT7 RNAPを合成するために、ポリメラーゼをN末端SNAPタグ24に融合させ、エシェリヒア・コリで組換え発現させた。次いで、SNAPタグを、BG基質と機能するオリゴヌクレオチドと反応させた。オリゴヌクレオチドテザーは、DNAハイブリダイゼーションを介してポリメラーゼに近接した分子ゲストの位置決めを可能にする。そのようなゲストの1つは、下流に遺伝子のない「フェイク」T7プロモーターDNAデュプレックスで構成される「ケージ」と呼ばれる競争的な転写ブロッカーでした(図2B)。オリゴヌクレオチドテザーを介してRNAPに結合すると、ケージはRNAP結合のための他のDNAテンプレートを上回ることによってポリメラーゼ活性を失速させ、RNAPを「オフ」状態でレンダリングする(図2C)。
ポリメラーゼを「ON」状態に活性化させるために、T7遺伝子のT7プロモーターの上流に一本鎖「オペレータ」ドメインを有するT7 DNAテンプレートが設計された。オペレータドメイン(すなわち、ドメインa*b*図2C)は、TMDSDを介してRNAPからケージを置き換え、RNAPを遺伝子のT7プロモーターに近い位置に置き、したがって転写を開始するように設計することができる。あるいは、DNAテンプレートは、オペレータ配列が「人工転写因子」と呼ばれる補助核酸鎖と相補的であるところも設計された(すなわち、図3AのTFAおよびTFB鎖)。両方のストランドが反応に導入されると、それらはオペレータサイトで組み立てられ、新しい擬似連続ドメインa*b*が作成されます。このドメインは、TMDSD を介してケージを置き換えて、転写を開始することができます (図 3B)。これらのストランドは外因性に供給されるか、または製造することができる。
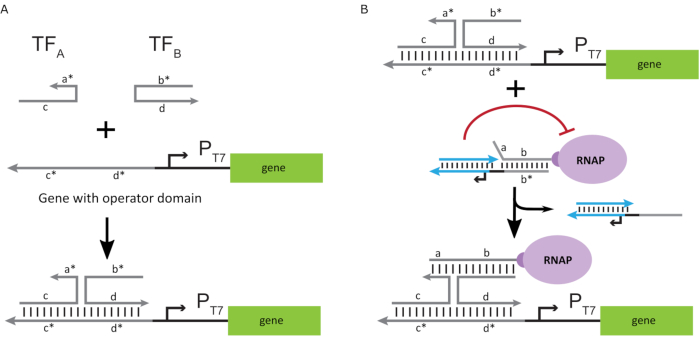
図3:3成分スイッチアクチベータを用いたポリメラーゼ活性の選択的プログラミング (A) 転写因子(TFA およびTFB)が存在する場合、それらはプロモーターのオペレータドメイン上流に結合し、トーホールド媒介DNA変位を通してケージを変位させることができる疑似一本鎖配列(a*b*)を形成する。(B) この a*b* ドメインは、TMDSDを介してケージを置き換えて転写を開始することができます。この図は、チョウとシー27から変更されています。略語: TF = 転写因子;RNAP = RNA ポリメラーゼ;TMDSD = 足掛け媒介DNA鎖変位。 この図の大きなバージョンを表示するには、ここをクリックしてください。
インビトロ転写制御に核酸ベースの転写因子を使用することで、デジタルロジック、フィードバック、信号カスケードなどの高度な回路挙動をスケーラブルに実装できます。例えば、上流の遺伝子からの転写産物が下流遺伝子を活性化するような核酸配列を設計することによって、ロジックゲートカスケードを構築することができます。この提案された技術によって可能なカスケードと多重化を利用する1つのアプリケーションは、ポータブル診断と分子データ処理のためのより洗練された分子コンピューティング回路の開発です。さらに、分子計算とデノボRNA合成機能の統合により、新たなアプリケーションが可能になります。例えば、分子回路は、ポイント・オブ・ケア・メディカル・アプリケーション用の機能性ペプチドまたはタンパク質をコードする機能性ペプチドまたはタンパク質をコードする、入力および出力治療用RNAまたはmRNAとして、ユーザー定義RNAの1つまたは組み合わせを検出するように設計することができる。
Protocol
Representative Results
Discussion
本研究は、N末端SNAPタグ組換えT7 RNAPとBG官能オリゴヌクレオチドを共有結合させることによってT7 RNAポリメラーゼの活性を制御するDNAナノテクノロジーに触発されたアプローチを示し、その後TMDSD反応をプログラムするために使用された。設計上、SNAPタグはポリメラーゼのN末語に位置付けられており、野生型T7 RNAPのC末語がタンパク質構造コア内に埋もれ、DNAテンプレート28…
Declarações
The authors have nothing to disclose.
Acknowledgements
L.Y.T.Cは、カナダ自然科学工学研究評議会(NSERC)ディスカバリー・グラント、カナダ第一研究優秀基金(CFREF)から資金を受け取るトロント大学の医学設計イニシアチブに関する研究基金探査(NFRF-E)の新フロンティアからの寛大な支援を認めています。
Materials
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
Referências
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -. J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981 (2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324 (2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -. B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607 (2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C., Doudna, J. A. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Bioquímica. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -. B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).

