الحمض النووي المربوطة الحمض النووي RNA بوليمراز للبرمجة في النسخ المختبري والحوسبة الجزيئية
Summary
نحن نصف هندسة رواية الحمض النووي المربوطة T7 الحمض النووي الحمض النووي RNA بوليمرات لتنظيم ردود الفعل في المختبر النسخ. نناقش خطوات تخليق البروتين وتوصيفه، والتحقق من صحة تنظيم النسخ لإثبات المفهوم، ومناقشة تطبيقاته في الحوسبة الجزيئية، والتشخيص، ومعالجة المعلومات الجزيئية.
Abstract
تكنولوجيا النانو الحمض النووي تمكن البرمجة الذاتي التجميع من الأحماض النووية في الأشكال التي يحددها المستخدم وديناميات لتطبيقات متنوعة. هذا العمل يدل على أن المفاهيم من تكنولوجيا النانو الحمض النووي يمكن استخدامها لبرنامج النشاط الأنزيمي من البوليميراز T7 الحمض النووي الريبي المستمدة من phage (RNAP) وبناء شبكات تنظيمية الجينات الاصطناعية قابلة للتطوير. أولا، تم تصميم RNAP T7 المربوطة oligonucleotide عن طريق التعبير عن RNAP N-TERMINALLY SNAP الموسومة والربط الكيميائي اللاحق للSNAP-tag مع oligonucleotide المعدلة بنزيلجوانين (BG). بعد ذلك ، يتم استخدام إزاحة حبلا الحمض النووي لبرنامج نسخ البوليميراز عند الطلب. بالإضافة إلى ذلك، يمكن استخدام تجميعات الحمض النووي المساعدة ك “عوامل نسخ اصطناعية” لتنظيم التفاعلات بين T7 RNAP المبرمجة بالحمض النووي مع قوالب الحمض النووي الخاصة به. يمكن لهذه الآلية التنظيمية للنسخ في المختبر تنفيذ مجموعة متنوعة من سلوكيات الدوائر مثل المنطق الرقمي ، وردود الفعل ، المتتالية ، والتعدد. تسهل قابلية إنشاء هذه البنية التنظيمية الجينية تجريد التصميم والتوحيد القياسي والتحجيم. وستمكن هذه الميزات من النماذج الأولية السريعة للأجهزة الوراثية المختبرية لتطبيقات مثل الاستشعار البيولوجي، والكشف عن الأمراض، وتخزين البيانات.
Introduction
تستخدم حوسبة الحمض النووي مجموعة من أوليغونوكليوتيدات مصممة كوسيلة للحساب. تتم برمجة هذه oligonucleotides مع تسلسل لتجميع ديناميكي وفقا للمنطق المحدد من قبل المستخدم والاستجابة لمدخلات الحمض النووي محددة. في دراسات إثبات المفهوم ، يتكون ناتج الحساب عادة من مجموعة من أوليغونوكليوتيدات الفلورسنت التي يمكن اكتشافها عن طريق الكهروضوئيات الهلامية أو قارئات لوحات الفلورسينس. على مدى السنوات ال 30 الماضية ، وقد ثبت الدوائر الحسابية الحمض النووي معقدة على نحو متزايد ، مثل مختلف الشلالات المنطق الرقمي ، وشبكات التفاعل الكيميائي ، والشبكات العصبية1،2،3. للمساعدة في إعداد هذه الدوائر الحمض النووي، وقد استخدمت نماذج رياضية للتنبؤ وظائف الدوائر الجينية الاصطناعية4،5، وقد وضعت أدوات حسابية لتصميم تسلسل الحمض النووي متعامدة6،7،8،9،10 . بالمقارنة مع أجهزة الكمبيوتر القائمة على السيليكون ، وتشمل مزايا أجهزة الكمبيوتر الحمض النووي قدرتها على التفاعل مباشرة مع الجزيئات الحيوية ، وتعمل في حل في غياب إمدادات الطاقة ، فضلا عن إحكام واستقرارها العام. مع ظهور تسلسل الجيل القادم ، كانت تكلفة تصنيع أجهزة الكمبيوتر الحمض النووي في انخفاض على مدى العقدين الماضيين بمعدل أسرع من قانون مور11. تطبيقات هذه الحواسيب المستندة إلى الحمض النووي بدأت تظهر الآن ، مثل لتشخيص المرض12،13، لتشغيل الفيزياء الحيوية الجزيئية14، وكمنصات لتخزين البيانات15.
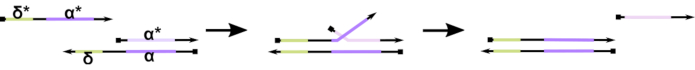
الشكل 1:آلية إزاحة حبلا الحمض النووي بوساطة موطئ القدم. موطئ القدم، δ، هو تسلسل حر وغير منضم على مزدوجة جزئية. عندما يتم تقديم مجال تكميلي (δ*) على حبلا ثان ، يعمل نطاق δ الحر كمسند إصبع قدم للتهجين ، مما يسمح لبقية الخيط (Ο*) بإزاحة منافسه ببطء من خلال رد فعل قابل للعكس مضغوط / فك الضغط يعرف باسم هجرة حبلا. مع زيادة طول δ، ينخفض ΔG لرد الفعل الأمامي، ويحدث الإزاحة بسهولة أكبر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
حتى الآن ، فإن غالبية أجهزة الكمبيوتر الحمض النووي استخدام عزر راسخة في مجال تكنولوجيا النانو الحمض النووي الديناميكي المعروف باسم التشريد حبلا الحمض النووي بوساطة موطئ القدم (TMDSD ، الشكل 1)16. يتكون هذا الشعار من ثنائية جزئيا تقطعت بهم السبل الحمض النووي (dsDNA) المزدوج المزدوج عرض قصيرة “موطئ قدم” يتدلى (أي 7- إلى 10 النيوكليوتيدات (NT)). يمكن أن تتفاعل خيوط “الإدخال” من الحمض النووي مع الدوبلين الجزئي من خلال موطئ القدم. يؤدي هذا إلى إزاحة أحد الخيوط من الدوبلين الجزئي، ويمكن أن يكون هذا الخيط المحرر بمثابة مدخل للدوبلين الجزئيين في المصب. وهكذا، TMDSD تمكن تتالي الإشارات ومعالجة المعلومات. من حيث المبدأ ، يمكن أن تعمل زخارف TMDSD المتعامدة بشكل مستقل في الحل ، مما يتيح معالجة المعلومات الموازية. كان هناك عدد من الاختلافات على رد فعل TMDSD، مثل تبادل حبلا الحمض النووي بوساطة موطئ القدم (TMDSE)17، “تسرب” موطئ قدم مع نطاقات مزدوجة طويلة18،أضواء غير متطابقة تسلسل19،و “اليد” بوساطة حبلاالنزوح 20. تسمح مبادئ التصميم المبتكرة هذه بمزيد من حيوية وديناميكيات TMDSD المضبوطة بدقة لتحسين أداء حوسبة الحمض النووي.
الدوائر الجينية الاصطناعية، مثل الدوائر الجينية النسخية، هي أيضا قادرة على حساب21،22،23. يتم تنظيم هذه الدوائر من خلال عوامل نسخ البروتين ، والتي تنشط أو تقمع نسخ الجين عن طريق الربط بعناصر الحمض النووي التنظيمية المحددة. بالمقارنة مع الدوائر المستندة إلى الحمض النووي ، فإن الدوائر النسخية لها العديد من المزايا. أولا، النسخ الأنزيمي له معدل دوران أعلى بكثير من دوائر الحمض النووي الحفازة الحالية، وبالتالي توليد نسخ أكثر من الإخراج لكل نسخة واحدة من المدخلات وتوفير وسيلة أكثر كفاءة لتضخيم الإشارات. بالإضافة إلى ذلك ، يمكن أن تنتج الدوائر النسخية جزيئات وظيفية مختلفة ، مثل aptamers أو ترميز الحمض النووي الريبي messenger (mRNA) للبروتينات العلاجية ، كخرجات حساب ، والتي يمكن استغلالها لتطبيقات مختلفة. ومع ذلك ، فإن أحد القيود الرئيسية للدوائر النسخية الحالية هو عدم قابليتها للتوسع. وذلك لأن هناك مجموعة محدودة جدا من عوامل النسخ القائمة على البروتين المتعامدة، وتصميم دي نوفو من عوامل نسخ البروتين الجديدة لا تزال صعبة من الناحية الفنية وتستغرق وقتا طويلا.
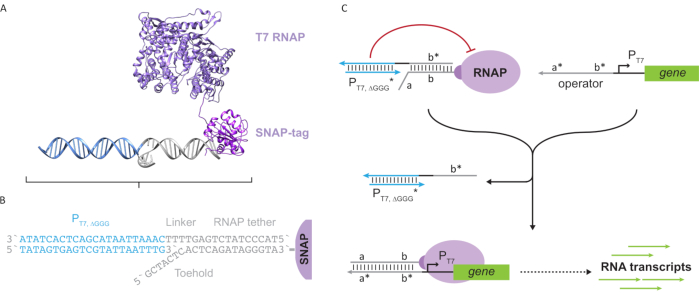
الشكل 2: التجريد وآلية “الحبل” و “قفص” مجمع البوليميراز. (A و B) يتم وضع علامة على حبل أوليغونوكليوتيد أنزيميا إلى بوليمراز T7 من خلال رد فعل SNAP-tag. قفص يتكون من “فو” T7 المروج مع تراكم حبل تكملة يسمح لها التهجين إلى الحبل ومنع النشاط النسخي. (ج) عندما يكون المشغل(أ * ب *) موجودا ، فإنه يرتبط بمؤشر القدم على حبل oligonucleotide(ab)ويشرد منطقة b * من القفص ، مما يسمح بالنص. تم تعديل هذا الرقم من تشو وشيه27. الاختصارات: RNAP = RNA بوليمراز. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
تقدم هذه الورقة لبنة جديدة للحوسبة الجزيئية تجمع بين وظائف الدوائر النسخية وقابلية التوسع في الدوائر القائمة على الحمض النووي. هذه الكتلة البناء هو T7 RNAP المرفقة بشكل مشترك مع حبل الحمض النووي واحد تقطعت بهم السبل (الشكل 2A). لتجميع هذا الحمض النووي المربوطة T7 RNAP، تم دمج البوليميراز إلى N-محطة SNAP-tag24 وأعرب عنه بشكل مركب في الإشريكية القولونية. ثم تفاعلت علامة SNAP مع أوليغونوكليوتيد وظيفية مع ركيزة BG. يسمح حبل أوليغونوكليوتيد بوضع الضيوف الجزيئيين على مقربة من البوليميراز عن طريق تهجين الحمض النووي. وكان أحد هؤلاء الضيوف مانع النسخ التنافسية المشار إليها باسم “قفص”، الذي يتكون من “فو” T7 المروج الحمض النووي المزدوج مع عدم وجود جين المصب(الشكل 2B). عندما ملزمة RNAP عبر الحبل oligonucleotide لها، القفص الأكشاك نشاط البوليميراز عن طريق المنافسة قوالب الحمض النووي الأخرى لربط RNAP، مما يجعل RNAP في حالة “إيقاف” (الشكل 2C).
لتنشيط البوليميراز إلى حالة “ON” ، تم تصميم قوالب الحمض النووي T7 مع نطاقات “المشغل” الوحيدة التي تقطعت بها السبل في المنبع لمروج T7 للجين. يمكن تصميم مجال المشغل (أي المجال أ * ب * الشكل 2C)لإزاحة القفص من RNAP عبر TMDSD ووضع RNAP في مكان قريب من مروج T7 للجين ، وبالتالي بدء النسخ. وبدلا من ذلك، صممت قوالب الحمض النووي أيضا حيث كان تسلسل المشغل مكملا لخيوط الحمض النووي المساعدة التي يشار إليها باسم “عوامل النسخ الاصطناعي” (أي فروع TFA و TFB في الشكل 3A). عندما يتم إدخال كلا الخيوط في رد الفعل ، فإنها سوف تتجمع في موقع المشغل ، وخلق مجال جديد شبه متجاورة أ * ب *. يمكن لهذا المجال ثم إزاحة القفص عبر TMDSD لبدء النسخ(الشكل 3B). ويمكن توفير هذه الخيوط إما بشكل خارجي أو إنتاجها.
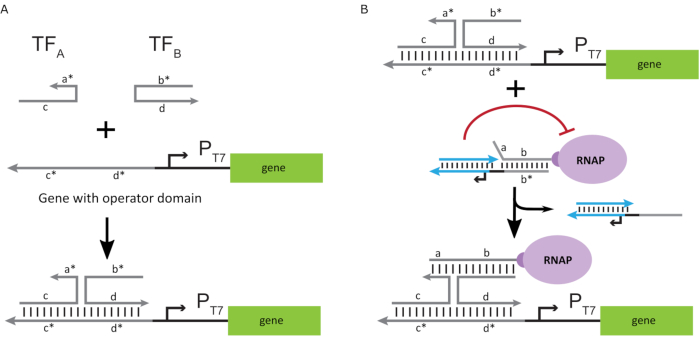
الشكل 3:البرمجة الانتقائية لنشاط البوليميراز من خلال منشط تبديل مكون من ثلاثة مكونات. (أ)عندما تكون عوامل النسخ (TFA و TFB)موجودة ، فإنها ترتبط بمجال المشغل في المنبع للمروج ، مما يشكل تسلسلا زائفا تقطعت به السبل(a *b *) قادرا على إزاحة القفص من خلال إزاحة الحمض النووي بوساطة موطئ القدم. (ب) هذا النطاق * ب * يمكن أن تحل محل القفص عبر TMDSD لبدء النسخ. تم تعديل هذا الرقم من تشو وشيه27. المختصرات: TF = عامل النسخ؛ RNAP = RNA بوليمراز; TMDSD = إزاحة حبلا الحمض النووي بوساطة موطئ القدم. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
استخدام عوامل النسخ المستندة إلى الحمض النووي لتنظيم النسخ في المختبر يسمح للتنفيذ القابل للتطوير لسلوكيات الدوائر المتطورة مثل المنطق الرقمي ، وردود الفعل ، وتتالي الإشارات. على سبيل المثال، يمكن للمرء بناء سلاسل بوابة المنطق عن طريق تصميم تسلسل الحمض النووي بحيث النصوص من الجينات المنبع تنشيط الجينات المصب. أحد التطبيقات التي تستغل المتتالية والمتعددة التي يمكن أن تكون قادرة على هذه التكنولوجيا المقترحة هو تطوير دوائر الحوسبة الجزيئية أكثر تطورا للتشخيص المحمول ومعالجة البيانات الجزيئية. وبالإضافة إلى ذلك، يمكن دمج الحوسبة الجزيئية وقدرات التوليف الحمض النووي الريبي دي نوفو تمكين تطبيقات جديدة. على سبيل المثال، يمكن تصميم الدائرة الجزيئية للكشف عن واحد أو مزيج من الرنانات المعرفة من قبل المستخدم كمدخلات وإخراج الرنانات العلاجية أو mRNAs ترميز الببتيدات الوظيفية أو البروتينات للتطبيقات الطبية نقطة الرعاية.
Protocol
Representative Results
Discussion
توضح هذه الدراسة نهجا مستوحى من تكنولوجيا النانو من الحمض النووي للسيطرة على نشاط T7 RNA polymerase من خلال اقتران مركب T7 RNAP المؤتلف N-terminally SNAP الموسومة ب Oligonucleotide الذي يعمل في BG ، والذي تم استخدامه لاحقا لبرنامج ردود فعل TMDSD. حسب التصميم ، تم وضع علامة SNAP في N-terminus من البوليميراز ، حيث يتم دفن C-terminus م…
Declarações
The authors have nothing to disclose.
Acknowledgements
وتعترف L.Y.T.C بالدعم السخي المقدم من صندوق الحدود الجديدة لاستكشاف البحوث (NFRF-E)، ومجلس أبحاث العلوم الطبيعية والهندسة الكندي (NSERC) منحة ديسكفري، ومبادرة الطب حسب التصميم في جامعة تورنتو، التي تتلقى التمويل من صندوق كندا للتميز البحثي الأول (CFREF).
Materials
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
Referências
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -. J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981 (2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324 (2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -. B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607 (2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C., Doudna, J. A. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Bioquímica. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -. B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).

