DNA系留RNA聚合酶用于可编程体外转录和分子计算
Summary
我们描述了一种新型DNA系留T7 RNA聚合酶的工程设计,以调节体外转录反应。我们讨论了蛋白质合成和表征的步骤,验证了概念验证转录调控,并讨论了其在分子计算、诊断和分子信息处理中的应用。
Abstract
DNA纳米技术使核酸的可编程自组装成用户规定的形状和动态,适用于各种应用。这项工作表明,DNA纳米技术的概念可用于编程噬菌体衍生的T7 RNA聚合酶(RNAP)的酶活性,并构建可扩展的合成基因调节网络。首先,通过表达N末端SNAP标记RNAP以及随后SNAP标签与苄基胍宁(BG)修饰的寡核苷酸的化学偶联来设计寡核苷酸系留的T7 RNAP。接下来,核酸链置换用于按需编程聚合酶转录。此外,辅助核酸组装体可用作”人工转录因子”,以调节DNA程序化的T7 RNAP与其DNA模板之间的相互作用。这种体外转录调节机制可以实现各种电路行为,如数字逻辑、反馈、级联和多路复用。这种基因调控架构的可组合性促进了设计抽象、标准化和缩放。这些功能将使体外遗传设备的快速原型设计成为可能,用于生物传感、疾病检测和数据存储等应用。
Introduction
DNA计算使用一组设计的寡核苷酸作为计算介质。这些寡核苷酸被编程为序列,以根据用户指定的逻辑动态组装并响应特定的核酸输入。在概念验证研究中,计算的输出通常由一组荧光标记的寡核苷酸组成,可以通过凝胶电泳或荧光板读数器进行检测。在过去的30年中,已经证明了越来越复杂的DNA计算电路,例如各种数字逻辑级联,化学反应网络和神经网络1,2,3。为了帮助制备这些DNA回路,数学模型已被用于预测合成基因回路4,5的功能,并且已经开发了用于正交DNA序列设计6,7,8,9,10的计算工具。.与硅基计算机相比,DNA计算机的优势包括它们能够直接与生物分子连接,在没有电源的情况下在溶液中运行,以及它们的整体紧凑性和稳定性。随着下一代测序的出现,合成DNA计算机的成本在过去二十年中一直在以比摩尔定律11更快的速度下降。这种基于DNA的计算机的应用现在开始出现,例如用于疾病诊断12,13,用于为分子生物物理学提供动力14,以及作为数据存储平台15。
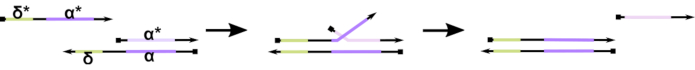
图1:脚趾介导的DNA链位移的机制。 δ,脚趾是部分双工上的自由、未绑定序列。当在第二条链上引入互补域(δ*)时,自由δ域充当杂交的支架,允许链的其余部分(ɑ*)通过称为链迁移的压缩/解压缩可逆反应缓慢地取代其竞争对手。随着δ长度的增加,正向反应的ΔG减小,位移更容易发生。 请点击此处查看此图的放大版本。
迄今为止,大多数DNA计算机都利用动态DNA纳米技术领域公认的基序,称为脚趾介导的DNA链置换(TMDSD,图1)16。该基序由部分双链DNA(dsDNA)双链组成,显示短的”脚趾”悬垂(即7至10个核苷酸(nt))。核酸”输入”链可以通过脚趾与部分双链相互作用。这导致其中一条链从部分双工移出,然后这个释放的链可以作为下游部分双工的输入。因此,TMDSD可实现信号级联和信息处理。原则上,正交TMDSD基序可以在解决方案中独立运行,从而实现并行信息处理。TMDSD反应存在许多变化,例如脚趾介导的DNA链交换(TMDSE)17,具有双长结构域18的”无泄漏”脚趾,序列不匹配的脚趾19和”手持”介导的链位移20。这些创新的设计原则允许更精细地调整TMDSD能量学和动力学,以提高DNA计算性能。
合成基因回路,如转录基因回路,也能够计算21、22、23。这些回路由蛋白质转录因子调节,蛋白质转录因子通过与特定的调节DNA元件结合来激活或抑制基因的转录。与基于DNA的电路相比,转录电路具有几个优点。首先,酶转录比现有的催化DNA回路具有更高的周转率,因此每个输入拷贝产生更多的输出拷贝,并提供更有效的信号放大手段。此外,转录电路可以产生不同的功能分子,如适配子或编码治疗蛋白的信使RNA(mRNA)作为计算输出,可以用于不同的应用。然而,当前转录电路的一个主要限制是它们缺乏可扩展性。这是因为基于正交蛋白质的转录因子集非常有限,并且新蛋白质转录因子的从头设计在技术上仍然具有挑战性且耗时。
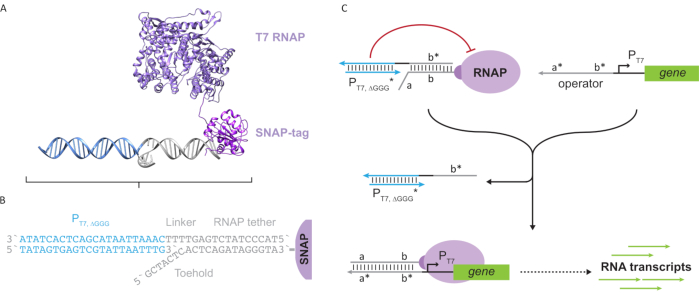
图2:”系绳”和”笼式”聚合酶复合物的提取和机理(A和B)寡核苷酸系绳通过SNAP标签反应酶被酶标记为T7聚合酶。由具有系绳补体悬垂的”人造”T7启动子组成的笼子使其能够与系绳杂交并阻断转录活性。(C)当操作者(a*b*)存在时,它结合到寡核苷酸系绳(ab)上的脚趾并取代笼子的b*区域,从而允许转录发生。这个数字是从Chou和Shih27修改而来的。缩写:RNAP = RNA 聚合酶。请点击此处查看此图的放大版本。
本文介绍了一种用于分子计算的新型构建块,它将转录电路的功能与基于DNA的电路的可扩展性相结合。该构建块是与单链DNA系绳共价连接的T7RNAP(图2A)。为了合成这种DNA系留的T7 RNAP,将聚合酶融合到N端SNAP标签24中并在大肠杆菌中重组表达。然后将SNAP标签与与BG底物功能化的寡核苷酸反应。寡核苷酸系绳允许分子来宾通过DNA杂交定位在靠近聚合酶的位置。其中一位客人是一种被称为”笼子”的竞争性转录阻滞剂,它由一个”人造”T7启动子DNA双链体组成,下游没有基因(图2B)。当通过其寡核苷酸系绳与RNAP结合时,笼子通过与其他DNA模板竞争RNAP结合来阻止聚合酶活性,从而使RNAP处于”OFF”状态(图2C)。
为了将聚合酶激活到”ON”状态,设计了具有基因T7启动子上游的单链”操作员”结构域的T7 DNA模板。操作结构域(即结构域a*b*图2C)可以设计为通过TMDSD将笼子从RNAP取代,并将RNAP定位在基因T7启动子的近端,从而启动转录。或者,还设计了DNA模板,其中操作序列与称为”人工转录因子”的辅助核酸链(即图3A中的TFA和TFB链)互补。当两条链都被引入反应中时,它们将在操作员现场组装,从而创建一个新的伪连续域a*b*。然后,该结构域可以通过TMDSD取代笼子以启动转录(图3B)。这些链可以外源供应或生产。
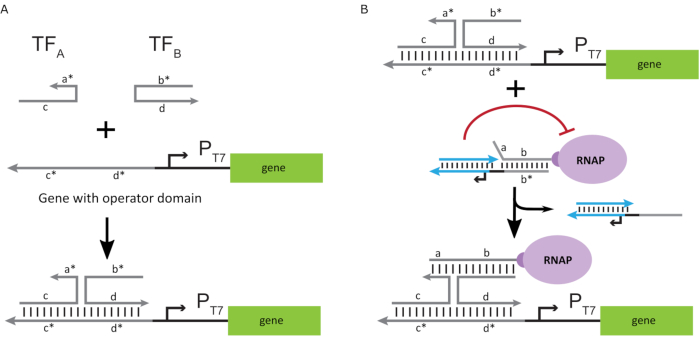
图3:通过三组分开关激活剂对聚合酶活性进行选择性编程。 (A)当转录因子(TFA 和TFB)存在时,它们与启动子上游的操作员结构域结合,形成一个伪单链序列(a*b*),能够通过脚趾介导的DNA置换来置换笼子。(B) 这个 a*b* 结构域可以通过 TMDSD 取代笼子以启动转录。这个数字是从Chou和Shih27修改而来的。缩写: TF = 转录因子;RNAP = RNA聚合酶;TMDSD = 脚趾介导的 DNA 链置换。 请点击此处查看此图的放大版本。
使用基于核酸的转录因子进行体外转录调控允许可扩展地实现复杂的电路行为,如数字逻辑、反馈和信号级联。例如,人们可以通过设计核酸序列来构建逻辑门级联,使得来自上游基因的转录本激活下游基因。利用该技术所提出的级联和多路复用能力的一个应用是开发更复杂的分子计算电路,用于便携式诊断和分子数据处理。此外,集成分子计算和从头RNA合成功能可以实现新的应用。例如,分子电路可以设计用于检测用户定义的RNA的一种或组合,作为输入和输出治疗性RNA或mRNA编码功能肽或蛋白质的mRNA,用于床旁医疗应用。
Protocol
Representative Results
Discussion
本研究展示了一种受DNA纳米技术启发的方法,通过N末端SNAP标记的重组T7 RNAP与BG功能化的寡核苷酸共价耦合来控制T7 RNA聚合酶的活性,随后用于编程TMDSD反应。根据设计,SNAP标签位于聚合酶的N端,因为野生型T7 RNAP的C端埋在蛋白质结构核心内并与DNA模板28进行重要接触。先前修改聚合酶C末端的尝试已导致酶活性完全丧失,除非引入其他补偿性突变。29,30</sup…
Declarações
The authors have nothing to disclose.
Acknowledgements
L.Y.T.C感谢来自研究探索新前沿(NFRF-E),加拿大自然科学与工程研究委员会(NSERC)发现补助金以及多伦多大学医学设计倡议的慷慨支持,该倡议获得加拿大第一研究卓越基金(CFREF)的资助。
Materials
| 0.5% polysorbate 20 (TWEEN 20) | BioShop | TWN510.5 | |
| 0.5M ethylenediaminetetraacetic acid (EDTA) | Bio Basic | SD8135 | |
| 10 mM sodium phosphate buffer (pH 7) | Bio Basic | PD0435 | Tablets used to make 10 mM buffer |
| 10% ammonium persulfate (APS) | Sigma Aldrich | A3678-100G | |
| 100 kDa Amicon Ultra-15 Centrifugal Filter Unit | Fisher Scientific | UFC910008 | |
| 100% acetone | Fisher Chemical | A18P4 | |
| 100% ethanol (EtOH) | House Brand | 39752-P016-EAAN | |
| 10x in vitro transcription (IVT) buffer | New England Biolabs | B9012 | |
| 10x Tris-Borate-EDTA (TBE) buffer | Bio Basic | A0026 | |
| 1M Isopropyl β- d-1-thiogalactopyranoside (IPTG) | Sigma Aldrich | I5502-1G | |
| 1M sodium bicarbonate buffer | Sigma Aldrich | S6014-500G | |
| 1M Tris(hydroxymethyl)aminomethane (Tris) | Sigma Aldrich | 648311-1KG | |
| 1X Tris-EDTA (TE) buffer | ThermoFisher | 12090015 | |
| 2M imidazole | Sigma Aldrich | 56750-100G | |
| 2-mercaptoethanol (BME) | Sigma Aldrich | M3148 | |
| 3M sodium acetate | Bio Basic | SRB1611 | |
| 40% acrylamide (19:1) | Bio Basic | A00062 | |
| 4x LDS protein sample loading buffer | Fisher Scientific | NP0007 | |
| 5M sodium chloride (NaCl) | Bio Basic | DB0483 | |
| 5mM dithiothreitol (DTT) | Sigma Aldrich | 43815-1G | |
| 6x gel loading dye | New England Biolabs | B7024S | |
| agarose B powder | Bio Basic | AB0014 | |
| BG-GLA-NHS | New England Biolabs | S9151S | |
| BL21 competent E. coli | Addgene | C2530H | |
| BLUeye prestained protein ladder | FroggaBio | PM007-0500 | |
| bromophenol blue | Bio Basic | BDB0001 | |
| coomassie blue (SimplyBlue SafeStain) | ThermoFisher | LC6060 | |
| cyanine dye (SYBR Gold nucleic acid gel stain) | Fisher Scientific | S11494 | |
| cyanine dye (SYBR Safe nucleic acid gel stain) | Fisher Scientific | S33102 | |
| dry dimethyl sulfoxide (DMSO) | Fisher Scientific | D12345 | |
| formamide | Sigma Aldrich | F9037-100ML | |
| glycerol | Bio Basic | GB0232 | |
| kanamycin sulfate | BioShop | KAN201.5 | |
| lysogeny broth | Sigma Aldrich | L2542-500ML | |
| malachite green oxalate | Sigma Aldrich | 2437-29-8 | |
| N,N,N'N'-Tetramethylethane-1,2-diamine (TEMED) | Sigma Aldrich | T9281-25ML | |
| NuPAGE MES SDS running buffer (20x) | Fisher Scientific | LSNP0002 | |
| NuPAGE Novex 4-12% Bis-Tris gel 1.0 mm 12-well | Life Technologies | NP0322BOX | |
| oligonucleotide (cage antisense) | IDT | N/A | TATAGTGAGTCGTATTAATTTG |
| oligonucleotide (cage sense) | IDT | N/A | TCAGTCACCTATCTGTTTCAAA TTAATACGACTCACTATA |
| oligonucleotide (malachite green aptamer antisense) | IDT | N/A | GGATCCATTCGTTACCTGGCT CTCGCCAGTCGGGATCCTATA GTGAGTCGTATTACAGTTCCAT TATCGCCGTAGTTGGTGTACT |
| oligonucleotide (malachite green aptamer sense) | IDT | N/A | TAATACGACTCACTATAGGATC CCGACTGGCGAGAGCCAGGT AACGAATGGATCC |
| oligonucleotide (Transcription Factor A) | IDT | N/A | AGTACACCAACTACGAGTGAG |
| oligonucleotide (Transcription Factor B) | IDT | N/A | TCAGTCACCTATCTGGCGATAA TGGAACTG |
| oligonucleotide with 3’ Amine modification (tether) | IDT | N/A | GCTACTCACTCAGATAGGTGAC TGA/3AmMO/ |
| Pierce strong ion exchange spin columns | Fisher Scientific | 90008 | |
| plasmid encoding SNAP T7 RNAP and kanamycin resistance genes | Genscript | N/A | custom gene insert |
| protein purification column (HisPur Ni-NTA spin column) | Fisher Scientific | 88226 | |
| rNTP mix | New England Biolabs | N0466S | |
| Roche mini quick DNA spin column | Sigma Aldrich | 11814419001 | |
| Triton X-100 | Sigma Aldrich | T8787-100ML | |
| Ultra Low Range DNA ladder | Fisher Scientific | 10597012 | |
| urea | BioShop | URE001.1 |
Referências
- Cherry, K. M., Qian, L. Scaling up molecular pattern recognition with DNA-based winner-take-all neural networks. Nature. 559 (7714), 370-376 (2018).
- Qian, L., Winfree, E., Bruck, J. Neural network computation with DNA strand displacement cascades. Nature. 475 (7356), 368-372 (2011).
- Chen, Y. -. J., et al. Programmable chemical controllers made from DNA. Nature Nanotechnology. 8 (10), 755-762 (2013).
- di Bernardo, D., Marucci, L., Menolascina, F., Siciliano, V. Predicting synthetic gene networks. Synthetic Gene Networks: Methods and Protocols. 813, 57-81 (2012).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Gould, N., Hendy, O., Papamichail, D. Computational tools and algorithms for designing customized synthetic genes. Frontiers in Bioengineering and Biotechnology. 2, (2014).
- MacDonald, J. T., Siciliano, V. Computational sequence design with R2oDNA Designer. Mammalian Synthetic Promoters. 1651, 249-262 (2017).
- Cervantes-Salido, V. M., Jaime, O., Brizuela, C. A., Martínez-Pérez, I. M. Improving the design of sequences for DNA computing: A multiobjective evolutionary approach. Applied Soft Computing. 13 (12), 4594-4607 (2013).
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. Journal of Computational Chemistry. 32 (1), 170-173 (2011).
- Fornace, M. E., Porubsky, N. J., Pierce, N. A. A unified dynamic programming framework for the analysis of interacting nucleic acid strands: enhanced models, scalability, and speed. ACS Synthetic Biology. 9 (10), 2665-2678 (2020).
- Wetterstrand, K. DNA sequencing costs: Data. Genome.gov. , (2020).
- Lopez, R., Wang, R., Seelig, G. A molecular multi-gene classifier for disease diagnostics. Nature Chemistry. 10 (7), 746-754 (2018).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Yurke, B., Turberfield, A. J., Mills, A. P., Simmel, F. C., Neumann, J. L. A DNA-fuelled molecular machine made of DNA. Nature. 406 (6796), 605-608 (2000).
- Lin, K. N., Volkel, K., Tuck, J. M., Keung, A. J. Dynamic and scalable DNA-based information storage. Nature Communications. 11 (1), 2981 (2020).
- Yurke, B., Mills, A. P. Using DNA to power nanostructures. Genetic Programming and Evolvable Machines. 4 (2), 111-122 (2003).
- Zhang, D. Y., Turberfield, A. J., Yurke, B., Winfree, E. Engineering entropy-driven reactions and networks catalyzed by DNA. Science. 318 (5853), 1121-1125 (2007).
- Wang, B., Thachuk, C., Ellington, A. D., Winfree, E., Soloveichik, D. Effective design principles for leakless strand displacement systems. Proceedings of the National Academy of Sciences. 115 (52), 12182-12191 (2018).
- Machinek, R. R. F., Ouldridge, T. E., Haley, N. E. C., Bath, J., Turberfield, A. J. Programmable energy landscapes for kinetic control of DNA strand displacement. Nature Communications. 5 (1), 5324 (2014).
- Cabello-Garcia, J., Bae, W., Stan, G. -. B. V., Ouldridge, T. E. Handhold-mediated strand displacement: a nucleic acid-based mechanism for generating far-from-equilibrium assemblies through templated reactions. bioRxiv. , (2020).
- Brophy, J. A. N., Voigt, C. A. Principles of genetic circuit design. Nature Methods. 11 (5), 508-520 (2014).
- Khalil, A. S., et al. A synthetic biology framework for programming eukaryotic transcription functions. Cell. 150 (3), 647-658 (2012).
- Swank, Z., Laohakunakorn, N., Maerkl, S. J. Cell-free gene-regulatory network engineering with synthetic transcription factors. Proceedings of the National Academy of Sciences. 116 (13), 5892-5901 (2019).
- Howland, S. W., Tsuji, T., Gnjatic, S., Ritter, G., Old, L. J., Wittrup, K. D. Inducing efficient cross-priming using antigen-coated yeast particles. Journal of immunotherapy. 31 (7), 607 (2008).
- Abil, Z., Ellefson, J. W., Gollihar, J. D., Watkins, E., Ellington, A. D. Compartmentalized partnered replication for the directed evolution of genetic parts and circuits. Nature Protocols. 12 (12), 2493-2512 (2017).
- Baugh, C., Grate, D., Wilson, C., Doudna, J. A. 2.8 Å crystal structure of the malachite green aptamer11. Journal of Molecular Biology. 301 (1), 117-128 (2000).
- Chou, L. Y. T., Shih, W. M. In vitro transcriptional regulation via nucleic acid-based transcription factors. ACS Synthetic Biology. 8 (11), 2558-2565 (2019).
- Lykke-Andersen, J., Christiansen, J. The C-terminal carboxy group of T7 RNA polymerase ensures efficient magnesium ion-dependent catalysis. Nucleic Acids Research. 26 (24), 5630-5635 (1998).
- Pu, J., Disare, M., Dickinson, B. C. Evolution of C-terminal modification tolerance in full-length and split T7 RNA Polymerase biosensors. Chembiochem. 20 (12), 1547-1553 (2019).
- Gardner, L. P., Mookhtiar, K. A., Coleman, J. E. Initiation, elongation, and processivity of carboxyl-terminal mutants of T7 RNA polymerase. Bioquímica. 36 (10), 2908-2918 (1997).
- Yin, J., Lin, A. J., Golan, D. E., Walsh, C. T. Site-specific protein labeling by Sfp phosphopantetheinyl transferase. Nature Protocols. 1 (1), 280-285 (2006).
- Warden-Rothman, R., Caturegli, I., Popik, V., Tsourkas, A. Sortase-tag expressed protein ligation: combining protein purification and site-specific bioconjugation into a single step. Analytical Chemistry. 85 (22), 11090-11097 (2013).
- Zhang, W. -. B., Sun, F., Tirrell, D. A., Arnold, F. H. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. Journal of the American Chemical Society. 135 (37), 13988-13997 (2013).

