PMS2 carente, ERCC1, Ku86, CcOI in Difetti del campo durante la progressione a tumore del colon
Summary
Ridotta espressione / assenza di PMS2 e / o ERCC1 nelle cripte intero è un evento frequente nel raggio di 10 cm su ogni lato degli adenocarcinomi del colon, probabilmente in base ad un difetto del campo con elevata mutevolezza e la progressione del cancro. Carenza di Ku86 o CcOI è molto meno frequente in questi difetti del campo.
Abstract
Nella carcinogenesi, il "difetto di campo" è riconosciuto clinicamente a causa della propensione dei sopravvissuti di alcuni tumori per sviluppare altri tumori maligni dello stesso tipo di tessuto, spesso in zona. Difetti del campo quali sono stati indicati nel cancro del colon. Le alterazioni molecolari che sono responsabili di un difetto del campo nei due punti dovrebbero essere rilevabili ad alta frequenza nel tessuto istologicamente normale che circonda un adenocarcinoma del colon o circonda un adenoma con neoplasia avanzata (sulla buona strada per un cancro al colon), ma a bassa frequenza nella mucosa del colon di pazienti senza neoplasia del colon.
Utilizzando immunoistochimica, cripte intero entro 10 cm su ogni lato degli adenocarcinomi del colon o avanzato neoplasie del colon sono risultate spesso ridotta o assente espressione di due proteine di riparazione del DNA, PMS2 e / o ERCC1. PMS2 è una proteina duplice ruolo, attivo nella riparazione del DNA mancata corrispondenza così come necessario in apoptosi delle cellule con danno al DNA in eccesso. ERCC1 è attiva in riparazione per escissione dei nucleotidi del DNA. L'espressione ridotta o assente di entrambi ERCC1 e PMS2 avrebbe creato cellule sia con maggiore capacità di sopravvivere (resistenza apoptosi) e aumento del livello di mutabilità. L'espressione ridotta o assente di entrambi ERCC1 e PMS2 è probabile che un primo passo nella progressione del cancro del colon.
Gene di riparazione del DNA Ku86 (attivo nel DNA non omologhe fine giunzione) e citocromo c ossidasi subunità I (coinvolti in apoptosi) erano stati entrambi segnalati per essere ridotta a espressione nelle aree vicino ai tumori della mucosa del colon. Tuttavia, la valutazione immunoistochimica dei livelli di espressione hanno mostrato solo a basse frequenze modeste di cripte ad essere carenti nella loro espressione in un difetto del campo che circonda il cancro del colon o circostanti neoplasia del colon avanzato.
Mostriamo, qui, il nostro metodo di valutazione delle cripte per l'espressione di ERCC1, PMS2, Ku86 e CcOI. Abbiamo dimostrato che la frequenza delle cripte tutto carenti per PMS2 e ERCC1 è spesso così grande come il 70% al 95% nel 20 centimetri lungo le zone circostanti una neoplasia del colon, mentre la frequenza delle cripte carente in Ku86 ha un valore medio del 2% e la frequenza delle cripte carenti in CcOI ha un valore mediano del 16% in questi settori. Tutto il colon è di 150 cm di lunghezza (circa 5 metri) e dispone di circa 10 milioni di cripte nel suo strato mucoso. Il difetto di PMS2 e ERCC1 che circondano un tumore del colon in tal modo possono includere milione cripte. E 'da una cripta difettoso che il cancro al colon deriva.
Protocol
Discussion
- E 'molto importante avere 3 sezioni di tessuto per vetrino. Le bolle possono verificarsi quando si applica i media portando l'anticorpo, in modo che alcune cripte o porzioni di cripte non può essere colorato in modo adeguato solo perché una bolla impedito l'anticorpo di reagire con la sezione di tessuto. Cripte sono circa 60 micron di diametro, e sezioni di tessuto sono 4 micron di spessore, quindi ci potrebbero essere ben 15 sezioni di tessuto attraverso la cripta stessa. Di solito è possibile trovare la cripta stessa, con morfologia della cripta come una guida, su sezioni di tessuto più posto su un vetrino.
- Nei nostri studi recenti, per i campioni di tessuto di grandi dimensioni, in una colorazione Sequenza non può dare una buona copertura anticorpale al tessuto. In questi casi, si può preferire di usare la mano colorazione del tessuto, dove una goccia di supporto contenente l'anticorpo è a mano posta sopra ogni campione di tessuto.
- Di vedere chiaramente la colorazione marrone che mostra l'espressione della proteina immunostained, e la colorazione blu che mostra DNA nucleare sul monitor del computer, il software photomicroscope Motic è regolata come segue: Guadagno 0, offset 0, Enhance 0, 255, Gamma 1.00, 0, Nitidezza 1, correzione del colore 7, R, G, B Gain 1.00, e R, G, Luminosità B 0. La risoluzione è impostata a 1024 x 768, e del bilanciamento del bianco è abilitata.
- Il nostro metodo può essere utilizzato con qualsiasi proteine ritenute importanti nella progressione del cancro del colon, per valutare la loro frequenza di carenza, o forse l'iperespressione, a difetti del campo che circonda i tumori del colon o adenomi con neoplasia in fase avanzata.
- Difetti del campo sono una parte di sviluppo del cancro
Il termine "campo di cancerizzazione" è stata la prima volta da Slaughter et al. Nel 1953 da 1 a descrivere un'area o "campo" di epitelio che è stato condizionato dai processi in gran parte sconosciuto (al momento) in modo da predisporre per lo sviluppo del cancro. L'utilizzo iniziale è stata, nel contesto di tumori orali. Da allora, il "campo di cancerizzazione" e "difetto di campo" sono state utilizzate più ampiamente per descrivere qualsiasi pre-neoplastiche del tessuto in cui nuovi casi di tumore hanno maggiori probabilità di verificarsi, e il concetto di cancerizzazione di campo in oncologia clinica ha ricevuto un'attenzione crescente 2 . Recentemente abbiamo esaminato le evidenze di difetti del campo nel cancro gastrointestinale 3. Inclusi in questa revisione sono stati i risultati di una dozzina di studi fornire la prova di difetti del campo nel colon.
Difetti del campo nella mucosa del colon probabilmente derivano dalla selezione naturale delle cellule mutanti o cellule alterate epigeneticamente tra le cellule staminali di una cripta in modo che una cellula staminale sopravvive successione 4 di nicchia. Instabilità genetica o di un fenotipo mutatore, forse a causa della riduzione di riparazione per escissione dei nucleotidi del DNA o DNA mismatch repair, dovrebbe accelerare questo processo (e un difetto frequente nei PMS2 era precedentemente rilevato nelle zone circostanti i tumori del colon 5). Se, in una popolazione normale di cellule staminali nella mucosa del colon, una cellula acquisisce un vantaggio selettivo attraverso una mutazione o un epimutation, tenderà ad espandersi clonale a spese delle cellule staminali vicini. Nella mucosa del colon, la nicchia delle cellule staminali è occupato da ≤ 5 celle 6 che poi danno luogo a tutti (circa 2.000) delle cellule della cripta. Una acquisizione della nicchia delle cellule staminali da un risultato cellula mutante o epigeneticamente alterati tipo di "conversione cripta" 7.
La diffusione di cloni mutato (cripte convertito) nell'epitelio del colon può più spesso si verifica per fissione cripta 8. Un esempio di fissione cripta è illustrato nella figura 2. In questo modo una zona di tessuto anomalo si pone. All'interno di tale patch, una seconda mutazione quali possono verificarsi in modo che una cripta dato acquisisce un vantaggio rispetto ad altre cripte all'interno della patch, e questa cripta si espanderà clonale formando una patch secondaria all'interno della patch originale. All'interno di questa nuova patch, il processo può essere ripetuto più tempo diversi, forse per decenni, fino a quando una cellula staminale maligno che si espande nasce clonale in un cancro. Se questo è il processo generale attraverso il quale sporadici adenocarcinomi del colon sorgono, poi adenocarcinomi del colon in genere devono essere associati, ed essere preceduto da, campi di anormalità. Così un pezzo di cripte convertito ci si potrebbe aspettare di avere difetti rilevabili nell'area circostante un adenoma con neoplasia avanzata, o attorno ad un cancro del colon.
Da questo modello di pre-progressione del tumore, ci si aspetterebbe di trovare alterazioni genetiche o epigenetiche relative al pre-tumorali progressione tra i campioni di tessuto prelevati dal non-neoplastici mucosa del colon nelle aree circostanti le lesioni neoplastiche del colon che possono progredire a cancro . In particolare, i difetti degli enzimi di riparazione del DNA o delle proteine necessarie per l'apoptosi, contribuirebbe alla progressione aumentando l'instabilità genomica, e ci si potrebbe aspettare di trovare in un campo difettoso. - Selezione per carenza di PMS2 quando le cellule sono deficient per ERCC1 e sottoposti ad un agente che danneggiano il DNA
Nara et al. 9 sono cresciute del 25 colture di cellule ovariche di criceto cinese 43-3B, tutti con una mutazione inattivando il gene di riparazione del DNA ERCC1, le culture e trattati con un agente che danneggiano il DNA, MNNG. Tra le cellule sopravvissute al trattamento danni al DNA, una singola cellula è stato isolato da ogni cultura e ha permesso di formare un clone. Tutti i cloni isolati sono stati circa 10 volte più resistente di aver ucciso dai MNNG rispetto al ceppo parentale. Dei cloni isolati, 22 sono stati dimostrato di essere difettoso in riparazione mancata corrispondenza del DNA (MMR). Cinque di questi 22 cloni sono stati analizzati mediante sequenziamento, e tre dei cinque sono stati carenti per PMS2. Questa selezione per difetto PMS2 era presumibilmente perché PMS2 deficit causato resistenza all'apoptosi, e quindi la capacità di sopravvivere al danno al DNA nelle cellule aggiunto ancora quando ERCC1 era assente. (Normalmente, PMS2 sensi livelli di danno al DNA, e induce le cellule per apoptosi quando danno al DNA è eccessivo.) Così, una carenza di PMS2 è stato selezionato per quando ERCC1-deficienti cellule sono sottoposte a danno al DNA. Cloni difettosi sia ERCC1 e PMS2 hanno mostrato di avere su un 500 a 1000 volte maggiore tasso di mutazione in caso di irradiazione con raggi UV rispetto alle cellule parentali. - Acidi biliari causano stress ossidativo, sono agenti che danneggiano il DNA e sono importanti nella progressione del cancro del colon
Desossicolico acido, un acido biliare, aumenta lo stress ossidativo attraverso la generazione di specie reattive dell'ossigeno (ROS) da più processi, compresi danneggiamento dei mitocondri 10. Vi è stato recentemente dimostrato che ROS causano cambiamenti epigenetici 11 e almeno 15 studi hanno dimostrato che gli acidi biliari, presso l'aumento delle concentrazioni che accompagna una dieta ricca di grassi, causa danni al DNA 12. Come recensione da Bernstein et al. 12, vi è una correlazione positiva tra il consumo di grassi nella dieta e incidenza del cancro del colon. Acidi biliari sono secreti nel tratto intestinale in risposta al grasso nella dieta, e gli acidi biliari agiscono come detergenti per facilitare la digestione dei grassi. Quindi, una dieta ricca di grassi è associata ad un aumento della secrezione della bile acida. Fecale concentrazioni di acidi biliari sono elevati in popolazioni con un'alta incidenza di cancro al colon, e l'eccessiva esposizione agli acidi biliari è considerata come un importante fattore di rischio per la carcinogenesi del colon 13. Acidi biliari può essere una causa di difetti del campo nel colon.
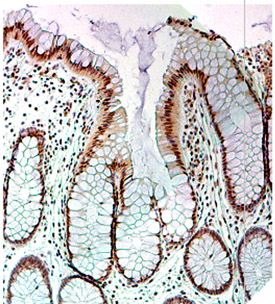
Figura 2. Fissione Una cripta del colon in 3 cripte.
Conclusione
Abbiamo trovato una frequenza elevata di cripte carente nei geni di riparazione del DNA e ERCC1 PMS2 (PMS2 è necessario anche per apoptosi) in difetti del campo che circonda i tumori del colon. Carenze di ERCC1 e PMS2 (a causa di cambiamenti epigenetici o mutazioni) possono essere primi passi nel instabilità genetica centrale di sviluppo del cancro del colon.
Acknowledgements
Finanziamento concesso dalla Southern Arizona Veterans Affairs Health Care sistema VA Merito Recensione concedere 0142 e Arizona Ricerca Biomedica Commissione concede 0803.
Materials
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| PMS2 antibody | BD Biosciences | 556415 | ||
| ERCC1 antibody | Thermo Fisher | MS-671-P1 | ||
| Ku86 antibody | Santa Cruz Biotech. | SC-9034 | ||
| CcO1 antibody | Invitrogen | 459600 | ||
| Biotinylated secondary | DAKO | E0413 | ||
| 22% BSA | Informagen, Inc. | 2327 | ||
| Sniper | Biocare Medical | BS966 | ||
| Vectastain ABC | Vector Labs | PK-6100 | ||
| DAB | Thermo Scientific | 34001 | ||
| Tween 20 | USB | 20605 | ||
| Cytoseal XYL | VWR | 48212-196 | ||
| Trizma | Sigma | T1503 |
References
- Slaughter, D. P., Southwick, H. W., Smejkal, W. Field cancerization in oral stratified squamous epithelium; clinical implications of multicentric origin. Cancer. 6, 963-968 (1953).
- Vauthey, J. N. Importance of field cancerisation in clinical oncology. Lancet Oncol. 1, 15-16 (2000).
- Bernstein, C. Field defects in progression to gastrointestinal tract cancers. Cancer Lett. 260, 1-10 (2008).
- Calabrese, P. Colorectal pretumor progression before and after loss of DNA mismatch repair. Am J Pathol. 164, 1447-1453 (2004).
- Bernstein, H. Reduced Pms2 expression in non-neoplastic flat mucosa from patients with colon cancer correlates with reduced apoptosis competence. Appl Immunohistochem Mol Morphol. 14, 166-172 (2006).
- Huang, E. H. Aldehyde dehydrogenase 1 is a marker for normal and malignant human colonic stem cells (SC) and tracks SC overpopulation during colon tumorigenesis. Cancer Res. 69, 3382-3389 (2009).
- Kim, K. M., Shibata, D. Methylation reveals a niche: stem cell succession in human colon crypts. Oncogene. 21, 5441-5449 (2002).
- Greaves, L. C. Mitochondrial DNA mutations are established in human colonic stem cells, and mutated clones expand by crypt fission. Proc Natl Acad Sci USA. 103, 714-719 (2006).
- Nara, K., Nagashima, F., Yasui, A. Highly elevated ultraviolet-induced mutation frequency in isolated Chinese hamster cell lines defective in nucleotide excision repair and mismatch repair proteins. Cancer Res. 61, 50-52 (2001).
- Payne, C. M. Deoxycholate induces mitochondrial oxidative stress and activates NF-kB through multiple mechanisms in HCT-116 colon epithelial cells. Carcinogenesis. 28, 215-222 (2007).
- Lim, S. O. Epigenetic changes induced by reactive oxygen species in hepatocellular carcinoma: methylation of the E-cadherin promoter. Gastroenterology. 135, 2128-2140 (2008).
- Bernstein, H. Bile acids as carcinogens in human gastrointestinal cancers. Mutat Res. 589, 47-65 (2005).
- Payne, C. M. hydrophobic bile acids, genomic instability, Darwinian selection, and colon carcinogenesis. Clinical and Experimental Gastroenterology. 1, 19-47 (2008).

