荧光各向异性作为一种工具来研究蛋白质相互作用
Summary
蛋白质相互作用是在一个细胞的功能的心脏。量热和光谱技术通常用于表征它们。这里我们介绍荧光各向异性作为一种工具来研究在Shwachman-Diamond综合征(SBDS)突变蛋白质和延伸因子样1 GTP酶(EFL1)之间的相互作用。
Abstract
蛋白质 – 蛋白质相互作用发挥活生物体的功能中起重要作用。一旦相互作用已被确定和验证是必要在结构和机械水平来表征它。几个生物化学和生物物理方法用于此目的存在。其中,荧光各向异性,特别使用一种强大的技术,当荧光团标记蛋白质的荧光强度保持在蛋白质 – 蛋白质相互作用常数。在该技术中,荧光团标记的蛋白是激发选择性地激发根据其与入射光束的相对取向的荧光团的子集的适当波长的垂直偏振光。所得发射还具有方向性,其在垂直和水平平面关系定义各向异性(r)的如下:R =(I VV -I V H)/(I VV + 2I V H),其中I VV和我<sUB> VH是垂直和水平分量的荧光强度分别。荧光各向异性是荧光团,即连接于蛋白,它是建立在蛋白质 – 蛋白质相互作用改变荧光团的表观分子大小的旋转扩散敏感。在本文中,使用荧光各向异性作为一种工具来研究蛋白质相互作用是例证,以解决Shwachman-Diamond综合征(SBDS)突变蛋白质和延伸因子样-1 GTP酶(EFL1)之间的绑定。传统上,用荧光团的蛋白质的标记上的硫醇基(半胱氨酸)或在蛋白质的氨基(N-末端胺或赖氨酸)进行。然而,SBDS拥有多项半胱氨酸和赖氨酸并没有让现场指挥它的标签。作为一种替代技术,染料4',5'-双(1,3,2- dithioarsolan -2-基)荧光素用于特异性标记一个四半胱氨酸基序,Cys-的Cys-Pro的基-Gly-半胱氨酸 – 半胱氨酸,在重组SBDS蛋白的C末端遗传工程。拟合实验数据提供了关于这些蛋白质之间的结合模式的数量和机械信息。
Introduction
细胞含有生物大分子是不断地与彼此交互的一许多。该协会产生了参与负责其在信号转导,基因表达和除其他细胞迁移的调节功能的细胞通路的复合物。发生在一个小区中的所有蛋白质 – 蛋白质相互作用包括称为相互作用组的网络。在酿酒酵母中的蛋白质的70%以上已被证明有相互作用伙伴1。理解的细胞的相互作用组和它们的功能提供关于复杂性和生物多样性的相关信息。数种方法已经描述于鉴定和表征蛋白质 – 蛋白质相互作用。不同的高通过放的方法,如酵母双杂交2,蛋白质片段互补测定法如图3所示 ,耦合到质谱和蛋白microarra亲和纯化4YS用于识别交互5,6。一旦确定,就必须对其进行验证,这可能在逐案的基础而改变。通常,这些实验涉及破坏相互作用本身在该相互作用对, 例如的个别成员的水平,通过基因缺失或蛋白之一的表达,然后寻找在属性或改变的其它部件的功能细胞水平。随后,生物物理技术7用于在分子水平来表征蛋白质-蛋白质相互作用。为此,虽然量热法和荧光光谱被用于定量和机械地描述它们的蛋白质复合物的结构通过X射线晶体学,核磁共振和低温电子显微镜来确定。
在这项工作中,荧光各向异性用作技术来表征GTP酶EFL1和SBD之间的相互作用S蛋白。这些蛋白质通过来自60S核糖体亚基8的表面促进真核起始因子6的释放参与核糖体的合成。该SBDS蛋白在称为Shwachman-Diamond综合征9和充当EFL1减小为鸟苷二磷酸10,11其亲和性的鸟嘌呤核苷酸交换因子的疾病的突变。在SBDS疾病的突变废除与EFL1的相互作用,从而防止其激活。
荧光各向异性在生物应用通常用于研究蛋白质 – 肽或蛋白质 – 核酸相互作用。它是基于荧光团与偏振光产生一个部分偏振的发射激发的原理。荧光各向异性由等式1定义:
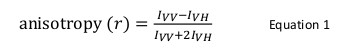
在那里我VV和VH我是垂直(VV)和水平(V H)的荧光强度偏振的发射时,样品被激发与垂直偏振光12。荧光各向异性是影响荧光团的旋转扩散速度的因素敏感,因而取决于温度,溶液的粘度和荧光团的表观分子大小。含有荧光团的增加,当它与另一蛋白和这种变化相互作用的蛋白质的表观大小然后可以作为各向异性的变化进行评估。更具体地说,在相对于它的荧光寿命溶液缓慢旋转的荧光团将有一个大I VV值和小我的VH的值,因此,将表现出相对大的各向异性。对于迅速相对于它们的荧光寿命滚入荧光团,我VV和VH我将类似和各向异性值将是12小</sup>( 图1)。此外,有一个良好的各向异性信号到噪声测量,有必要有一个与相似于感兴趣的分子的旋转相关时间荧光寿命的荧光团。否则,就不可能准确地记录游离蛋白在复之间以及在各向异性的差异。例如,荧光探针与接近4纳秒如荧光素或若丹明附着到100道尔顿的低分子量化合物一生各向异性为0.05。绑定到160 kDa的将其各向异性值提高到0.29的分子;可精确地测量的差。与此相反,参与结合反应,其增加的分子大小而变化,从65至1000 kDa的相同荧光探针只会造成0.28各向异性变化到0.3,这是太小而不能精确测量。在这种情况下,用400纳秒的使用寿命的探针会更适合12。
<p类=“jove_content”>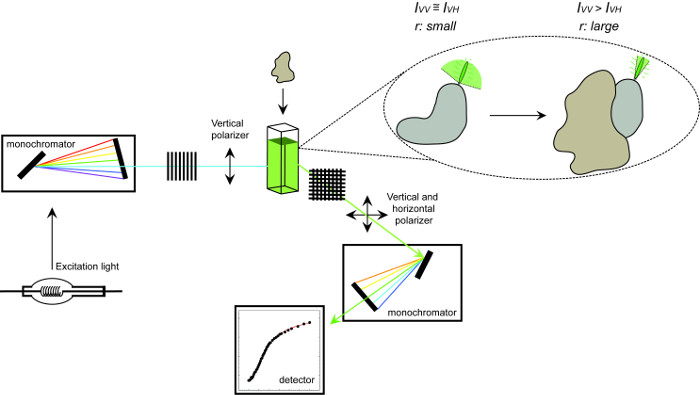
图1.用于测量荧光各向异性和过程的设备的示意图。用于执行蛋白质-蛋白质相互作用的实验测量荧光各向异性的设备的示意图。滚入快速显示小各向异性结合后相互作用的合作伙伴增加了荧光。 请点击此处查看该图的放大版本。
荧光应用需要荧光团在任何所研究的分子的存在。研究蛋白质 – 蛋白质相互作用有三种类型的荧光团:1)中存在的蛋白质中的色氨酸残基,2)化学连接的荧光团和3)荧光融合伙伴,例如绿色荧光蛋白(GFP)及其deriva表3-6。大多数蛋白质具有在其结构色氨酸残基,从而来测量相互作用的最简单的方法是通过监视在相应的荧光光谱的变化或通过在色氨酸残基的荧光强度监视更改。然而,色氨酸残基可存在于两种蛋白质的分析复杂化。另一方面,对于荧光团以改变其荧光性质由于交互它需要被位于或结合位点附近,它可以与交互本身干涉。使用笨重的荧光,如GFP时,这种需要特别注意。如果没有这些荧光团可用于结合研究是必要的,那么,引入外源性荧光团所涉及的蛋白之一。许多化学合成的荧光团存在,并且可被共价连接到通过其反应性基团的蛋白质,如胺基(赖氨酸或N末端的侧链)和在半胱氨酸的硫醇基团。 F与异硫氰酸酯和琥珀酰亚胺酯luorophore衍生物与酰胺基团反应而碘乙酰胺和马来酰亚胺是硫醇反应性基团13。在荧光应用中最常用的染料是荧光素的衍生物,和若丹明绿染料,香豆素,BODIPY荧光团和Alexa Fluor染料。市售的荧光团及其使用的详细列表可在参考文献14,15找到。对于成功的标记,反应性基团必须在蛋白质的表面上暴露出来,但由于大量通常存在于多肽的反应性官能团的是很难得到的位点特异性修饰。在这项研究的目的蛋白质,SBDS,含有5游离半胱氨酸和33的赖氨酸,可能导致在多个位点标记。非均匀标签可能影响结合和不同的荧光分子可能结合后引起不同的荧光强度信号将复杂的数据分析。奥雅纳rcome这一问题,我们使用了闪光灯荧光团,4',5'-双(1,3,2- dithioarsolan -2-基)荧光素到网站直接标记的SBDS蛋白。这是一arsenoxide染料与在一个基序知道作为由序列CCXXCC,其中X是除半胱氨酸16,17之外的任何氨基酸的闪光标记为四个间隔半胱氨酸的高亲和性。这四半胱氨酸基序被添加到通过遗传工程N-或蛋白质的C末端与适当的接头,以防止蛋白质的总体折叠的破坏在一起。由闪光染料和Flash标签的一对最初设计为位点特异性标签的蛋白在活细胞中17,但它也可以使用,因为它是这里例举标记纯化的蛋白在体外 。此外,酶的策略也已经被开发,以使蛋白18的位点特异性官能化。
在这个手稿中,我们描述的荧光各向异性的用处山工具来研究蛋白质 – 蛋白质相互作用。结合可以通过结合曲线形状的简单检查来评估而定量信息可以从实验数据的拟合而获得。
Protocol
Representative Results
Discussion
与蛋白质大多数生化实验不仅需要纯的蛋白质,而且大量的它们,而不管使用的技术。由于这个原因,用于这种类型的实验的蛋白质被异源表达得到,因为它是这里提出的情况。花期光谱需要荧光团的研究分子中的存在。芳香族残基构成的蛋白质的固有荧光团,但是,使用其信号来研究蛋白质 – 蛋白质相互作用变得复杂分析,因为它们是共同的大多数蛋白质和将存在于两个,受体蛋白和配体。此?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Authors acknowledge the financial support from CONACyT project numbers 167359 and 177138, and from DGAPA-UNAM project number IN201615.
Materials
| 0.5 mm Glass beads | Biospec Products | 11079105 | |
| Tris Base | Formedium | TRIS01 | Ultra pure |
| Glycerol | Sigma-Aldrich | G5516 | |
| dye 4’, 5’-bis(1,3,2 dithioarsolan-2-yl) fluorescein | ThermoFischer Scientific | LC6090 | This kit contains the dye to label a FlAsH tag |
| Ampiciline | IBI Shelton Scientific, Inc | IB02040 | |
| D(+)-Glucose Anhydrous | Formedium | GLU03 | |
| D(+)-Galactose | Formedium | GAL03 | |
| L-Leucine | Formedium | DOC0157 | |
| L-Tryptofan | Formedium | DOC0189 | |
| Bezamidine hydrochloride | Sigma-Aldrich | B6506-5G | |
| PMSF | Gold Biotechnology, Inc | P-470-25 | Phenylmethylsulfonyl fluoride |
| NaCl | Formedium | NAC02 | Sodium Chloride |
| Glycerol | Tecsiquim, S.A. de C.V. | GT1980-6 | |
| MgCl2 | Merck Millipore Corporation | 1725711000 | Magnesium Chloride |
| Imidazole | Sigma-Aldrich | I2399-500G | |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-100ML | |
| K2HPO4 | Sigma-Aldrich | P3786-500G | Potassium phosphate dibasic |
| NaH2PO4 | Sigma-Aldrich | S3139-500G | Sodium phosphate monobasic |
| Yeast nitrogen base without amino acids | Formedium | CYN0410 | |
| Yeast extract | Formedium | YEM03 | Micro Granulated |
| L-Tyroisne | Formedium | DOC0193 | |
| Adenine sulphate | Formedium | DOC0230 | |
| Casamino acids | Formedium | CAS03 | |
| Tryptone | IBI Shelton Scientific, Inc | IB49182 | |
| IPTG | Formedium | IPTG025 | |
| Name of Material | Company | Catalog Number | Comments/Description |
| Filtration units | Merck Millipore Corporation | UFC901096 | Amicon Ultra-15, membrana PLGC Ultracel-PL, 10 kDa |
| Membrane Filter | Merck Millipore Corporation | GSWP04700 | Membrane Filter, mixed cellulose esters, Hydrophilic, 0.22 µm, 47 mm, white, plain |
| Ni2+ affinity column | QIAGEN | 30760 | Cartridge pre-filled with 5 ml Ni-NTA Superflow |
| Strong Sulfopropyl cation exchanger column | GE Healthcare Life Science | 17-5157-01 | HiTrap SP Sepharose FF 5 ml |
| Size Exclusion column | GE Healthcare Life Science | 28989335 | HiLoad 16/600 Superdex 200 PG |
| Fluorescence cell | Hellma Analytics | 111-057-40 | |
| Name of Equipment | Company | Catalog Number | Comments/Description |
| Spectrophotometer | Agilent Technologies | G6860AA | Cary 60 UV-Vis |
| Shaker | ThermoFischer Scientific | SHKA4000-7 | MaxQ 4000 Benchtop temperature range Ambient-15° to 60°C |
| Centrifuge | ThermoFischer Scientific | 75004271 | Heraeus Megafuge 16R |
| FPLC | Pharmacia Biotech | Discontinued | FPLC system conductivity UV-MM II monitor P500 pump fraction |
| Spectrofluorometer | Olis | No applicable | Olis DM 45 with Polarization Toolbox |
References
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440 (7084), 637-643 (2006).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Michnick, S. W., Hien Ear, P., Landry, C., Malleshaiah, M. K., Messier, V. A toolkit of protein-fragment complementation assays for studying and dissecting large-scale and dynamic protein-protein interactions in living cells. Methods in Enzymology. 470, 336-366 (2010).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Dwane, S., Kiely, P. A. Tools used to study how protein complexes are assembled in signaling cascades. Bioeng Bugs. 2 (5), 247-259 (2011).
- Snider, J., et al. Fundamentals of protein interaction network mapping. Mol Syst Biol. 11 (12), 848 (2015).
- Fersht, A., Baldwin, R. L. . Structure and mechanism in protein science: a guide to enzyme catalysis and protein folding. , 191-214 (2002).
- Menne, T. F., et al. The Shwachman-Bodian-Diamond syndrome protein mediates translational activation of ribosomes in yeast. Nat Genet. 39 (4), 486-495 (2007).
- Boocock, G. R., et al. Mutations in SBDS are associated with Shwachman-Diamond syndrome. Nat Genet. 33 (1), 97-101 (2003).
- Garcia-Marquez, A., Gijsbers, A., de la Mora, E., Sanchez-Puig, N. Defective Guanine Nucleotide Exchange in the Elongation Factor-like 1 (EFL1) GTPase by Mutations in the Shwachman-Diamond Syndrome Protein. J Biol Chem. 290 (29), 17669-17678 (2015).
- Gijsbers, A., Garcia-Marquez, A., Luviano, A., Sanchez-Puig, N. Guanine nucleotide exchange in the ribosomal GTPase EFL1 is modulated by the protein mutated in the Shwachman-Diamond syndrome. Biochem Biophys Res Commun. 437 (3), 349-354 (2013).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2010).
- Nishigaki, T., Treviño, C. L., Gòmez, I. . Tools to understand protein-protein interactions. 37, 1-14 (2012).
- Johnson, I. . The Molecular Probes Handbook: A Guide to Fluorescent Probes and Labeling Technologies. , (2010).
- Sabnis, R. W. . Handbook of Fluorescent Dyes and Probes. , (2015).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: synthesis and biological applications. J Am Chem Soc. 124 (21), 6063-6076 (2002).
- Griffin, B. A., Adams, S. R., Tsien, R. Y. Specific covalent labeling of recombinant protein molecules inside live cells. Science. 281 (5374), 269-272 (1998).
- Rashidian, M., Dozier, J. K., Distefano, M. D. Enzymatic labeling of proteins: techniques and approaches. Bioconjug Chem. 24 (8), 1277-1294 (2013).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: A laboratory manual. , (2001).
- Neuhoff, V., Arold, N., Taube, D., Ehrhardt, W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 9 (6), 255-262 (1988).
- West, R. W., Chen, S. M., Putz, H., Butler, G., Banerjee, M. GAL1-GAL10 divergent promoter region of Saccharomyces cerevisiae contains negative control elements in addition to functionally separate and possibly overlapping upstream activating sequences. Genes Dev. 1 (10), 1118-1131 (1987).
- Ito, H., Fukuda, Y., Murata, K., Kimura, A. Transformation of intact yeast cells treated with alkali cations. J Bacteriol. 153 (1), 163-168 (1983).
- Eftink, M. R. Fluorescence methods for studying equilibrium macromolecule-ligand interactions. Methods Enzymol. 278, 221-257 (1997).
- Han, H., et al. Binding of Substrates to the Central Pore of the Vps4 ATPase Is Autoinhibited by the Microtubule Interacting and Trafficking (MIT) Domain and Activated by MIT Interacting Motifs (MIMs). J Biol Chem. 290 (21), 13490-13499 (2015).
- Sanchez-Puig, N., Veprintsev, D. B., Fersht, A. R. Binding of natively unfolded HIF-1alpha ODD domain to p53. Mol Cell. 17 (1), 11-21 (2005).
- Trusch, F., et al. The N-terminal Region of the Ubiquitin Regulatory X (UBX) Domain-containing Protein 1 (UBXD1) Modulates Interdomain Communication within the Valosin-containing Protein p97. J Biol Chem. 290 (49), 29414-29427 (2015).
- Kamp, F., Beyer, K. Binding of alpha-synuclein affects the lipid packing in bilayers of small vesicles. The Journal of Biological Chemsitry. 281, 9251-9259 (2006).
- Bujalowski, W. M., Jezewska, M. J. Fluorescence Intensity, Anisotropy, and Transient Dynamic Quenching Stopped-Flow Kinetics. Spectroscopic Methods of Analysis. 875, 105-133 (2012).
- Asano, N., et al. Direct interaction between EFL1 and SBDS is mediated by an intrinsically disordered insertion domain. Biochem Biophys Res Commun. 443 (4), 1251-1256 (2014).

