Fluorescens anisotropi som et verktøy for å studere protein-protein interaksjoner
Summary
Protein interaksjoner er i hjertet av en celle funksjon. Kalorimetriske og spektroskopiske teknikker blir ofte brukt for å karakterisere dem. Her beskriver vi fluorescens anisotropi som et verktøy for å studere samspillet mellom protein mutert i Shwachman-Diamond syndrom (SBDS) og Forlengelse faktor-lignende en GTPase (EFL1).
Abstract
Protein-protein-interaksjoner spiller en viktig rolle i funksjonen av en levende organisme. Når en interaksjon har blitt identifisert og validert er det nødvendig å karakterisere den på det strukturelle og mekanistiske nivå. Mange biokjemiske og biofysiske metoder finnes for slike formål. Blant dem, er fluorescens anisotrofien en kraftfull teknikk spesielt anvendes når fluorescensintensiteten av en fluorofor-merket protein forblir konstant ved protein-protein-interaksjon. I denne teknikken er en fluorofor-merket protein eksiteres med vertikalt polarisert lys av en passende bølgelengde som selektivt eksiterer en undergruppe av fluoroforene i henhold til deres relative orientering i den innkommende strålen. Den resulterende utslipp har også en retnings hvis forhold i de vertikale og horisontale plan definerer anisotropi (r) som følger: r = (jeg VV -I VH) / (I VV + 2I VH), hvor jeg vv og jeg <sub> VH er fluorescensstyrkene til de vertikale og horisontale komponenter, henholdsvis. Fluorescens anisotropi er følsom for rotasjons diffusjon av en fluorofor, nemlig den tilsynelatende molekylstørrelse på en fluorofor festet til et protein, som er endret ved protein-protein-interaksjon. I den foreliggende teksten, ble bruken av fluorescens anisotropi som et verktøy for å studere protein-protein interaksjoner eksemplifisert for å løse bindingen mellom proteinet mutert i den Shwachman-Diamond syndrom (SBDS) og forlengelsen faktor som-en GTPase (EFL1). Konvensjonelt blir merking av et protein med en fluorofor utført på tiolgrupper (cystein) eller i aminogruppene (N-terminale amin eller lysin) av proteinet. Men besitter SBDS flere cysteiner og lysines som ikke tillater nettstedet rettet merking av det. Som et alternativ teknikk, fargestoffet 4 ', ble 5'-bis (1,3,2 dithioarsolan-2-yl) fluorescein brukes for spesifikt å merke en tetracysteine motiv, Cys-Cys-Pro-Gly-Cys-Cys, genetisk konstruert i den C-terminale ende av det rekombinante SBDS protein. Montering av eksperimentelle data gitt kvantitativ og mekanistisk informasjon om innbindingsmodus mellom disse proteinene.
Introduction
Celler inneholder et mangfold av biomacromolecules som hele tiden kommuniserer med hverandre. Denne assosiasjonen gir opphav til komplekser som deltar i de cellulære veier som er ansvarlige for deres funksjon i signaltransduksjon, regulering av genekspresjon og cellevandring blant andre. Alt protein-protein interaksjoner som forekommer i en celle omfatter et nettverk kjent som interactome. I Saccharomyces cerevisiae mer enn 70% av sitt protein har vist seg å ha samvirkende partnerne 1. Forstå interactome av en celle og deres funksjoner gi relevant informasjon om kompleksiteten og mangfoldet av levende organismer. Flere metoder har blitt beskrevet å identifisere og karakterisere protein-protein interaksjoner. Forskjellig høy gjennom benyttet metoder som gjær to-hybrid 2, protein-fragment komplemente assays 3, affinitetsrensing 4 koblet til massespektrometri og protein microarrays brukes til å identifisere en interaksjon 5,6. Når identifisert, er det nødvendig å validere det, og dette kan variere fra sak til sak. Vanligvis disse eksperimentene involverer forstyrre samspillet selv på nivå med de enkelte medlemmer av samspillet paret, for eksempel ved genet sletting eller overekspresjon av en av proteiner, og deretter se etter endringer i egenskaper eller funksjon av den andre medlemmet på cellenivå. Deretter blir biofysiske teknikker 7 som brukes til å karakterisere protein-protein interaksjon på molekylært nivå. For dette formål, er strukturen av proteinkomplekser bestemt ved røntgen-krystallografi, kjernemagnetisk resonans og kryo-elektronmikroskopi mens kalorimetri og fluorescens-spektroskopi blir brukt for å kvantitativt og mekanistisk beskrive dem.
I dette arbeidet ble fluorescens anisotropi brukt som en teknikk for å karakterisere samspillet mellom GTPase EFL1 og SBDS-proteinet. Disse proteinene deltar i syntesen av ribosomer ved å fremme frigivelse av eukaryotisk initiering faktor 6 fra overflaten av den 60S ribosomale subenhet 8. Den SBDS proteinet er mutert på en sykdom som er kjent som den Shwachman-Diamond syndrom 9 og virker som et guanin nukleotid utveksling faktor for EFL1 avtagende dets affinitet for guanosin difosfat 10,11. Sykdoms mutasjoner i SBDS avskaffe samspillet med EFL1 og dermed hindre aktivering.
Fluorescens anisotropi er ofte brukt i biologiske anvendelser for å studere protein-peptid eller protein-nukleinsyre-interaksjoner. Den er basert på det prinsipp at en fluoroforen eksiteres med polarisert lys resulterer i en delvis polarisert utslipp. Fluorescens anisotropi er definert ved ligning 1:
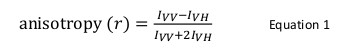
der jeg vv og jeg VH erfluorescensstyrkene den vertikalt (VV) og horisontalt (VH) polarisert utslipp når prøven er spent med vertikalt polarisert lys 12. Fluorescens anisotropi er følsom for faktorer som påvirker frekvensen av rotasjons diffusjon av fluoroforen og således avhenger av temperaturen, viskositeten av løsningen og den tilsynelatende molekylstørrelse av fluoroforen. Den tilsynelatende størrelse av et protein inneholdende en fluorofor øker når den kommuniserer med et annet protein og en slik endring kan deretter evalueres som en endring i anisotropi. Nærmere bestemt vil en fluorofor som roterer langsomt i oppløsning i forhold til dets fluoriserende levetid vil ha en stor I VV verdi og liten jeg VH verdi, og derfor vil oppvise en forholdsvis stor anisotropi. For fluorophores som tørkes raskt i forhold til deres fluorescerende levetid, jeg vv og jeg VH vil være like og deres anisotropi verdien vil være liten 12 </sup> (Figur 1). I tillegg, for en god anisotropi signal til støymåling, er det nødvendig å ha en fluorofor med en fluorescens levetid lik den rotasjonskorrelasjonstiden til molekylet av interesse. Ellers er det ikke mulig å nøyaktig registrere forskjellen i anisotrofien mellom den frie protein, og at i komplekset. For eksempel, anisotropien av en fluorescerende probe med en levetid nær 4 nsek som for eksempel fluorescein eller rhodamin festet til en lavmolekylær forbindelse med 100 Da er 0,05. Binding til et molekyl av 160 kDa vil øke dens anisotropi verdien til 0,29; en forskjell som kan måles nøyaktig. I motsetning til dette, vil det samme fluorescerende probe som er involvert i en bindingsreaksjon, som plutselig økning i molekylstørrelse varierer fra 65 til 1000 kDa bare resultere i en endring anisotropi på 0,28 til 0,3, noe som er for små til å måles nøyaktig. I dette scenariet vil en sonde med en levetid på 400 ns være mer egnet 12.
<pclass = "jove_content">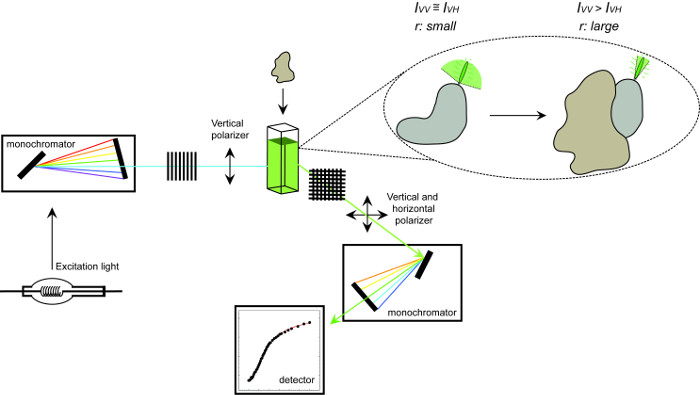
Figur 1. Skjematisk representasjon av utstyret som brukes til å måle fluorescens anisotropi og prosedyren. Skjematisk representasjon av utstyret som brukes til å utføre en protein-protein interaksjon eksperiment som måler fluorescensen anisotropi. Fluoroforer som ristes rask visnings liten anisotropi som øker ved binding til et samspill partner. Klikk her for å se en større versjon av dette tallet.
Fluorescensanvendelser krever tilstedeværelse av en fluorofor i hvilket som helst av molekylene studert. For å undersøke protein-protein interaksjoner er det tre typer fluoroforer: 1) tryptofanrester som er tilstede i proteiner, 2) kjemisk festet fluoroforer og 3) fluoriserende fusjonspartnere som grønt fluorescerende protein (GFP) og dens derivatanter. De fleste proteiner har tryptofanrester på sin struktur, og dermed den enkleste måten for å måle en interaksjon er ved å overvåke endringene i den tilsvarende fluorescensspektra eller ved å overvåke endringer i fluorescens-intensiteten av tryptofanrester. Imidlertid kan tryptofanrester være til stede i begge proteiner kompliserer analysen. På den annen side, for en fluorofor til å endre sin fluoriserende egenskaper på grunn av en interaksjon det er behov for å være plassert på eller nær bindingssetet og det kunne interferere med interaksjonen selv. Dette trenger spesiell oppmerksomhet når du bruker store fluoroforer som GFP. Hvis ingen av disse fluoroforer kan brukes til bindingsstudier er det nødvendig, da, å innføre ytre fluoroforer til den ene av de involverte proteiner. Mange kjemisk syntetiserte fluoroforer eksistere og kan være kovalent bundet til proteiner via sine reaktive grupper slik som aminogrupper (sidekjede av lysin eller N-terminus) og tiol- grupper i cystein. Fluorophore derivater med isothiocyanate og suksinimidyl estere reagere med amidgrupper mens iodoacetamide og maleimide er tiol-reaktive grupper 13. De mest vanlige fargestoffer som brukes i fluorescensanvendelser er derivater av fluorescein og rhodamin grønne farvestoffer, koumariner, BODIPY fluoroforer og Alexa Fluor fargestoffer. En detaljert liste over kommersielt tilgjengelige fluoroforer og deres anvendelse kan finnes i referanser 14,15. For vellykket merking, må den reaktive gruppe være eksponert på overflaten av proteinet, men på grunn av det store antall reaktive funksjonelle grupper som typisk er tilstede i polypeptidene det er svært vanskelig å få sete-spesifikk modifisering. Proteinet av interesse i denne studien, SBDS, inneholder 5 gratis cysteiner og 33 lysines som kan resultere i flere språk merking. Ikke-uniform merking kan påvirke binding og vil komplisere dataanalyse som ulike fluoroformolekyler kan lokke fram ulike fluorescerende intensitet signaler ved binding. Overcome dette problemet, brukte vi Flash fluorophore, 4 ', 5'-bis (1,3,2 dithioarsolan-2-yl) fluorescein til nettstedet Direkt label SBDS protein. Dette er en arsenoxide fargestoff med en høy affinitet for fire adskilte cysteiner i et motiv kjent som flash-kode som består av sekvensen CCXXCC hvor X er hvilken som helst aminosyre annen enn cystein 16,17. Dette tetracysteine motiv tilsettes til N- eller C-terminale enden av proteinet ved hjelp av genteknologi sammen med en egnet linker for å forhindre forstyrrelse av den samlede fold av proteinet. Paret består av Flash fargestoff og Flash-koden ble opprinnelig utviklet for å stedsspesifikke label proteiner i levende celler 17, men det kan også brukes til å merke rensede proteiner in vitro som det er eksemplifisert her. I tillegg har enzymatiske strategier også blitt utviklet for å muliggjøre stedsspesifikke funksjonalisering av proteiner 18.
I dette manuskriptet beskriver vi nytten av fluorescens anisotropi enet verktøy for å studere protein-protein interaksjoner. Binding kan vurderes ved enkel inspeksjon av bindingen kurveformen mens kvantitativ informasjon kan fås fra tilpasning av de eksperimentelle data.
Protocol
Representative Results
Discussion
De fleste biokjemiske forsøk med proteiner krever ikke bare rent protein, men også store mengder av dem, uavhengig av hvilken teknikk som brukes. Av denne grunn er de proteinene som benyttes for denne type forsøk erholdt ved heterolog ekspresjon, slik det var tilfellet presentert her. Florescence spektroskopi krever tilstedeværelse av en fluorofor i den studerte molekylet. Aromatiske rester utgjør den iboende fluoroforene av et protein, men ved hjelp av sitt signal for å studere protein-protein interaksjoner kompl…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Authors acknowledge the financial support from CONACyT project numbers 167359 and 177138, and from DGAPA-UNAM project number IN201615.
Materials
| 0.5 mm Glass beads | Biospec Products | 11079105 | |
| Tris Base | Formedium | TRIS01 | Ultra pure |
| Glycerol | Sigma-Aldrich | G5516 | |
| dye 4’, 5’-bis(1,3,2 dithioarsolan-2-yl) fluorescein | ThermoFischer Scientific | LC6090 | This kit contains the dye to label a FlAsH tag |
| Ampiciline | IBI Shelton Scientific, Inc | IB02040 | |
| D(+)-Glucose Anhydrous | Formedium | GLU03 | |
| D(+)-Galactose | Formedium | GAL03 | |
| L-Leucine | Formedium | DOC0157 | |
| L-Tryptofan | Formedium | DOC0189 | |
| Bezamidine hydrochloride | Sigma-Aldrich | B6506-5G | |
| PMSF | Gold Biotechnology, Inc | P-470-25 | Phenylmethylsulfonyl fluoride |
| NaCl | Formedium | NAC02 | Sodium Chloride |
| Glycerol | Tecsiquim, S.A. de C.V. | GT1980-6 | |
| MgCl2 | Merck Millipore Corporation | 1725711000 | Magnesium Chloride |
| Imidazole | Sigma-Aldrich | I2399-500G | |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-100ML | |
| K2HPO4 | Sigma-Aldrich | P3786-500G | Potassium phosphate dibasic |
| NaH2PO4 | Sigma-Aldrich | S3139-500G | Sodium phosphate monobasic |
| Yeast nitrogen base without amino acids | Formedium | CYN0410 | |
| Yeast extract | Formedium | YEM03 | Micro Granulated |
| L-Tyroisne | Formedium | DOC0193 | |
| Adenine sulphate | Formedium | DOC0230 | |
| Casamino acids | Formedium | CAS03 | |
| Tryptone | IBI Shelton Scientific, Inc | IB49182 | |
| IPTG | Formedium | IPTG025 | |
| Name of Material | Company | Catalog Number | Comments/Description |
| Filtration units | Merck Millipore Corporation | UFC901096 | Amicon Ultra-15, membrana PLGC Ultracel-PL, 10 kDa |
| Membrane Filter | Merck Millipore Corporation | GSWP04700 | Membrane Filter, mixed cellulose esters, Hydrophilic, 0.22 µm, 47 mm, white, plain |
| Ni2+ affinity column | QIAGEN | 30760 | Cartridge pre-filled with 5 ml Ni-NTA Superflow |
| Strong Sulfopropyl cation exchanger column | GE Healthcare Life Science | 17-5157-01 | HiTrap SP Sepharose FF 5 ml |
| Size Exclusion column | GE Healthcare Life Science | 28989335 | HiLoad 16/600 Superdex 200 PG |
| Fluorescence cell | Hellma Analytics | 111-057-40 | |
| Name of Equipment | Company | Catalog Number | Comments/Description |
| Spectrophotometer | Agilent Technologies | G6860AA | Cary 60 UV-Vis |
| Shaker | ThermoFischer Scientific | SHKA4000-7 | MaxQ 4000 Benchtop temperature range Ambient-15° to 60°C |
| Centrifuge | ThermoFischer Scientific | 75004271 | Heraeus Megafuge 16R |
| FPLC | Pharmacia Biotech | Discontinued | FPLC system conductivity UV-MM II monitor P500 pump fraction |
| Spectrofluorometer | Olis | No applicable | Olis DM 45 with Polarization Toolbox |
References
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440 (7084), 637-643 (2006).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Michnick, S. W., Hien Ear, P., Landry, C., Malleshaiah, M. K., Messier, V. A toolkit of protein-fragment complementation assays for studying and dissecting large-scale and dynamic protein-protein interactions in living cells. Methods in Enzymology. 470, 336-366 (2010).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Dwane, S., Kiely, P. A. Tools used to study how protein complexes are assembled in signaling cascades. Bioeng Bugs. 2 (5), 247-259 (2011).
- Snider, J., et al. Fundamentals of protein interaction network mapping. Mol Syst Biol. 11 (12), 848 (2015).
- Fersht, A., Baldwin, R. L. . Structure and mechanism in protein science: a guide to enzyme catalysis and protein folding. , 191-214 (2002).
- Menne, T. F., et al. The Shwachman-Bodian-Diamond syndrome protein mediates translational activation of ribosomes in yeast. Nat Genet. 39 (4), 486-495 (2007).
- Boocock, G. R., et al. Mutations in SBDS are associated with Shwachman-Diamond syndrome. Nat Genet. 33 (1), 97-101 (2003).
- Garcia-Marquez, A., Gijsbers, A., de la Mora, E., Sanchez-Puig, N. Defective Guanine Nucleotide Exchange in the Elongation Factor-like 1 (EFL1) GTPase by Mutations in the Shwachman-Diamond Syndrome Protein. J Biol Chem. 290 (29), 17669-17678 (2015).
- Gijsbers, A., Garcia-Marquez, A., Luviano, A., Sanchez-Puig, N. Guanine nucleotide exchange in the ribosomal GTPase EFL1 is modulated by the protein mutated in the Shwachman-Diamond syndrome. Biochem Biophys Res Commun. 437 (3), 349-354 (2013).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2010).
- Nishigaki, T., Treviño, C. L., Gòmez, I. . Tools to understand protein-protein interactions. 37, 1-14 (2012).
- Johnson, I. . The Molecular Probes Handbook: A Guide to Fluorescent Probes and Labeling Technologies. , (2010).
- Sabnis, R. W. . Handbook of Fluorescent Dyes and Probes. , (2015).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: synthesis and biological applications. J Am Chem Soc. 124 (21), 6063-6076 (2002).
- Griffin, B. A., Adams, S. R., Tsien, R. Y. Specific covalent labeling of recombinant protein molecules inside live cells. Science. 281 (5374), 269-272 (1998).
- Rashidian, M., Dozier, J. K., Distefano, M. D. Enzymatic labeling of proteins: techniques and approaches. Bioconjug Chem. 24 (8), 1277-1294 (2013).
- Maniatis, T., Fritsch, E. F., Sambrook, J. . Molecular cloning: A laboratory manual. , (2001).
- Neuhoff, V., Arold, N., Taube, D., Ehrhardt, W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 9 (6), 255-262 (1988).
- West, R. W., Chen, S. M., Putz, H., Butler, G., Banerjee, M. GAL1-GAL10 divergent promoter region of Saccharomyces cerevisiae contains negative control elements in addition to functionally separate and possibly overlapping upstream activating sequences. Genes Dev. 1 (10), 1118-1131 (1987).
- Ito, H., Fukuda, Y., Murata, K., Kimura, A. Transformation of intact yeast cells treated with alkali cations. J Bacteriol. 153 (1), 163-168 (1983).
- Eftink, M. R. Fluorescence methods for studying equilibrium macromolecule-ligand interactions. Methods Enzymol. 278, 221-257 (1997).
- Han, H., et al. Binding of Substrates to the Central Pore of the Vps4 ATPase Is Autoinhibited by the Microtubule Interacting and Trafficking (MIT) Domain and Activated by MIT Interacting Motifs (MIMs). J Biol Chem. 290 (21), 13490-13499 (2015).
- Sanchez-Puig, N., Veprintsev, D. B., Fersht, A. R. Binding of natively unfolded HIF-1alpha ODD domain to p53. Mol Cell. 17 (1), 11-21 (2005).
- Trusch, F., et al. The N-terminal Region of the Ubiquitin Regulatory X (UBX) Domain-containing Protein 1 (UBXD1) Modulates Interdomain Communication within the Valosin-containing Protein p97. J Biol Chem. 290 (49), 29414-29427 (2015).
- Kamp, F., Beyer, K. Binding of alpha-synuclein affects the lipid packing in bilayers of small vesicles. The Journal of Biological Chemsitry. 281, 9251-9259 (2006).
- Bujalowski, W. M., Jezewska, M. J. Fluorescence Intensity, Anisotropy, and Transient Dynamic Quenching Stopped-Flow Kinetics. Spectroscopic Methods of Analysis. 875, 105-133 (2012).
- Asano, N., et al. Direct interaction between EFL1 and SBDS is mediated by an intrinsically disordered insertion domain. Biochem Biophys Res Commun. 443 (4), 1251-1256 (2014).

