Gleichzeitige Messung von HDAC1 und HDAC6 Tätigkeit in HeLa-Zellen mit UHPLC-MS
Summary
Das vorliegende Verfahren dient der Isoform-spezifische Inhibitoren der Histon Deacetylases (HDAC) in HeLa-Zellen durch die UHPLC-MS-Analyse von mehreren Substraten zu identifizieren. Dies ist ein Antikörper-freie Methode entwickelt, um HDAC1 und HDAC6 Tätigkeit im Wohnzimmer wiedergeben Zelle Umgebung, im Gegensatz zu Single-Isoform zellfreie Assays.
Abstract
Die Suche nach neuen Histon-Deacetylase (HDAC)-Inhibitoren ist zunehmend an Bedeutung in der Wirkstoffforschung. Isoform Selektivität ist die Genehmigung des Romidepsin, eine Klasse im Rampenlicht seit ich HDAC Inhibitor für Krebstherapie und die klinische Untersuchung der HDAC6-spezifische Hemmstoffe für Multiples Myelom. Das vorliegende Verfahren dient die inhibitorische Aktivität der Testverbindungen für HDAC1 und HDAC6 in den Zellen zu bestimmen. Die Isoform Aktivität wird anhand die ultra-high-Performance liquide Chromatography-Massenspektrometrie (UHPLC-MS) Analyse der spezifischen Substraten mit behandelten und unbehandelten HeLa-Zellen inkubiert. Die Methode hat den Vorteil der Reflexion der endogenen HDAC-Aktivität innerhalb der Umgebung der Zelle, im Gegensatz zur zellfreien biochemischen Assays auf isolierte Isoformen durchgeführt. Darüber hinaus da es auf die Quantifizierung der synthetische Substrate basiert, erfordert die Methode nicht die Antikörper Anerkennung der endogenen Schimmelpilzschäden Proteine. Es ist leicht anpassbar an verschiedene Zelllinien und einen automatisierten Prozess. Die Methode hat sich bereits bei der Suche nach HDAC6-selektive Verbindungen in Neuroblasten bewährt. Repräsentative Ergebnisse werden hier gezeigt, mit dem standard HDAC-Inhibitoren Trichostatin ein (unspezifisch), MS275 (HDAC1-spezifisch) und Tubastatin (HDAC6-spezifisch) mit HeLa-Zellen.
Introduction
HDACs gehören zu einer Familie von Enzymen in der Lage, die Histone innerhalb der Chromatinstruktur deacetylate. Sie haben andere Protein-Substrate in der Zellflüssigkeit und befinden sich in verschiedenen Kompartimenten der Zelle. Insgesamt 18 HDAC-Isoformen wurden bisher identifiziert und haben wurde im Zusammenhang mit mehreren Zellmechanismen, einschließlich der Regulierung von Transkriptionsfaktoren und Genexpression sowie Zelle signalisieren und1,2,3,4,5,6,7. Katalytische HDAC-Inhibitoren haben als mögliche therapeutische Medikamente für die Krebstherapie entwickelt. Die meisten HDAC-Inhibitoren derzeit von der FDA zugelassen sind für die Behandlung von T-Zell-Lymphom und Multiples Myelom, und unspezifische HDAC-Inhibitoren, wie z. B. Vorinostat (SAHA), Belinostat und Panobinostat8,9. Jedoch eine Reihe von Nebenwirkungen wurden im Zusammenhang mit Pan-Hemmer, und die Suche nach Isoform-spezifische kleine Moleküle ist ein heißes Thema in der medizinischen Chemie und Drug Discovery. Entsprechend der Klasse ich (HDAC1-3 und HDAC8) selektiver Hemmstoff-Romidepsin ist ein bereits zugelassenes Medikament10, während HDAC6-spezifische Inhibitoren derzeit in klinischen Studien mit erhöhten therapeutisches Potenzial in multiplen Myeloms11,12,13,14,15sind.
Screening-Tests zur Charakterisierung von HDAC Inhibitoren die Inkubation von einem HDAC-Substrat mit einer enzymatischen Quelle (einzelne Isoform, nukleare Extrakt oder Zelle lysate) basieren. Das Substrat ist in der Regel eine kleine Peptidsequenz eine Acetyl Lysin Rückstände mit einem spaltbaren Fluorophor (z. B. Cumarin), gekoppelt mit z. B. N-(4-methyl-7-aminocoumarinyl)-Nα-(t-butoxycarbonyl)-Nω-acetyllysineamide (MAL)16. Zur Unterscheidung zwischen Isoform-spezifische Aktivitäten separate zellfreie Assays unter Einbeziehung jedes Isoform notwendig sind und möglicherweise nicht wider die echte Isoform Aktivität in lebenden Zellen. Isoform-spezifische Substrate sind im Handel erhältlich, wie z. B. Benzyl (S)-[1-(4-methyl-2-oxo-2H-chromen-7-ylcarbamoyl)-5-propionylaminopentyl]carbamate (MOCPAC, HDAC1 spezifische Substrat) und (S)-[5-acetylamino-1-(2-oxo-4-trifluoromethyl-2H-chromen-7-ylcarbamoyl)pentyl]carbamic-Säure Tert-Butyl-Ester (BATCP, HDAC6 spezifische Substrat) (Abbildung 1 b). Allerdings wird ein Multi-Substrat-Gemisch mit MAL, MOCPAC und BATCP gegeben zu lebenden Zellen nicht die Erkennung der einzelnen deacetylated Produkte von fluorometrisch Messung erlauben angesichts der Tatsache, dass sie die gleichen spaltbaren Fluorophor tragen.
Die hier beschriebene Methode ermöglicht die Erkennung und relative Quantifizierung jedes Substrat und ihrer deacetylated Produkte in HeLa-Zellen mit einem Multi-Substrat-Assay, gefolgt von UHPLC-ESI-MS/MS-Analyse17. Ein HDAC-Test wird auf HeLa-Zellen ermöglichen die direkte Identifizierung von HDAC hemmende Aktivität und der Besonderheit des Testverbindungen auf endogene HDACs durchgeführt. Gibt es ein Fokus auf HDAC1 und HDAC6, die gleichzeitig ausgewertet werden. Um diese enzymatischen Messungen in einem einzigen Inkubation Assay zu erreichen, ist eine Mischung aus unspezifischen und spezifischen HDAC-Substrate behandelten und unbehandelten HeLa-Zellen auf eine 96-Well-Platte vergoldet hinzugefügt. Im Anschluss an eine Inkubationsschritt sind Zellen lysiert, veröffentlichte er die Substrate und ihre jeweiligen Reaktionsprodukte, die sind getrennt und mit einer UHPLC-MS-Methode (Abbildung 1) nachgewiesen. Die deacetylated Produkte der MAL, MOCPAC und BATCP Substrate sind die deacetylated MAL (dMAL), deacetylated MOCPAC (dMOCPAC) und deacetylated BATCP (dBATCP), beziehungsweise. Dosis-Wirkungs-Kurven können mit aktiven Verbindungen aufgebaut werden.
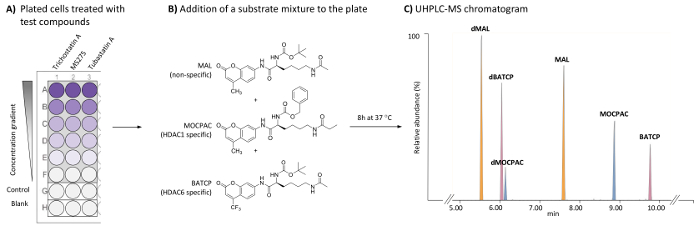
Abbildung 1: allgemeine Regelung für die zellbasierte HDAC-Assay, HDAC1 und HDAC6-spezifische Inhibitoren durch die UHPLC-MS-Analyse von mehreren Substraten zu identifizieren. (A) Schema eines typischen 96-Well-Platte mit behandelt (Testverbindungen) und unbehandelten (Kontrolle) HeLa-Zellen, sowie zellfreie Rohlinge. (B) chemische Struktur von Substraten durch endogene HDACs deacetylated als eine Mischung (21 µM) hinzugefügt. (C) typische UHPLC-MS Chromatogramm zeigt die Gipfeln die hinzugefügte Substrate (MAL, MOCPAC und BATCP) und ihre deacetylated Produkte (dMAL, dMOCPAC, und dBACTP, beziehungsweise). Bitte klicken Sie hier für eine größere Version dieser Figur.
Protocol
Representative Results
Discussion
HDAC-Hemmung ist ein heißes Thema in der Drogeentdeckung, mit dem aktuellen Fokus auf HDAC6-selektive Inhibitoren für Krebs-Therapie-21. HDAC Selektivität bemisst sich in der Regel durch eine Reihe von hohem Durchsatz, zellfreie Assays, die beabsichtigen, die inhibitorische Potenz gegenüber einzelnen HDAC Isoformen21bestimmen. Jedoch die Selektivität der Inhibitor muss zusätzlich bestätigt werden in lebenden Zellen durch die Auswertung des Acetylierung Status des end…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Die Autoren erkennen COST Action CM1406 (epigenetische chemische Biologie). Forschung berichtet in dieser Publikation wurde von der Stiftung Pierre Mercier unterstützt.
Materials
| BATCP | Sigma-Aldrich | B4061 | |
| MAL | Sigma-Aldrich | SCP0168 | Synonym: BOC-Ac-Lys-AMC |
| MOCPAC | Sigma-Aldrich | M2195 | |
| MS275 | Sigma-Aldrich | EPS002 | |
| Trichostatin A | Sigma-Aldrich | T8552 | |
| Tubastatin A | Sigma-Aldrich | SML0044 | |
| Acetonitrile, HPLC grade | Fisher Scientific | 10660131 | |
| Formic acid, LC/MS grade | Fisher Scientific | 10596814 | |
| H2O, HPLC grade | distilled H2O filtered through a Milli-Q purification system | ||
| HeLa cells | ATCC | ATCC CRM-CCL-2 | |
| Cell dissociation reagent TrypLE Express | ThermoFisher Scientific | 12604013 | |
| DMSO, cell culture grade | Applichem | 146463 | |
| DPBS | ThermoFisher Scientific | 14190144 | |
| Fetal bovine serum | Biowest | S1810 | |
| MEM | ThermoFisher Scientific | 22561021 | |
| Penicillin-Streptomycin | Bioconcept | 4-01F00-H | |
| 10X RIPA buffer | Abcam | ab156034 | |
| SigmaFast protease inhibitor tablets | Sigma-Aldrich | S8820 | |
| 96-well plates, sterile, flat-bottom, tissue culture treated Corning | VWR | 29442-058 | |
| 96-well plates, non-sterile, V-bottom Corning | VWR | 29442-404 | used in the centrifugation step |
| 96-well plate, conical bottom, Nunc | ThermoFisher Scientific | 249944 | compatible with the Acquity UHPLC system |
| T75 cell culture flasks Corning | Sigma-Aldrich | CLS430641 | |
| Peelable heat sealing foil | Waters | 186002789 | |
| Acquity UPLC system | Waters | ||
| Eppendorf Centrifuge 5810 R | Fisher Scientific | 05-413-323 | |
| Integration software: MassLynx V4.1 | Waters | Catalog number not available | |
| Combi thermo-sealer SP-0669/240 | Waters | Catalog number not available | |
| Quattro micro API Tandem Quadrupole System | Waters | Catalog number not available |
References
- Arrowsmith, C. H., Bountra, C., Fish, P. V., Lee, K., Schapira, M. Epigenetic protein families: a new frontier for drug discovery. Nat Rev Drug Discov. 11 (5), 384-400 (2012).
- Henikoff, S., Greally, J. M. Epigenetics, cellular memory and gene regulation. Curr Biol. 26 (14), R644-R648 (2016).
- Kim, C., et al. HDAC6 inhibitor blocks amyloid beta-induced impairment of mitochondrial transport in hippocampal neurons. PLoS One. 7 (8), (2012).
- Kawaguchi, Y., et al. The deacetylase HDAC6 regulates aggresome formation and cell viability in response to misfolded protein stress. Cell. 115 (6), 727-738 (2003).
- Zhao, Y., et al. Acetylation of p53 at lysine 373/382 by the histone deacetylase inhibitor depsipeptide induces expression of p21(Waf1/Cip1). Mol Cell Biol. 26 (7), 2782-2790 (2006).
- Berger, S. L. Histone modifications in transcriptional regulation. Curr Opin Genet Dev. 12 (2), 142-148 (2002).
- Simões-Pires, C., et al. HDAC6 as a target for neurodegenerative diseases: what makes it different from the other HDACs?. Mol Neurodegener. 8 (7), 1-16 (2013).
- Mottamal, M., Zheng, S., Huang, T. L., Wang, G. Histone deacetylase inhibitors in clinical studies as templates for new anticancer agents. Molecules. 20 (3), 3898-3941 (2015).
- Manal, M., Chandrasekar, M. J., Gomathi Priya, J., Nanjan, M. J. Inhibitors of histone deacetylase as antitumor agents: A critical review. Bioorg Chem. 67, 18-42 (2016).
- Mack, G. S. To selectivity and beyond. Nat Biotech. 28 (12), 1259-1266 (2010).
- . Study of ACY-1215 alone and in combination with bortezomib and dexamethasone in multiple myeloma (ACY-1215) Available from: https://clinicaltrials.gov/ct2/show/NCT01323751 (1211)
- . Study of ACY-1215 in combination with lenalidomide, and dexamethasone in multiple myeloma Available from: https://clinicaltrials.gov/ct2/show/NCT01583283 (2012)
- . 13ACY-1215 (ricolinostat) in combination with pomalidomide and low-dose dex in relapsed-and-refractory multiple myeloma Available from: https://clinicaltrials.gov/ct2/show/NCT01997840 (2013)
- . Study of ACY-241 alone and in combination with pomalidomide and dexamethasone in multiple myeloma Available from: https://clinicaltrials.gov/ct2/show/NCT02400242 (2015)
- Yee, A. J., et al. Ricolinostat plus lenalidomide, and dexamethasone in relapsed or refractory multiple myeloma: a multicentre phase 1b trial. Lancet Oncol. 17 (11), 1569-1578 (2016).
- Jung, M. Homogenous non-isotopic assays for histone deacetylase activity. Expert Opin Ther Pat. 13 (6), 935 (2003).
- Zwick, V., Simões-Pires, C., Cuendet, M. Cell-based multi-substrate assay coupled to UHPLC-ESI-MS/MS for a quick identification of class-specific HDAC inhibitors. J Enzyme Inhibi Med Chem. 31 (1), 209-214 (2016).
- Khan, N., et al. Determination of the class and isoform selectivity of small-molecule histone deacetylase inhibitors. Biochem J. 409 (2), 581-589 (2008).
- Glaser, K. B., et al. Differential protein acetylation induced by novel histone deacetylase inhibitors. Biochem Biophys Res Commun. 325 (3), 683-690 (2004).
- Butler, K. V., et al. Rational design and simple chemistry yield a superior, neuroprotective HDAC6 inhibitor, tubastatin A. J Am Chem Soc. 132 (31), 10842-10846 (2010).
- Jung, M., Yong, K. -. J., Velena, A., Lee, S., Sippl, W., Jung, M. Cell-Based Assays for HDAC Inhibitor Hit Validation. Epigenetic Targets in Drug Discovery. , (2010).
- Milli, A., et al. Proteomic analysis of cellular response to novel proapoptotic agents related to atypical retinoids in human IGROV-1 ovarian carcinoma cells. J Proteome Res. 10 (3), 1191-1207 (2011).
- Zwick, V., et al. Synthesis of a selective HDAC6 inhibitor active in neuroblasts. Bioorg Med Chem Lett. 26 (20), 4955-4959 (2016).
- Ciossek, T., Julius, H., Wieland, H., Maier, T., Beckers, T. A homogeneous cellular histone deacetylase assay suitable for compound profiling and robotic screening. Anal Biochem. 372 (1), 72-81 (2008).
- Heltweg, B., Dequiedt, F., Marshall, B. L., Brauch, C., Yoshida, M., et al. Subtype selective substrates for histone deacetylases. J Med Chem. 47 (21), 5235-5243 (2004).
- Copeland, R. A. . Evaluation of enzyme inhibitors in drug discovery. , (2005).

