Medição simultânea de HDAC1 e atividade HDAC6 em HeLa células usando UHPLC-MS
Summary
O presente método serve para identificar a isoforma específica inibidores da histona deacetilases (HDAC) em células HeLa pela análise de vários substratos UHPLC-MS. Este é um método livre de anticorpos desenvolvido para refletir a atividade HDAC1 e HDAC6 do vida ambiente celular, em contraste com o single-isoforma celular livre ensaios.
Abstract
A busca de novos inibidores de histona deacetilase (HDAC) é de crescente interesse na descoberta da droga. Isoform seletividade tem sido o centro das atenções desde a aprovação da romidepsin, uma classe eu HDAC inibidor para terapia do câncer e a investigação clínica de inibidores de HDAC6 específicos para mieloma múltiplo. O presente método é usado para determinar a atividade inibitória de compostos de teste em HDAC1 e HDAC6 nas células. A atividade isoform é medida usando a cromatografia líquida de ultraelevado-desempenho – análise de espectrometria de massa (UHPLC-MS) de substratos específicos incubados com as células HeLa tratadas e não tratadas. O método tem a vantagem de refletir a atividade HDAC endógena dentro do ambiente celular, em contraste com a bioquímicos celular livre ensaios realizados sobre isoformas isoladas. Além disso, porque é baseado na quantificação dos substratos sintéticos, o método não requer o reconhecimento de anticorpos endógenos proteínas acetilado. É facilmente adaptável para várias linhas de células e um processo automatizado. O método já provou útil em encontrar compostos HDAC6-seletiva em neuroblastos. Resultados representativos são mostrados aqui com a padrão HDAC inibidores tricostatina um (não-específica), MS275 (HDAC1-específico), e tubastatin um HDAC6 (específico) usando as células HeLa.
Introduction
Das HDACs pertencem a uma família de enzimas capazes de deacetylate histones dentro da estrutura da cromatina. Eles também têm outros substratos de proteína no citosol e localizam-se em vários compartimentos da célula. Um total de 18 HDAC isoformas foram identificados até agora e têm sido relacionados a vários mecanismos de célula, incluindo a regulação de factores de transcrição e expressão gênica, bem como sinalização celular e transporte1,2,3,4,5,6,7. Catalíticos inibidores HDAC têm emergido como drogas terapêuticas potenciais para a terapia do câncer. A maioria dos inibidores HDAC atualmente aprovados pela FDA são para o tratamento de linfoma de células T e mieloma múltiplo e são inibidores HDAC não específicos, tais como vorinostat (Santos), belinostat e panobinostat8,9. No entanto, uma série de efeitos colaterais foram associados com pan-inibidores, e a busca de pequenas moléculas específicas isoform é um tema quente na descoberta química e drogas medicinal. Nesse sentido, a classe romidepsin de inibidor seletivo (HDAC1-3 e HDAC8) é uma droga já aprovada10, embora inibidores de HDAC6 específicas estão atualmente em ensaios clínicos, com maior potencial terapêutico em mieloma múltiplo11,12,13,14,15.
Seleção de ensaios de caracterização de HDAC inibidores baseiam-se na incubação de um substrato HDAC com uma fonte enzimática (única isoforma, extrato nuclear ou lisado celular). O substrato é geralmente uma sequência de pequenos peptídeos contendo um resíduo de lisina acetil acoplado a um fluoróforo cleavable (por exemplo, cumarina), tais como N-(4-methyl-7-aminocoumarinyl)-Nα-(t-butoxycarbonyl)-Nω-acetyllysineamide (MAL)16. Para distinguir entre isoform-atividades específicas, separadas sem célula ensaios envolvendo cada isoform são necessários e podem não refletir a atividade real isoform em pilhas vivas. Isoforma específica substratos são comercialmente disponíveis, tais como benzílico (S)-[1-(4-methyl-2-oxo-2H-chromen-7-ylcarbamoyl)-5-propionylaminopentyl]carbamate (MOCPAC, substrato específico HDAC1) e (éster de ácido tert-butil S)-[5-acetylamino-1-(2-oxo-4-trifluoromethyl-2H-chromen-7-ylcarbamoyl)pentyl]carbamic (BATCP, HDAC6 substrato específico) (figura 1B). No entanto, uma mistura de substrato multi contendo MAL, MOCPAC e BATCP, dado que as células vivas não permitirá a detecção de produtos deacetylated individuais por medição fluorométrica, dado que carregam o fluoróforo cleavable mesmo.
O método descrito aqui permite a detecção e quantificação relativa de cada substrato e seu produto deacetylated em células HeLa, usando um ensaio de substrato multi seguido de análise de UHPLC-ESI-MS/MS17. Um ensaio HDAC é realizado em células de HeLa, para permitir a identificação directa da atividade inibitória de HDAC e da especificidade dos compostos de teste na HDACs endógenas. Há um foco em HDAC1 e HDAC6, que são avaliados simultaneamente. Para alcançar estas medições enzimáticas em um ensaio de incubação único, uma mistura de substratos HDAC não-específica e específicas é adicionada ao tratados e não tratadas células HeLa banhadas em uma placa de 96 poços. Após uma etapa de incubação, as células lysed para liberar os substratos e seus produtos de reação respectivos, que são separados e detectados usando um método de UHPLC-MS (Figura 1). Os produtos deacetylated de substratos MAL, MOCPAC e BATCP são o MAL deacetylated (dMAL), deacetylated MOCPAC (dMOCPAC) e deacetylated BATCP (dBATCP), respectivamente. Curvas dose-resposta podem ser construídas com compostos ativos.
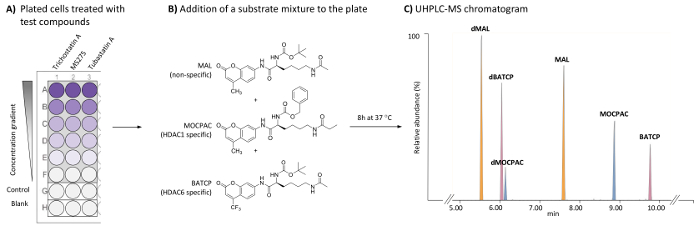
Figura 1: esquema geral para o ensaio HDAC baseada em célula identificar inibidores específicos de HDAC1 e HDAC6 pela análise de vários substratos UHPLC-MS. (A) esquema de uma típica placa de 96 poços contendo tratados (teste compostos) e as células HeLa não tratadas (controle), bem como espaços em branco sem célula. (B) estrutura química de substratos adicionado como uma mistura (21 µM cada) para ser deacetiladas por HDACs endógenas. (C) cromatograma típico UHPLC-MS, mostrando os picos de substratos adicionados (MAL, MOCPAC e BATCP) e seus produtos deacetylated (dMAL, dMOCPAC e dBACTP, respectivamente). Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
Inibição de HDAC é um tema quente na descoberta de medicamentos, com foco atual em inibidores de HDAC6-seletivo para terapia de câncer21. Seletividade HDAC geralmente é avaliada por uma série de ensaios de alta produtividade, sem célula, que pretende determinar a potência inibitória no sentido individual HDAC isoformas21. No entanto, a seletividade de um inibidor deve ser adicionalmente confirmada em células vivas, avaliando o status de acetilação de substratos …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Os autores reconhecem custo ação CM1406 (epigenéticas biologia química). Pesquisa reportada nesta publicação foi apoiada pela Fundação Pierre Mercier.
Materials
| BATCP | Sigma-Aldrich | B4061 | |
| MAL | Sigma-Aldrich | SCP0168 | Synonym: BOC-Ac-Lys-AMC |
| MOCPAC | Sigma-Aldrich | M2195 | |
| MS275 | Sigma-Aldrich | EPS002 | |
| Trichostatin A | Sigma-Aldrich | T8552 | |
| Tubastatin A | Sigma-Aldrich | SML0044 | |
| Acetonitrile, HPLC grade | Fisher Scientific | 10660131 | |
| Formic acid, LC/MS grade | Fisher Scientific | 10596814 | |
| H2O, HPLC grade | distilled H2O filtered through a Milli-Q purification system | ||
| HeLa cells | ATCC | ATCC CRM-CCL-2 | |
| Cell dissociation reagent TrypLE Express | ThermoFisher Scientific | 12604013 | |
| DMSO, cell culture grade | Applichem | 146463 | |
| DPBS | ThermoFisher Scientific | 14190144 | |
| Fetal bovine serum | Biowest | S1810 | |
| MEM | ThermoFisher Scientific | 22561021 | |
| Penicillin-Streptomycin | Bioconcept | 4-01F00-H | |
| 10X RIPA buffer | Abcam | ab156034 | |
| SigmaFast protease inhibitor tablets | Sigma-Aldrich | S8820 | |
| 96-well plates, sterile, flat-bottom, tissue culture treated Corning | VWR | 29442-058 | |
| 96-well plates, non-sterile, V-bottom Corning | VWR | 29442-404 | used in the centrifugation step |
| 96-well plate, conical bottom, Nunc | ThermoFisher Scientific | 249944 | compatible with the Acquity UHPLC system |
| T75 cell culture flasks Corning | Sigma-Aldrich | CLS430641 | |
| Peelable heat sealing foil | Waters | 186002789 | |
| Acquity UPLC system | Waters | ||
| Eppendorf Centrifuge 5810 R | Fisher Scientific | 05-413-323 | |
| Integration software: MassLynx V4.1 | Waters | Catalog number not available | |
| Combi thermo-sealer SP-0669/240 | Waters | Catalog number not available | |
| Quattro micro API Tandem Quadrupole System | Waters | Catalog number not available |
References
- Arrowsmith, C. H., Bountra, C., Fish, P. V., Lee, K., Schapira, M. Epigenetic protein families: a new frontier for drug discovery. Nat Rev Drug Discov. 11 (5), 384-400 (2012).
- Henikoff, S., Greally, J. M. Epigenetics, cellular memory and gene regulation. Curr Biol. 26 (14), R644-R648 (2016).
- Kim, C., et al. HDAC6 inhibitor blocks amyloid beta-induced impairment of mitochondrial transport in hippocampal neurons. PLoS One. 7 (8), (2012).
- Kawaguchi, Y., et al. The deacetylase HDAC6 regulates aggresome formation and cell viability in response to misfolded protein stress. Cell. 115 (6), 727-738 (2003).
- Zhao, Y., et al. Acetylation of p53 at lysine 373/382 by the histone deacetylase inhibitor depsipeptide induces expression of p21(Waf1/Cip1). Mol Cell Biol. 26 (7), 2782-2790 (2006).
- Berger, S. L. Histone modifications in transcriptional regulation. Curr Opin Genet Dev. 12 (2), 142-148 (2002).
- Simões-Pires, C., et al. HDAC6 as a target for neurodegenerative diseases: what makes it different from the other HDACs?. Mol Neurodegener. 8 (7), 1-16 (2013).
- Mottamal, M., Zheng, S., Huang, T. L., Wang, G. Histone deacetylase inhibitors in clinical studies as templates for new anticancer agents. Molecules. 20 (3), 3898-3941 (2015).
- Manal, M., Chandrasekar, M. J., Gomathi Priya, J., Nanjan, M. J. Inhibitors of histone deacetylase as antitumor agents: A critical review. Bioorg Chem. 67, 18-42 (2016).
- Mack, G. S. To selectivity and beyond. Nat Biotech. 28 (12), 1259-1266 (2010).
- . Study of ACY-1215 alone and in combination with bortezomib and dexamethasone in multiple myeloma (ACY-1215) Available from: https://clinicaltrials.gov/ct2/show/NCT01323751 (1211)
- . Study of ACY-1215 in combination with lenalidomide, and dexamethasone in multiple myeloma Available from: https://clinicaltrials.gov/ct2/show/NCT01583283 (2012)
- . 13ACY-1215 (ricolinostat) in combination with pomalidomide and low-dose dex in relapsed-and-refractory multiple myeloma Available from: https://clinicaltrials.gov/ct2/show/NCT01997840 (2013)
- . Study of ACY-241 alone and in combination with pomalidomide and dexamethasone in multiple myeloma Available from: https://clinicaltrials.gov/ct2/show/NCT02400242 (2015)
- Yee, A. J., et al. Ricolinostat plus lenalidomide, and dexamethasone in relapsed or refractory multiple myeloma: a multicentre phase 1b trial. Lancet Oncol. 17 (11), 1569-1578 (2016).
- Jung, M. Homogenous non-isotopic assays for histone deacetylase activity. Expert Opin Ther Pat. 13 (6), 935 (2003).
- Zwick, V., Simões-Pires, C., Cuendet, M. Cell-based multi-substrate assay coupled to UHPLC-ESI-MS/MS for a quick identification of class-specific HDAC inhibitors. J Enzyme Inhibi Med Chem. 31 (1), 209-214 (2016).
- Khan, N., et al. Determination of the class and isoform selectivity of small-molecule histone deacetylase inhibitors. Biochem J. 409 (2), 581-589 (2008).
- Glaser, K. B., et al. Differential protein acetylation induced by novel histone deacetylase inhibitors. Biochem Biophys Res Commun. 325 (3), 683-690 (2004).
- Butler, K. V., et al. Rational design and simple chemistry yield a superior, neuroprotective HDAC6 inhibitor, tubastatin A. J Am Chem Soc. 132 (31), 10842-10846 (2010).
- Jung, M., Yong, K. -. J., Velena, A., Lee, S., Sippl, W., Jung, M. Cell-Based Assays for HDAC Inhibitor Hit Validation. Epigenetic Targets in Drug Discovery. , (2010).
- Milli, A., et al. Proteomic analysis of cellular response to novel proapoptotic agents related to atypical retinoids in human IGROV-1 ovarian carcinoma cells. J Proteome Res. 10 (3), 1191-1207 (2011).
- Zwick, V., et al. Synthesis of a selective HDAC6 inhibitor active in neuroblasts. Bioorg Med Chem Lett. 26 (20), 4955-4959 (2016).
- Ciossek, T., Julius, H., Wieland, H., Maier, T., Beckers, T. A homogeneous cellular histone deacetylase assay suitable for compound profiling and robotic screening. Anal Biochem. 372 (1), 72-81 (2008).
- Heltweg, B., Dequiedt, F., Marshall, B. L., Brauch, C., Yoshida, M., et al. Subtype selective substrates for histone deacetylases. J Med Chem. 47 (21), 5235-5243 (2004).
- Copeland, R. A. . Evaluation of enzyme inhibitors in drug discovery. , (2005).

