Bimolecular पूरित अपनत्व शुद्धि (BiCAP) द्वारा विदारक मल्टी प्रोटीन सिगनल कॉम्प्लेक्स
Summary
यह पांडुलिपि Bimolecular पूरकता संबध शुद्धिकरण (BiCAP) के लिए प्रोटोकॉल का वर्णन करती है । इस उपंयास विधि विशिष्ट अलगाव और किसी भी दो बातचीत प्रोटीन के बहाव proteomic लक्षण वर्णन की सुविधा है, जबकि संयुक्त राष्ट्र के परिसर में व्यक्तिगत प्रोटीन के रूप में के रूप में अच्छी तरह से प्रतिस्पर्धा बाध्यकारी भागीदारों के साथ गठित परिसरों को छोड़कर ।
Abstract
प्रोटीन परिसरों के विधानसभा कई सेल संकेत मार्ग के विनियमन अंतर्निहित एक केंद्रीय तंत्र है । जैव चिकित्सा अनुसंधान का एक प्रमुख ध्यान केंद्रित है कि कैसे इन गतिशील प्रोटीन परिसरों के लिए एक विशिष्ट जैविक प्रतिक्रिया प्रत्यक्ष करने के लिए कई स्रोतों से संकेत एकीकृत अधिनियम, और कैसे यह कई रोग सेटिंग्स में विनियमित हो जाता है । इस प्रमुख जैव रासायनिक तंत्र के महत्व के बावजूद प्रयोगात्मक तकनीकों की कमी है जो इन बहु-आणविक सिगनल कॉम्प्लेक्सों के विशिष्ट और संवेदनशील deconvolution को सुगम कर सकती है ।
यहां इस कमी के माध्यम से एक प्रोटीन पूरक परख के संयोजन के माध्यम से संबोधित किया है एक अनुरूप-विशिष्ट nanobody, जो हम Bimolecular पूरक संबंध शुद्धि (BiCAP) का कार्यकाल है । इस उपंयास तकनीक विशिष्ट अलगाव और बहाव प्रोटीन बातचीत के किसी भी जोड़ी के proteomic लक्षण वर्णन की सुविधा, संयुक्त राष्ट्र के बहिष्कार-परिसर में व्यक्तिगत प्रोटीन और प्रतिस्पर्धा बाध्यकारी भागीदारों के साथ गठित परिसरों ।
BiCAP तकनीक अनुप्रवाह प्रयोगात्मक परख की एक विस्तृत सरणी के लिए अनुकूल है, और इस तकनीक से afforded विशिष्टता के उच्च डिग्री प्रोटीन जटिल विधानसभा के यांत्रिकी में और अधिक सूक्ष्म जांच की अनुमति देता है से वर्तमान में प्रयोग संभव है मानक संबध शुद्धि तकनीक ।
Introduction
प्रोटीन जटिल विधानसभा कई संकेत मार्ग1,2की spatiotemporal विशिष्टता को बनाए रखने में एक महत्वपूर्ण प्रक्रिया है । जबकि इस विनियामक भूमिका की महत्वपूर्ण प्रकृति व्यापक रूप से मान्यता प्राप्त है, इन परिसरों को परिशीलन करने के लिए उपलब्ध प्रायोगिक तकनीकों का अभाव है । अधिकांश interactomics अध्ययन व्यक्तिगत प्रोटीन, या व्यक्तिगत जटिल घटकों के अनुक्रमिक संवर्धन के साथ बातचीत पर ध्यान केंद्रित । यहां हम एक विशिष्ट प्रोटीन डिमर के अलगाव के लिए एक तकनीक पेश करते हुए घटक प्रोटीन के व्यक्तिगत moieties को छोड़कर के रूप में अच्छी तरह से प्रतिस्पर्धा बाध्यकारी भागीदारों के साथ गठित परिसरों3। हम इस तकनीक Bimolecular पूरक समानता शुद्धि (BiCAP) कहा जाता है, के रूप में यह एक पहले से विद्यमान प्रोटीन टुकड़ा पूरक परख, Bimolecular प्रतिदीप्ति पूरकता (BiFC) का एक संयोजन है, एक के उपंयास के प्रयोग के साथ रचना-विशिष्ट रिकॉमबिनेंट nanobody की ओर GFP और उसके डेरिवेटिव ( सामग्री की तालिका देखें).
एक ठेठ प्रोटीन टुकड़ा पूरक परख की अभिव्यक्ति पर निर्भर करता है “चारा” और “शिकार” प्रोटीन ऐसे luciferase4, β-galactosidase5, या ग्रीन फ्लोरोसेंट प्रोटीन (GFP)6 के रूप में रिपोर्टर के टुकड़े विभाजित करने के लिए जुड़े ( चित्र 1a) । चारा और शिकार प्रोटीन की बातचीत के माध्यम से, विभाजित रिपोर्टर डोमेन के लिए एक कार्यात्मक संरचना में गुना प्रोत्साहित किया जाता है, चारा और शिकार प्रोटीन की बातचीत की अनुमति के लिए visualized या quantified । BiCAP इस तकनीक का एक संस्करण है कि GFP संस्करण वीनस के टुकड़ों का उपयोग किया से अनुकूलित किया गया था । फ्लोरोसेंट प्रोटीन पूरक परख एक लाइव सेल में प्रोटीन-प्रोटीन बातचीत visualizing के लिए एक लोकप्रिय तरीका है, लेकिन अब तक इस एक समारोह7तक ही सीमित किया गया है । BiCAP इस संबंध में एक महत्वपूर्ण अग्रिम का प्रतिनिधित्व करता है, इस तकनीक के रूप में न केवल दृश्य के लिए अनुमति देता है, लेकिन यह भी अलगाव और परिणामस्वरूप प्रोटीन-प्रोटीन संपर्क के पूछताछ ।
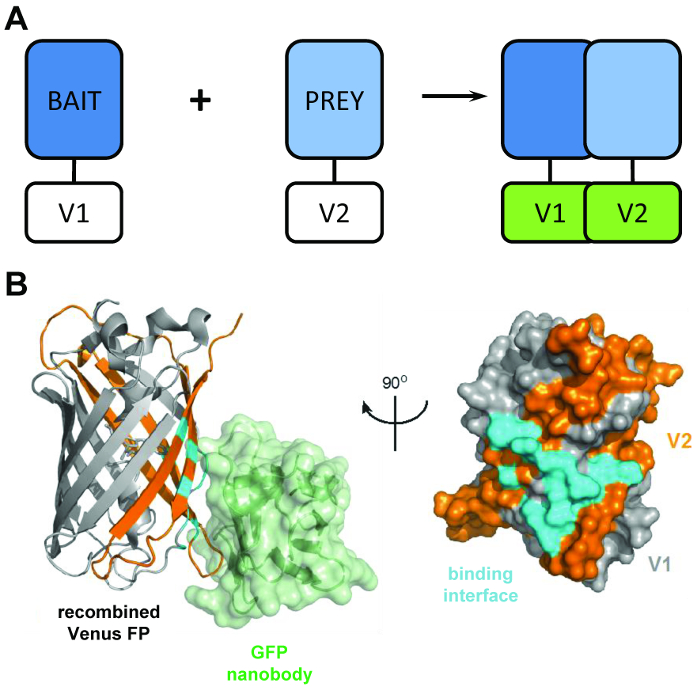
चित्रा 1: BiCAP तकनीक के पीछे संरचनात्मक प्रमुख । (क) एक योजनाबद्ध bimolecular प्रतिदीप्ति पूरक दिखा ‘ चारा ‘ और ‘ शिकार ‘ प्रोटीन के साथ टैग की गई मुख्य रूपरेखा के पीछे N-टर्मिनल V1 या C-पूर्ण लंबाई वीनस प्रोटीन के टर्मिनल V2 टुकड़े के साथ चिह्नित । (ख) GFP nanobody (हरा) और संयुक्त वीनस के बीच संपर्क अंतरफलक (सियान) का संरचनात्मक विश्लेषण, V1 (ग्रे) और V2 (नारंगी) टुकड़े की स्थिति (PDB प्रवेश 3OGO) दिखा । यह आंकड़ा republished fromCroucher एट अल.3 आळे से अनुमति के साथ पुनर्मुद्रित है । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।
BiCAP तकनीक वीनस के दो गैर फ्लोरोसेंट टुकड़े का उपयोग करता है (V1 और V2 नाम) जो अपनत्व का एक कम डिग्री के साथ सहयोगी जब तक एक बातचीत उनके संलयन भागीदारों के बीच होता है । इस उदाहरण में, दो विभाजन डोमेन fluorophore (चित्र 1b)6के कार्यात्मक β-बैरल संरचना में reफ़ोल्ड । BiCAP के प्रमुख नवाचार रिकॉमबिनेंट GFP nanobody की शुरूआत से आता है, जो epitope के β-बैरल पर तीन आयामी GFP (और वीनस जैसे वेरिएंट) जो सही ढंग से संयुक्त और तह fluorophore पर ही मौजूद है पहचानता है ( चित्र 1b)8. महत्वपूर्ण, GFP nanobody व्यक्तिगत वीनस टुकड़ों में से किसी को बांध नहीं है । यह प्रोटीन dimers के अलगाव की सुविधा के बाद ही दो प्रोटीन अपने इच्छा के एक परिसर का गठन किया है, विधियों है कि रासायनिक प्रेरित, मजबूर बातचीत9का उपयोग करने से प्राप्त की तुलना में अधिक प्रतिनिधि परिणाम के लिए अग्रणी ।
BiCAP एक शक्तिशाली तकनीक है कि विशेष रूप से बहु-प्रोटीन परिसरों पर केंद्रित है, जो संभवतः बहाव अनुप्रयोगों के एक नंबर के साथ संयुक्त किया जा सकता है भूमिका की हमारी समझ की दानेदार में सुधार करने के लिए इन परिसरों संकेत में खेलने transduction . यह भी सीटू मेंप्रोटीन बातचीत के दृश्य की अनुमति देने की महत्वपूर्ण विशेषता शामिल है । तिथि करने के लिए, BiCAP रिसेप्टर tyrosine कळेनासे (RTK) dimers3के interactome का विश्लेषण करने का एक प्रभावी तरीका के रूप में प्रदर्शित किया गया है, लेकिन इस विधि के अनुकूलन क्षमता का मतलब है कि यह लगभग किसी भी प्रोटीन संपर्क संदर्भ में अपनाया जा सकता है ।
Protocol
Representative Results
Discussion
BiCAP विशिष्ट प्रोटीन dimers अलग करने के लिए एक शक्तिशाली तरीका है, जबकि व्यक्तिगत घटकों को छोड़कर और उनके प्रतिस्पर्धा बाध्यकारी भागीदारों3। BiCAP एक प्रतिदीप्ति प्रोटीन पूरक परख के अनुकूलन पर आधारित…
Disclosures
The authors have nothing to disclose.
Acknowledgements
डी. आर. सी. कैंसर संस्थान एनएसडब्ल्यू फेलो और डी. एन. एस पहले कैंसर संस्थान एनएसडब्ल्यू फेलो थे । इस पांडुलिपि में प्रस्तुत अनुसंधान निष्कर्षों कैंसर संस्थान एनएसडब्ल्यू द्वारा वित्त पोषित किया गया (13/FRL/1-02 और 09/CDF/2-39), NHMRC (परियोजना अनुदान GNT1052963), विज्ञान फाउंडेशन आयरलैंड (11/SIRG/B2157), एनएसडब्ल्यू कार्यालय विज्ञान और चिकित्सा अनुसंधान, अतिथि परिवार फेलोशिप आणि Mostyn कुटुंब फाउंडेशन. J.F.H. और R.S. एक ऑस्ट्रेलियाई स्नातकोत्तर पुरस्कार के प्राप्तकर्ता थे ।
Materials
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid – Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) – 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
References
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).

